Построение слабых и сильных паттернов для заданного мотива
- Проект JalView с общим видом выравнивания и выбранным мотивом: motifs.jar
Для этой работы использовалось выравнивание aln17_1.fa из задания 9. В нем был выбран мотив - консервативный участок последовательностей. Мотив был выбран сканированием последовательностей на мотивы из базы ProSite. ProSite нашел мотив SH2 (Src homology 2) во всех последовательностях. Этот мотив состоит примерно из 100 аминокислотных остатков, его функция - связываться с фосфотирозин-содержащими белками, часто участвующими в сигнальных каскадах. Однако для дальнейшей работы я взяла часть этого мотива с 1 по 41 аминокислоты, поскольку дальше шел малоконсервативный участок с гэпами.
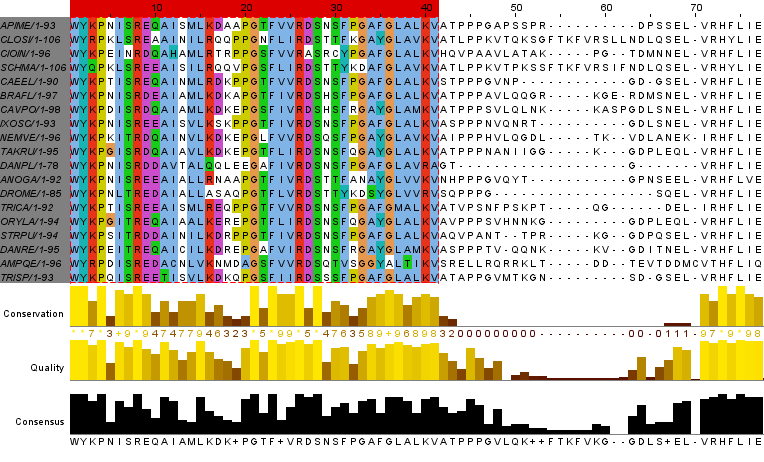
Рис.1. Мотив в выравнивании
Сильный паттерн
Для этого мотива был создан сильный паттерн. Он строился по принципу: если в колонке встречаются более 5 разных аминокислот, и они не состоят в одной группе (например, гидрофобных алифатических), то в паттерне ставился x, иначе все аминокислоты перечислялись. Цель сильного паттерна - найти исключительно гомологичные последовательности, хотя это может обернуться тем, что некоторые гомологи не подойдут под паттерн и не будут найдены.
Паттерн получился такой:
W-Y-[KRQ]-P-x-[IL]-[STN]-R-[DE]-[EQDA]-[AT]-[IVHC]-x-[LAIMV]-[LV]-[KRQA]-x(3)-[PEA]-G-x-F-[IVL]-[IV]-R-[DA]-S-x-[STC]-[FYV]-x-[GDN]-[ASG]-[FY]-[GA]-[LM]-[AVT]-[LIVM]-[KR]-[VA]То, что паттерн находит все исходные последовательности, было проверено на сайте ProSite (опция 3 - Submit PROTEIN sequences and MOTIFS to scan them against each other). Затем паттерн был проверен на базе UniProt:
- SwissProt: нашлось всего 12 белков, все гомологи белка тензина. Этот белок находится в местах фокальных контактов клеток. Он связывает актиновые филаменты, и содержит сайт связывания с фосфотирозином, который взаимодействует с бета-интегрином. Таким образом он связывает актиновые филаменты с интегриновыми рецепторами. [x][x]
- TrEMBL: 280 совпадений среди 279 последовательностей. В основном это белки с неизвестной функцией или гомологичные тензину, но были и белки, разрушающие клатриновые оболочки везикул, и белок, связывающий нуклеотиды, и белковая фосфатаза. Белок, в котором было найдено 2 сайта, был белком с неизвестной функцией из морского ежа.
В общем, сильный паттерн работает достаточно хорошо, потому что в проверенной базе SwissProt нашел только гомологичные тензину белки, а в TrEMBL подавляющее число известных белков были также гомологичными.
Слабый паттерн
В отличие от сильного, слабый паттерн делается для того, чтобы найти как можно больше гомологов, поэтому условия на колонки ставятся слабее. Обычно в таком случае, кроме гомологов, находится и большое число ложных находок.
Слабый паттерн:
W-Y-x-P-x(3)-R-x(12)-G-x-F-x(2)-R-x-S-x(6)-[FY]-x(4)-[KR]-xЗдесь в основном были перечислены аминокислоты, если в колонке была только одна, либо они все были одной группы. В остальных случаях ставился x. Паттерн находит все искомые последовательности.
- SwissProt: нашлось те же 12 белков, гомологов тензина.
- TrEMBL: нашлось 310 совпадений в 309 последовательностях. В подавляющем большинстве это также тензин или белки с неизвестной функцией.