В заданной последовательности нуклеотидов можно найти границы генов - они заключены между старт-кодоном и стоп-кодоном. Для найденных рамок считывания можно подтвердить определенными алгоритмами, что этот ген существует.
Задание 1
Был дан контиг микробиома кардинального отдела желудка валлаби. Нужно было его проаннотировать. Сначала я получила открытые рамки считывания программой getorf: getorf -table 11 -find 1 -minsize 60, где table задает генетический код (11 - бактериальный), find указывает, что нужно выдать транслированные последовательности от старт-кодона до стоп-кодона, а minsize задает минимальный размер рамки. Файл со списком открытых рамок считывания: [x]
Начало и конец даны в нуклеотидах, т.к. во-первых, getorf дает такой вывод, а во-вторых, он же может сдвигать рамку считывания относительно начала на 1-3 нуклеотида, тогда сложно отсчитывать аминокислоты.
| Начало | Конец | Длина в а.о. | Цепь | Описание | Характеристики бласта |
| 33 | 719 | 229 | + | Белок рестрикции I типа | Самый близкий гомолог - белок P10485.2 из E.coli с E-value 3e-12, покрытием 95% и идентичностью 47%. Другие хорошие гомологи - белки рестрикции I типа из других бактерий (микоплазма, метанокалдококк). |
| 736 | 1326 | 197 | + | Белок системы рестрикции I типа | Ближайший описанный гомолог - P71344.1 из Haemophilus influenzae. Другие гомологи - неизвестные белки из Methanocaldococcus jannaschii и белки рестрикции I типа из микоплазмы и E.coli |
| 1410 | 1970 | 187 | + | не найдено достоверных гомологов | - |
| 1754 | 1987 | 78 | + | не найдено достоверных гомологов | - |
| 1846 | 2058 | 71 | + | не найдено достоверных гомологов | - |
| 2123 | 2395 | 91 | + | не найдено достоверных гомологов | - |
| 2107 | 2778 | 224 | + | белок системы рестрикции I типа | Совпадение со 100% покрытием и 44% идентичностью с белком P17224.1 из E.coli. Также хорошие совпадения с белками рестрикции I типа из стафилококков |
| 2570 | 2370 | 67 | - | не найдено достоверных гомологов | - |
| 2766 | 2134 | 211 | - | не найдено достоверных гомологов | - |
| 2096 | 1866 | 77 | - | не найдено достоверных гомологов | - |
| 2086 | 1850 | 79 | - | не найдено достоверных гомологов | - |
| 1688 | 1386 | 101 | - | не найдено достоверных гомологов | построены недостоверные выравнивания с фосфорибозиламиноимидазол-сукцинокарбоксамид синтазами из разных бактерий с покрытием около 40% и минимальным E-value 0.25 |
| 1140 | 703 | 146 | - | не найдено достоверных гомологов | Сделал несколько выравниваний с маленьким покрытием (23%) и высоким E-value (4-5) для рецепторов ацетилхолина M1 из нескольких млекопитающих. Поскольку белок должен быть бактериальный, выравнивание явно недостоверно. |
| 818 | 546 | 91 | - | не найдено достоверных гомологов | - |
| 661 | 464 | 66 | - | не найдено достоверных гомологов | - |
| 442 | 215 | 76 | - | не найдено достоверных гомологов | - |
По результатам бласта для трех генов были найдены гомологи среди белков рестрикции I тела; возможно, данный контиг - это оперон генов белков этой системы. Все три гомолога были найдены на одной цепи и не имели перекрываний. Все остальные рамки считывания, вероятно, являются случайными последовательностями, для большинства из них были построены недостоверные выравнивания с совершенно разными белками из разных организмов. Известно, что вероятность получить открытую рамку считывания длины не меньше данной убывает экспоненциально. Большинство получившихся последовательностей без гомологов имеют длину менее 100 аминокислотных остатков, хотя некоторые сравнимы длиной с опознанными генами (> 146 оснований).
Предсказание генов эвристическим алгоритмом с помощью программы GeneMark
Гены можно предсказывать методом, описанным в первом задании, а можно с помощью "эвристического алгоритма", который, по-видимому, не использует выравнивания и поиск гомологов, а использует какие-то другие особенности последовательностей, позволяющие сказать, что это ген. Эвристический алгоритм работает гораздо быстрее (у меня сработал за 1 секунду).
Я использовала программу GeneMark для поиска генов в том же контиге. Алгоритм построил график, где показал вероятность наличия гена в данном участке последовательности.
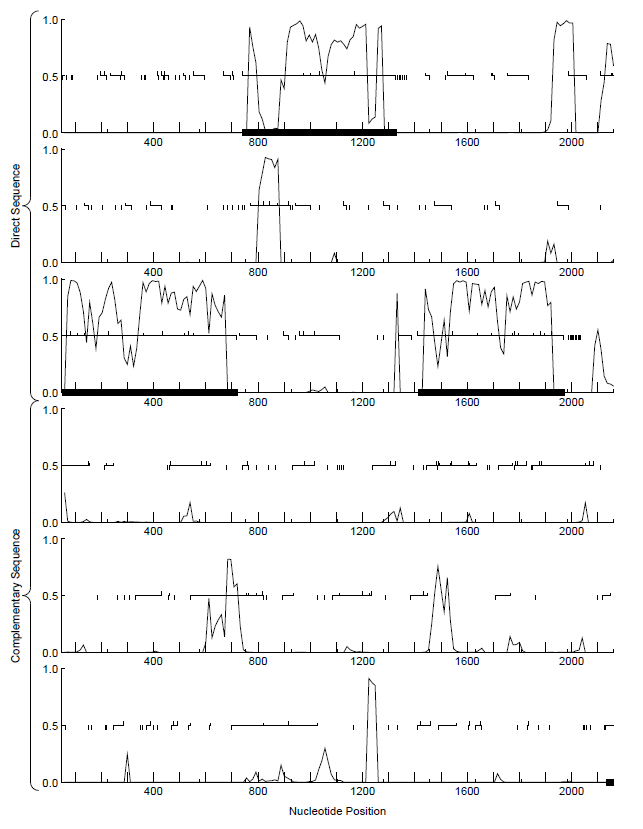
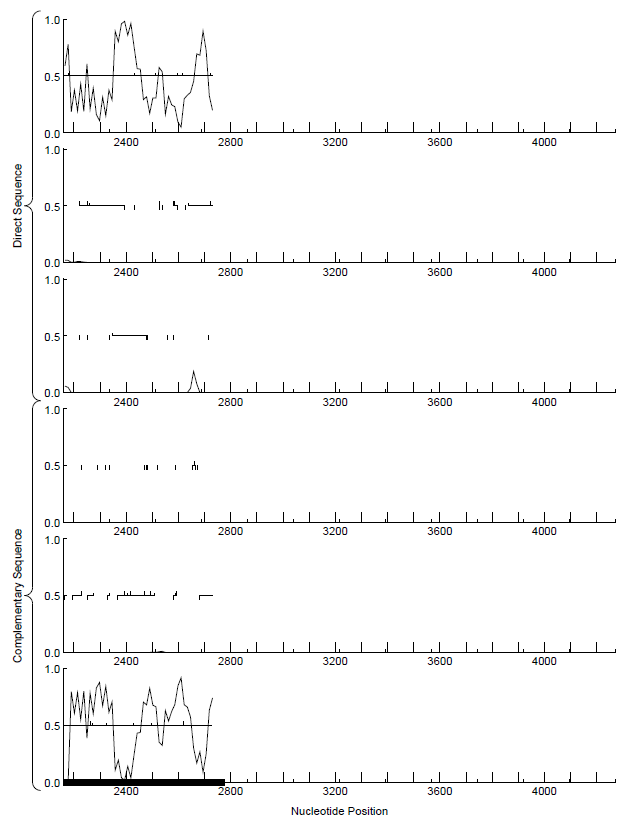
Рис.1. Гены контига, предсказанные программой GeneMark. По горизонтальной оси - номер нуклеотида последовательности, по вертикальной - вероятность существования гена в данной области. Шесть графиков для одной области соответствуют трем рамкам считывания для прямой и трем для обратной цепи. Предположительные гены подчеркнуты жирным.
| Начало | Конец | Длина в нукл. | Направление цепи |
| 51 | 722 | 672 | + |
| 742 | 1329 | 588 | + |
| 1413 | 1973 | 561 | + |
| >2778 | 2131 | 648 | - |
Сравнение результатов
На таблицу ниже приведено сравнение найденных генов методом поиска гомологов и методом GeneMark. Видно, что три гена приблизительно перекрываются, но их позиции начала и концов различаются. Почему различаются начала? Дело в том, что мы использовали таблицу генетического кода 11 - она работает для бактерий, архей и растительных пластид. В этой таблице старт - это не только ATG, но и некоторые другие кодоны. Например, ген 2 в варианте GeneMark начинается с ATG, а в варианте getorf+BLAST - с TTG. Во всех генах, найденных обоими методами, разница между позицией начала кратна трем, т.е. не сдвинута рамка считывания. Третий по счету ген был найден GeneMark, но не найден getorf+BLAST. Четвертый ген GeneMark посчитал расположенным на обратной цепи, хотя на графике выше видно, что он может быть и на прямой. Видимо, GeneMark не смог рассчитать направление, т.к. часть гена выходит за рамки контига.
| № гена | Начало гена | Конец гена | Цепь | ||
| getorf и BLAST | GeneMark | getorf и BLAST | GeneMark | ||
| 1 | 33 | 51 | 719 | 722 | + |
| 2 | 736 | 742 | 1326 | 1329 | + |
| 3 | - | 1413 | - | 1973 | + |
| 4 | 2107 | 2131 | 2778 | 2778 | + (-?) |