WebGeSTer
База WebGeSTer позволяет находить Rho-независимые терминаторы бактерий. По утверждениям авторов, она является наибольшим сборником терминаторов транскрипции с более 2 200 000 бактериальных терминаторов. Алгоритм WebGeSTer позволяет найти Rho-независимые терминаторы в любом полностью секвенированном геноме бактерии, выдавая также структурные параметры терминатора: длину стебля и петли (шпилька является обязательным элементом Rho-независимого терминатора), U-конец, а также несовпадения и гэпы.
На сайте WebGeSTer есть 2 главных инструмента - поиск по базе данных (WebGeSTer DB) и поиск по загруженному пользователем геному (WebGeSTer).
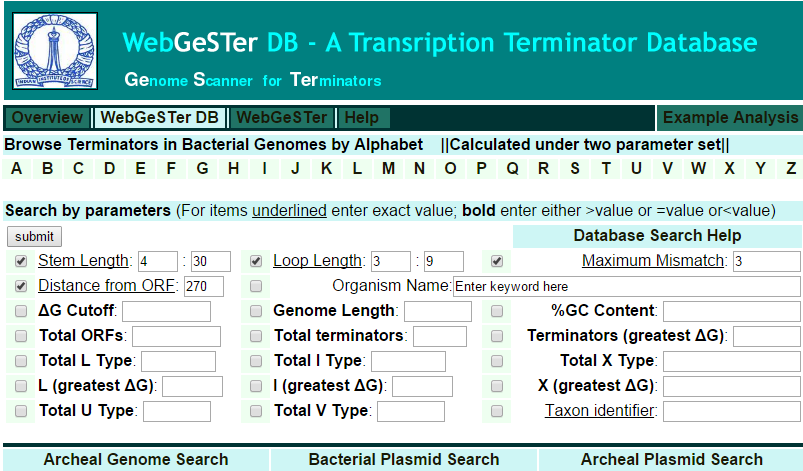
На вкладке WebGeSTer DB есть много параметров для поиска, такие как длина шпильки, дистанция от ORF, ограничение по ΔG, таксономия. Параметры I, L, U и V type отвечают за различные типы шпилек:
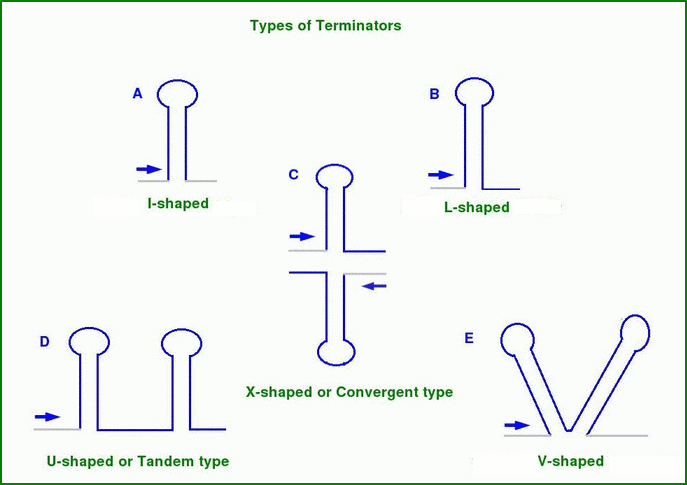
К сожалению, я не смогла заставить WebGeSTer DB принять параметры, отличные от стандартных, поскольку он просто не выдавал результат. Если запустить его со стандартными параметрами, он покажет список всех проанализированных геномов, которые у него есть. Я зашла на страницу Bacillus subtilis: там есть таблица с количеством генов на двух цепях, терминаторов различных типов и их лучший ΔG:
Также есть интерактивная карта генома:

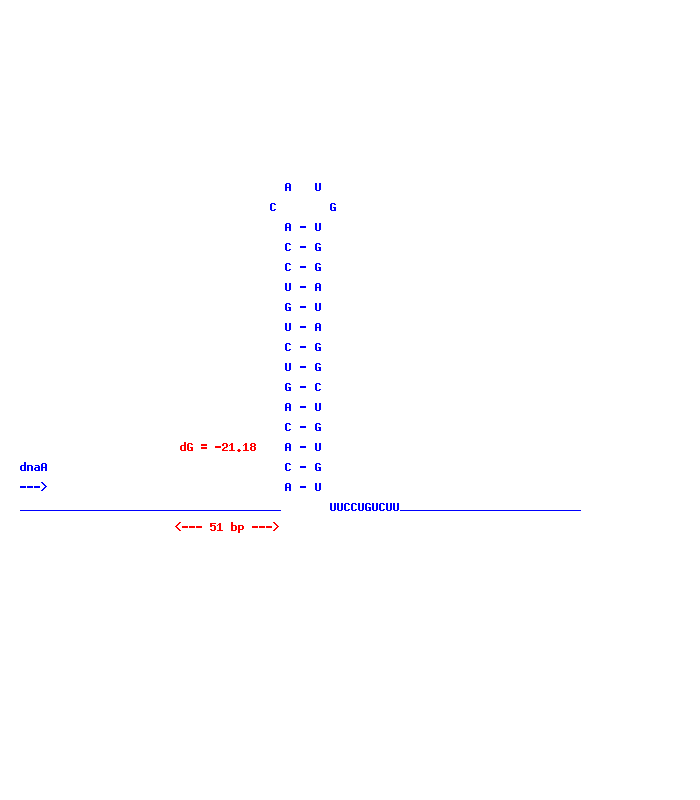
По ней можно перемещаться, задавая координаты или название гена. Красным выделены гены со ссылкой на их запись в ncbi, а шпилькой обозначены терминаторы. Они ведут на структуры для каждой шпильки:
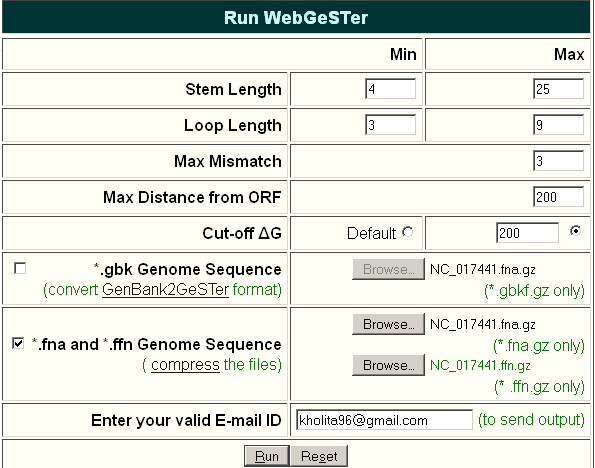
На вкладке Run WebGeSTer можно указать параметры для поиска и загрузить файл с геномом. Стандартные параметры: Stem Length 4:30, Loop Length 3:9, Max Mismatch 3, Max Distance from ORF 270. Я выбрала параметры, как выше на рисунке, и смотрела терминаторы в геноме Chlamydia trachomatis Sweden2. Вычада в форме такой же таблицы, как в WebGeSTer DB, но он находит терминаторы только на одной цепи.
Опция 'Graph' позволяет построить график, показывающий распределение терминаторов к низу от генов. Предлагаются опции:- palins: Palindromic structures on both strands.
- palinsreg: Palindromic structures on the regular strand.
- palinscmp: Palindromic structures on the complementary strand.
- tandem: Multiple structures found downstream of the same gene on both strands.
- tandemreg: Multiple structures found downstream of the same gene on the regular strand.
- tandemcmp: Multiple structures found downstream of the same gene on the complementary strand.
- vpalins: V-shaped structures found on both strands.
- vpalinsreg: V-shaped structures found on the regular strand.
- vpalinscmp: V-shaped structures found on the complementary strand.
- greatest_dg: are the Best structures (most negative ?G) found downstream of the genes on both strands.
- greatest_dg_reg: are the Best structures (most negative ?G) found downstream of the genes on the regular strand.
- greatest_dg_cmp: are the Best structures (most negative ?G) found downstream of the genes on the complementary strand.
- converge_palins: X-shaped structures that can function as terminator for two convergent genes on different strands taking palins file as input.
- converge_greatest_dg: Best X-shaped structures that can function as terminator for two convergent genes on different strands taking greatest_dg file as input.
'View' позволяет выбрать L, I, или все терминаторы. 'Point of Termination' отмечает терминацию на начале, конце или середине шпильки. Другие параметры позволяют выбрать размер окна, расстояние от ORF и ΔG. Я выбрала greatest_dg, View All, и терминацию на начале.
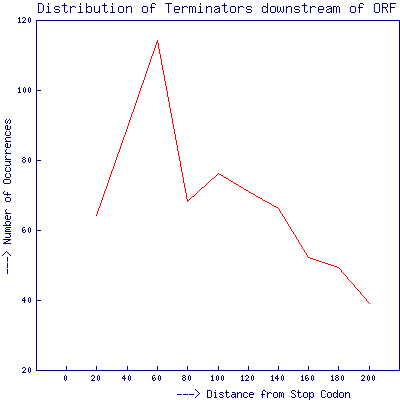
Получившийся график отражает расстояние терминаторов с лучшим ΔG от стоп-кодона. Пик находится на 60, дальше количество терминатором падает с расстоянием.