Проект JalView: TRANSMEMBRANE.jvp
Мне был выдан трансмембранный белок с идентификатором в базу PDB 4cz9. Это Na+/H+ антипортер из археи Pyrococcus abyssi. С помощью BLAST была составлена выборка из 24 последовательностей гомологов 4cz9 - по 8 из бактерий, архей и эукариот. Вот файл: [x]
Эти последовательности (вместе с оригинальной) были выровнены MUSCLE и полученное выравнивание было открыто в Jmol. К оригинальной последовательности
была прикреплена структура (опция Structure->Associate sequence with structure). Структура белка представлена на рисунке 1:
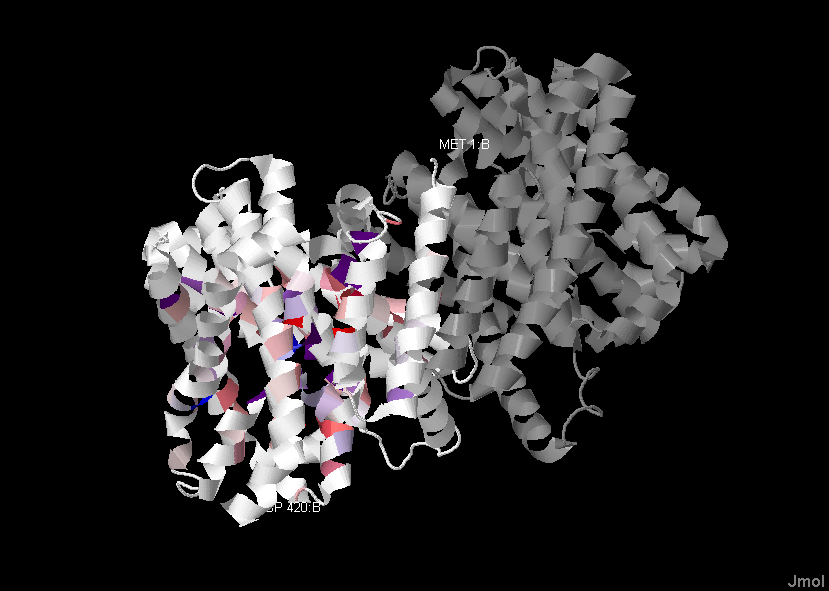
рис 1. Структура Na+/H+ антитранспортера из Pyrococcus abyssi. Раскраска по гидрофобности с консервативностью >30%. Белок ориентирован так, что сверху находится внеклеточная часть, а внизу - внутриклеточная.
К выравниванию были добавлены 2 строки с аннотациями. В первой (TM_Real) буквами M помечены области, предположительно являющиеся трансмембранными участками исходя из структуры. Такими областями я считала примерно параллельные альфа-спирали. Таких в белке оказалось 14.
Один из гомологов 4cz9, WP_013466313 из Thermococcus barophilus, был подан программе TMHMM, которая предсказывает трансмембранные участки. Программа нашла 13 участков.
# gi|503231652|ref|WP_013466313.1| Length: 446 # gi|503231652|ref|WP_013466313.1| Number of predicted TMHs: 13 # gi|503231652|ref|WP_013466313.1| Exp number of AAs in TMHs: 262.50225 # gi|503231652|ref|WP_013466313.1| Exp number, first 60 AAs: 41.28801 # gi|503231652|ref|WP_013466313.1| Total prob of N-in: 0.05081 # gi|503231652|ref|WP_013466313.1| POSSIBLE N-term signal sequence gi|503231652|ref|WP_013466313.1| TMHMM2.0 outside 1 12 gi|503231652|ref|WP_013466313.1| TMHMM2.0 TMhelix 13 30 gi|503231652|ref|WP_013466313.1| TMHMM2.0 inside 31 34 gi|503231652|ref|WP_013466313.1| TMHMM2.0 TMhelix 35 57 gi|503231652|ref|WP_013466313.1| TMHMM2.0 outside 58 66 gi|503231652|ref|WP_013466313.1| TMHMM2.0 TMhelix 67 89 gi|503231652|ref|WP_013466313.1| TMHMM2.0 inside 90 95 gi|503231652|ref|WP_013466313.1| TMHMM2.0 TMhelix 96 118 gi|503231652|ref|WP_013466313.1| TMHMM2.0 outside 119 122 gi|503231652|ref|WP_013466313.1| TMHMM2.0 TMhelix 123 145 gi|503231652|ref|WP_013466313.1| TMHMM2.0 inside 146 157 gi|503231652|ref|WP_013466313.1| TMHMM2.0 TMhelix 158 180 gi|503231652|ref|WP_013466313.1| TMHMM2.0 outside 181 206 gi|503231652|ref|WP_013466313.1| TMHMM2.0 TMhelix 207 229 gi|503231652|ref|WP_013466313.1| TMHMM2.0 inside 230 235 gi|503231652|ref|WP_013466313.1| TMHMM2.0 TMhelix 236 258 gi|503231652|ref|WP_013466313.1| TMHMM2.0 outside 259 262 gi|503231652|ref|WP_013466313.1| TMHMM2.0 TMhelix 263 280 gi|503231652|ref|WP_013466313.1| TMHMM2.0 inside 281 299 gi|503231652|ref|WP_013466313.1| TMHMM2.0 TMhelix 300 322 gi|503231652|ref|WP_013466313.1| TMHMM2.0 outside 323 331 gi|503231652|ref|WP_013466313.1| TMHMM2.0 TMhelix 332 354 gi|503231652|ref|WP_013466313.1| TMHMM2.0 inside 355 360 gi|503231652|ref|WP_013466313.1| TMHMM2.0 TMhelix 361 383 gi|503231652|ref|WP_013466313.1| TMHMM2.0 outside 384 397 gi|503231652|ref|WP_013466313.1| TMHMM2.0 TMhelix 398 420 gi|503231652|ref|WP_013466313.1| TMHMM2.0 inside 421 446
Найденные трансмембранные спирали были размечены во второй строке аннотаций (TM_Predicted). Найденные области неплохо совпали с выявленнвми по структуре. Выравнивание приведено на рис. 2, а также находится в приведенном проекте Jmol. В области 460-520 у предсказания TMHMM одна спираль, а не две, но в структуре 4cz9 на этом месте действительно идут 2 спирали в одном направлении, разделенные небольшим участком из двух аминокислот. В гомологе этот участок вполне может быть одной альфа-спиралью.

Видно, что в трансмембранных областях довольно много консервативных позиций, тогда как области между ними вариабельны. Часто встречаются гидрофобные аминокислоты: лейцин, изолейцин, валин, фенилаланин, глицин. Это логично, т.к. трансмембранные участки находятся в гидрофобной липидной мембране. Встречаются и заряженные аминокислоты, например аргинин на 440 позиции. Это возможно, т.к. трансмембранные участки образуют структуру (альфа-спирали), и заряд может "погашаться" на взаимодействия с другими аминокислотами в спирали.