В процессе выполнения прошлого практикума было получено дерево из выравнивания с помощью программы JalView. Построение производилось методом Neighbor-Joining using % identity. Это дерево было укоренено с помощью программы retree.exe. Для открытия файл нужно переименовать в intree. При запуске программа картинку дерева и опции:
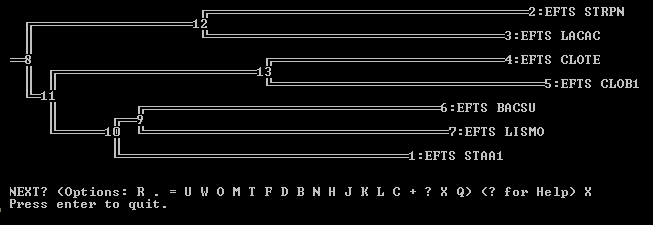
Опция "M" укореняет дерево в средней точке. Опция "W" записывает дерево в файл outtree.
Полученное дерево было открыто в программе MEGA опцией User Tree > Display Newick Trees. Полученное дерево:
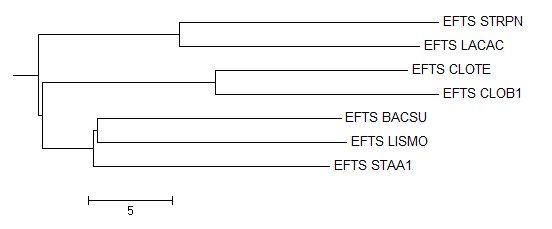
Видно, что построенное по выравниванию белков дерево отличается от сделанного в первом практикуме (которое соответствует реальной филогении бактерий). Ветвь {CLOTE, CLOB1}, внешняя в оригинальном дереве, здесь объединена в одну группу с {BACSU, LISMO, STAA1}, а {STRPN, LACAC} стала внешней.