В этом задании я взяла белок сукцинил-КоА-синтетазу 1EUC и нашла ее структурные гомологи с помощью сервиса PDBeFold. Среди результатов я выбрала четыре с rmsd между 0.8 и 2.5, и Nalign больше 50%: 1cqi:A, 1oi7:A, 3mwd:B и 2yv1:A. Эти четыре белка, вместе с 1eucб были поданы PDBeFold на структурное выравнивание. Из выдачи сервера я скачала множественное выравнивание (1euc_alignment_structural.fasta) и PDB-файлы с выравненными белками, которые затем открыла в PyMol.
Эти же пять белков были поданы MUSCLE на множественное выравнивание (1euc_align_muscle.fasta). Полученное выравнивание отличается от структурного: например, область 31-37 белка 3MWD PDBeFold считает вставкой, а MUSCLE - частью блока выравнивания (черный прямоугольник на рис.1).

Рис.1. Сравнение структурного выравнивания (сверху) и выравнивания MUSCLE. Получено с помощью JalView.
Если открыть структурное выравнивание, то видно, что у 3MWD в этом месте петля, не выровненная с какими-либо участками других белков (рис.2, розовый):
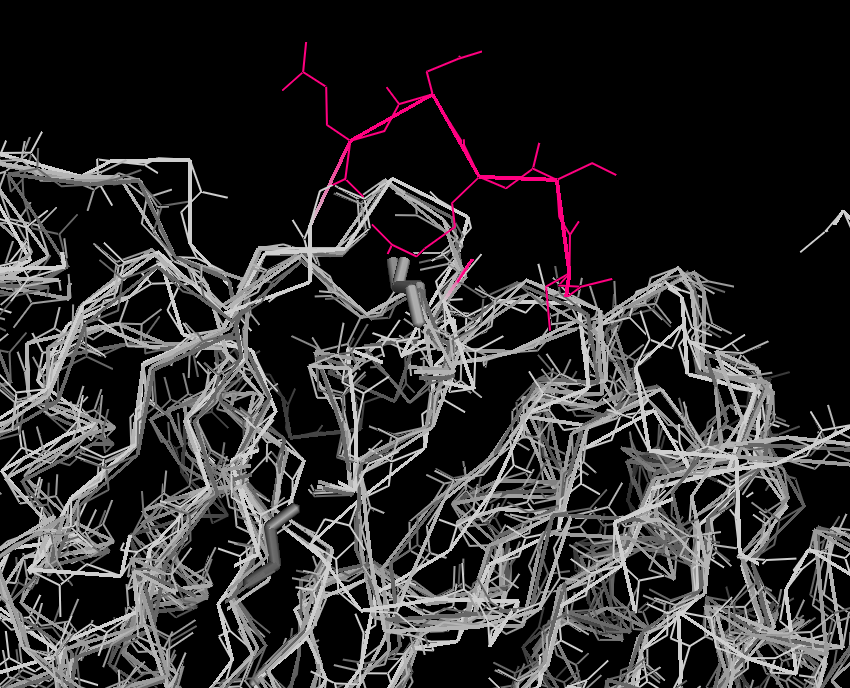
Рис.2. Участок структурного выравнивания гомологов 1EUC. Рисунок получен в PyMol.