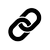1. Раскраска участка поверхности: pymol
Я рассматривала комплекс димерного пуринового репрессора PurR с ДНК, лежащего в базе PDB под идентификатором 1ZAY. Я воспроизвела в PyMol димер из асимметрической единицы командой:
symexp sym, 1zay, (1zay), 1
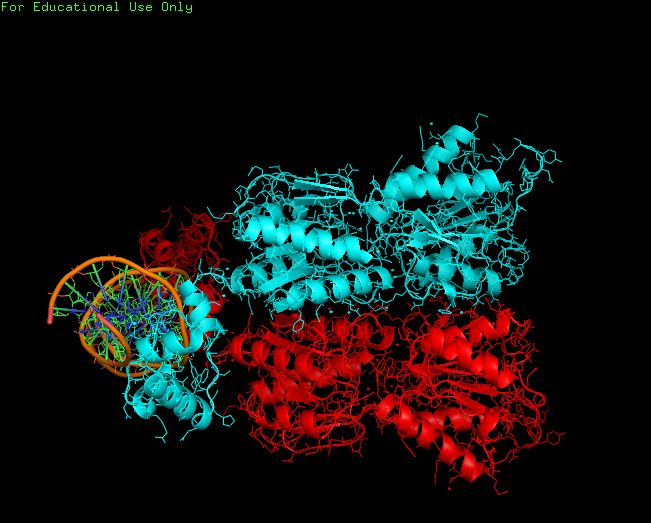
Рис.1. Димер 1ZAY в комплексе с ДНК
Далее были созданы поверхности контакта макромолекул. Контактами считалось расстояние между атомами ≤ 4Å. Это осуществлялось командой:
select S around N
где S - нужный объект, N - расстояние, на котором от S ищутся контакты.
а) контакт мономера белка с симметричным мономером на фоне остовной модели мономера:

Рис.2. Поверхность контакта между мономерами 1ZAY
б) поверхность контакта димера белков с двойной спиралью ДНК на фоне остовной модели части белка, вовлечённой в контакт:
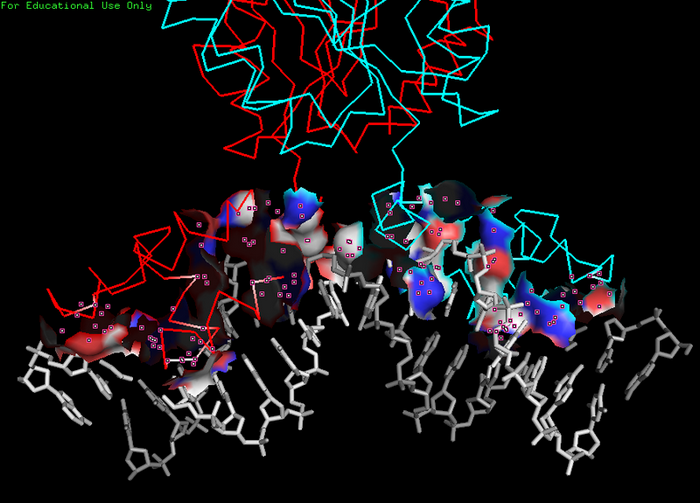
Рис.2. Повернхность контакта димера белков с ДНК
в) поверхность контакта ДНК с димером белков на фоне проволочной (sticks) модели двойной спирали:
Рис.3. Поверхность контакта ДНК с димером белков
2. Гидрофобные кластеры
С помощью программы CluD были найдены гидрофобные кластеры размером не менее 10 атомов. Программа нашла 2 кластера, один соответствующий регуляторному домену, а второй - ДНК-связывающему. С помощью скрипта *, переводящего скрипт RasMol в скрипт PyMol, в PyMol были выделены атомы, соответствующие найденным гидрофобным кластерам. На рис. 4 показана поверхность контакта между мономерами 1ZAY с выделенными гидрофобными участками:
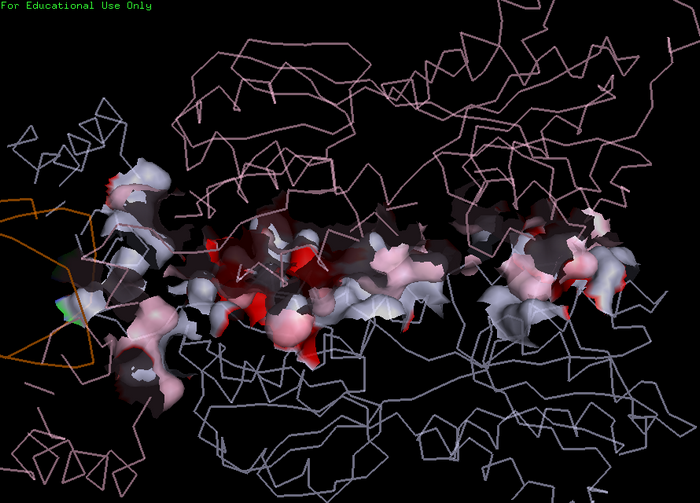
Рис.4. Поверхность контакта мономеров 1ZAY. Красным отмечены области, входящие в гидрофобные кластеры.
Отдельная благодарность Диме Пензару за предоставленный скрипт