Практикум 11
Практикум 11
Выбор и описание семейства доменов
С помощью таблицы доменов Pfam был выбран белковый домен PF18087. Его характеристики представлены ниже:
- AC: PF18087
- ID: RuBisCo_chap_C
- Seed: 85
- Full: 334
- Name: Rubisco Assembly chaperone C-terminal domain
- Domain Architectures: 84
- Swiss-Prot: 9
- 3D structures: 10
- Taxonomy: Чаще встречется у бактерий (615), также у эукариот (423).

- Function: С-концевой домен (β-домен) белка RuBsiCo Assembly Chaperone (Raf1). Этот домен образует промежуточные комплексы с RbcL, предшествующие синтезу RuBisCo (RuBisCo - гексадекадимер, состоит из 8 больших субъединиц RbcL, кодируемых хлоропласты геномом, и 8 малых судъединиц RbcS, кодируемых ядром). Также β-домен имеет функцию димеризации для образования полимеров вида Raf12-RbcL2 и Raf18-RbcL8. β-домены связываются вокруг экватора с каждым димером RbcL, чтобы вместе с α-доменом Raf1 образовать октамеры, необходимые для синтеза RuBisCo.
Описание выравнивания seed с точки зрения гомологичности всех последовательностей или их подмножества
Блоков, которые содержат все последовательности в выравнивании не оказалось, однако есть консервативные во всех последовательностях одиночные аминокислоты: 66.
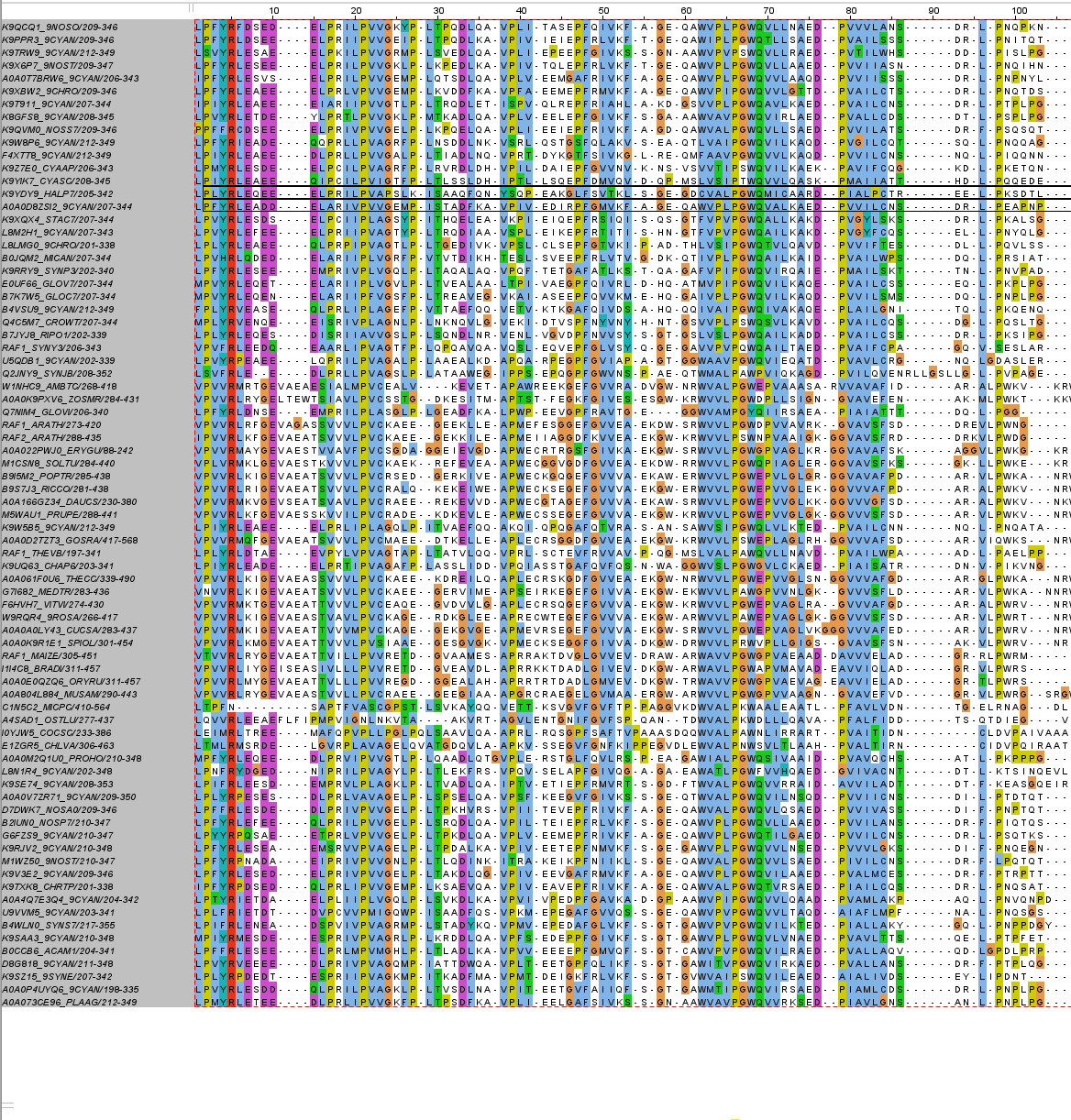
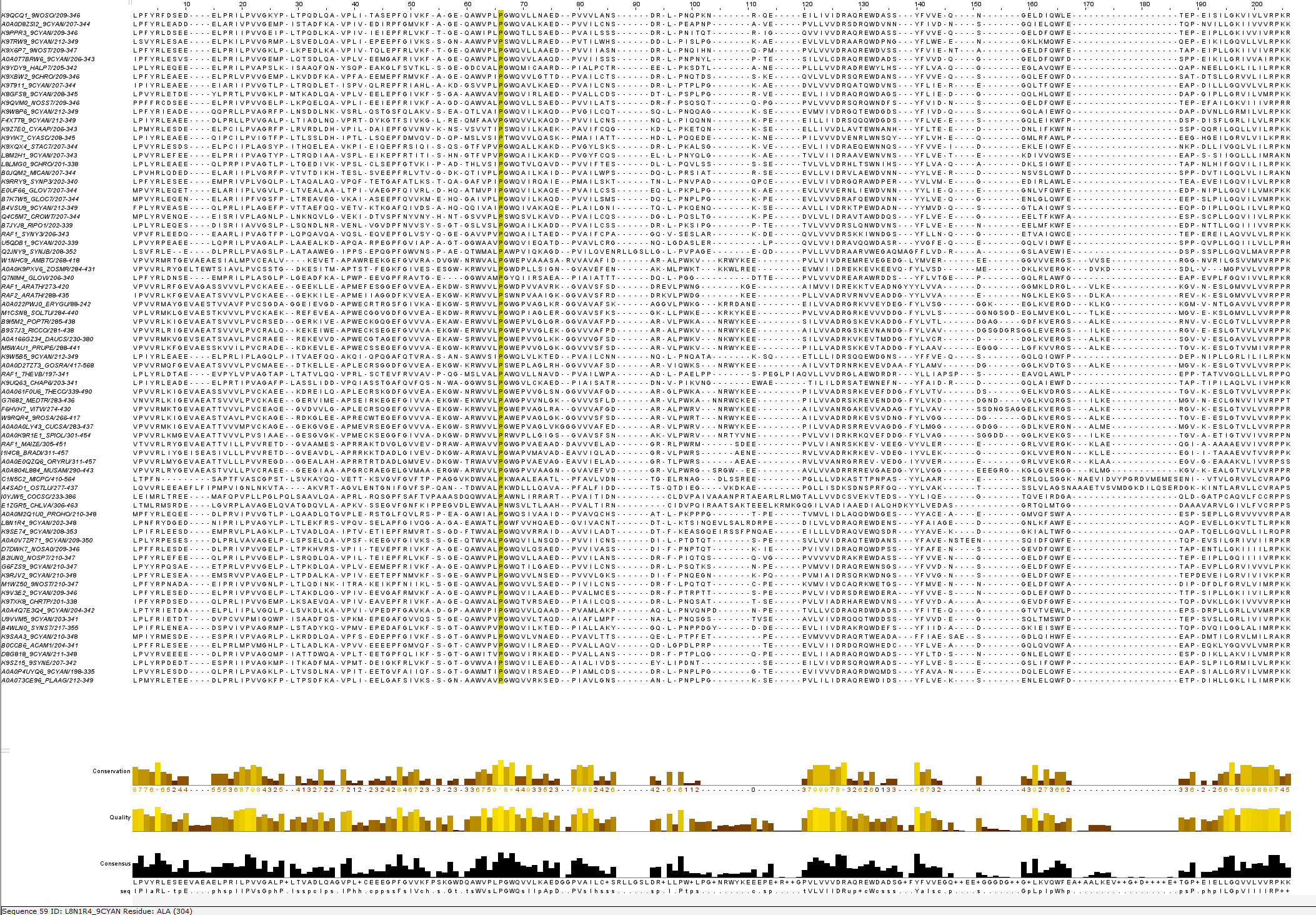
- Ссылка на проект в Jalview
Были выбрано наиболее схожие последовательности: таким же способом выровняны в новом окне. Максимально достоверные блоки - 17-18, 58-59, 104-105, 128-129. Единичные аминокислоты, консервативные во всех последовательностях - 5, 41, 56, 67, 78, 93, 99, 118, 137.
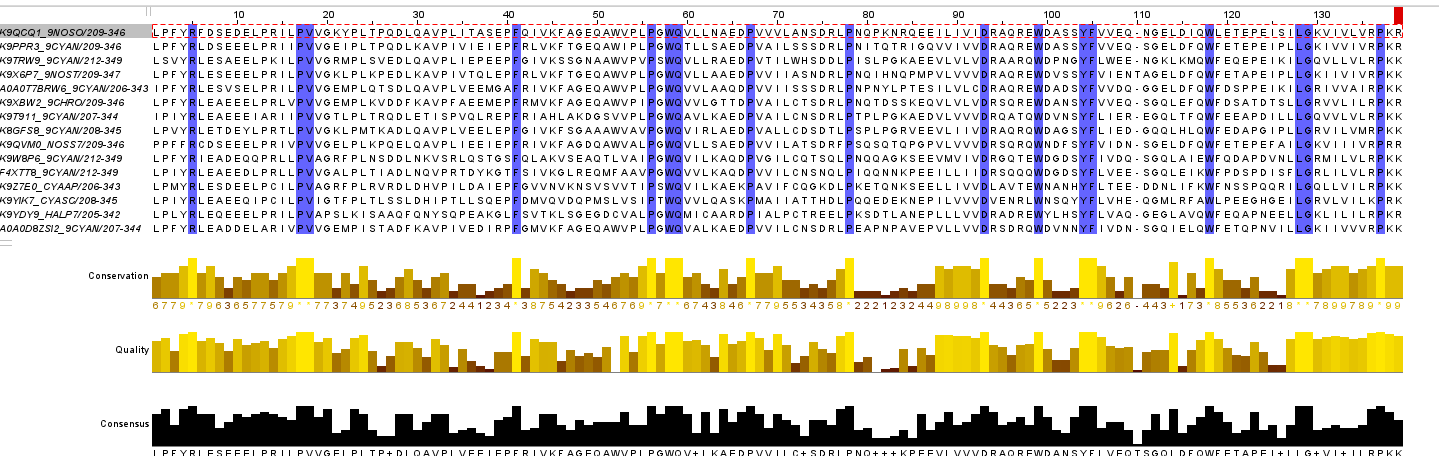
- Ссылка на проект в Jalview
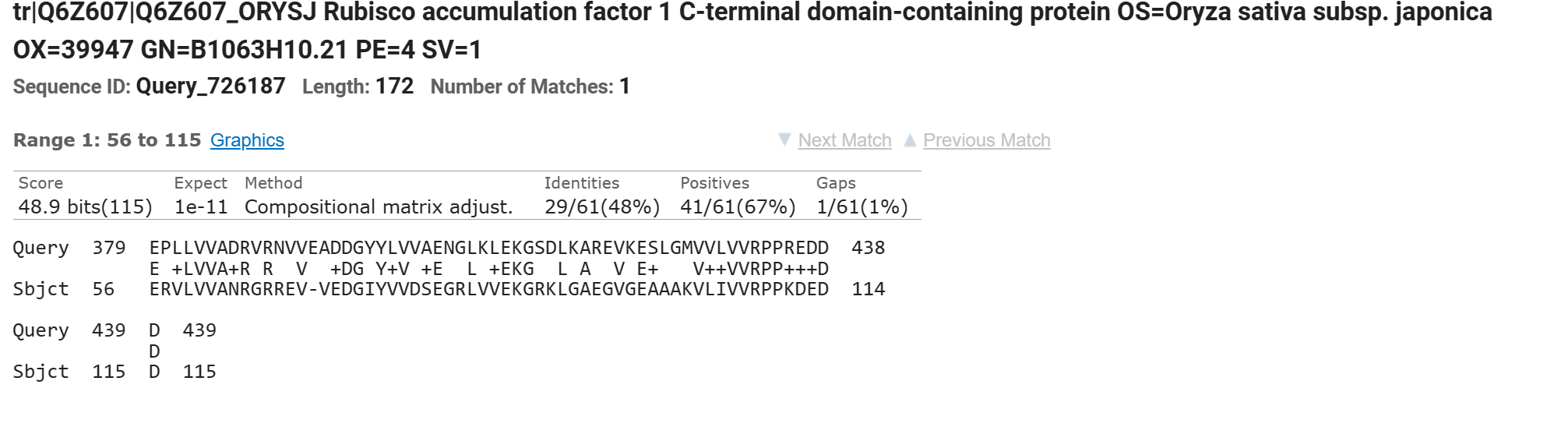
Карта локального сходства (dotplot) двух последовательностей с одним и тем же доменом, но с разной доменной архитектурой
Была выбрана доменная архитектура PF18579 - PF18578 - PF18087 (AC белка: Q6Z607, Organism: Oryza sativa subsp. japonica (Rice), последовательность 1) и доменная архитектура PF18087 (AC белка: Q9SR19, Organism: Arabidopsis thaliana (Mouse-ear cress), последовательность 2).
На карте локального сходства 1 разрыв, соответствующий инделям в выравнивании.