Практикум 7
Сравнение предсказаний трансмембранных участков в бета-листовом белке
Для выполнения данного задания был выбран белок BtuB. Ниже представлена информация о белке:
Таблица 1. Информация о белке BtuB.
Название белка (русский)
Транспортер кобаламина для наружной мембраны BtuB
Название белка (английский)
Outer membrane cobalamin transporter BtuB
Идентификатор PDB
1NQE
Идентификатор UniProt
BTUB_ECOLI
Организм
Escherichia coli (strain K12)
Мембрана
Бактериальная грамотрицательная наружная мембрана
Функция белка
Белок является транспортером витамина B12 через наружную мембрану в периплазматическое пространство.
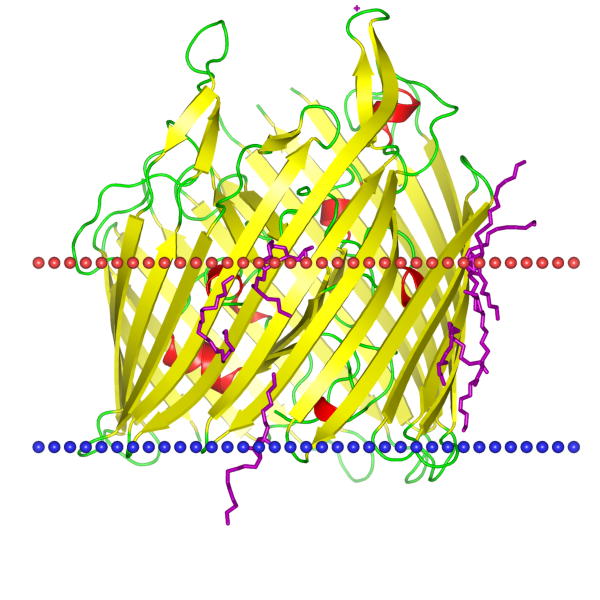
Рис. 1. Структура и топология белка BtuB, приведенная в OPM.
Координаты трансмембранных участков белка, приведенные в OPM:
1(136-145), 2(149-158), 3(164-173), 4(201-209), 5(214-222), 6(249-257), 7(261-269), 8(297-306), 9(309-316), 10(339-348), 11(351-359), 12(371-378), 13(383-390), 14(418-426), 15(429-436), 16(464-472), 17(475-482), 18(503-510), 19(514-521), 20(547-554), 21(559-566), 22(586-593).
Для проведения сравнения была взята fasta последовательность трансмембранного белка с UniProt.
Координаты трансмембранных участков белка, предсказанные DeepTMHMM: 1(159-166), 2(169-177), 3(186-193), 4(220-228), 5(235-242), 6(269-276), 7(282-288), 8(317-324), 9(329-336), 10(360-367), 11(371-378), 12(391-399), 13(403-410), 14(438-444), 15(450-456), 16(484-490), 17(496-502), 18(523-528), 19(535-542), 20(566-572), 21(580-589), 22(605-612).
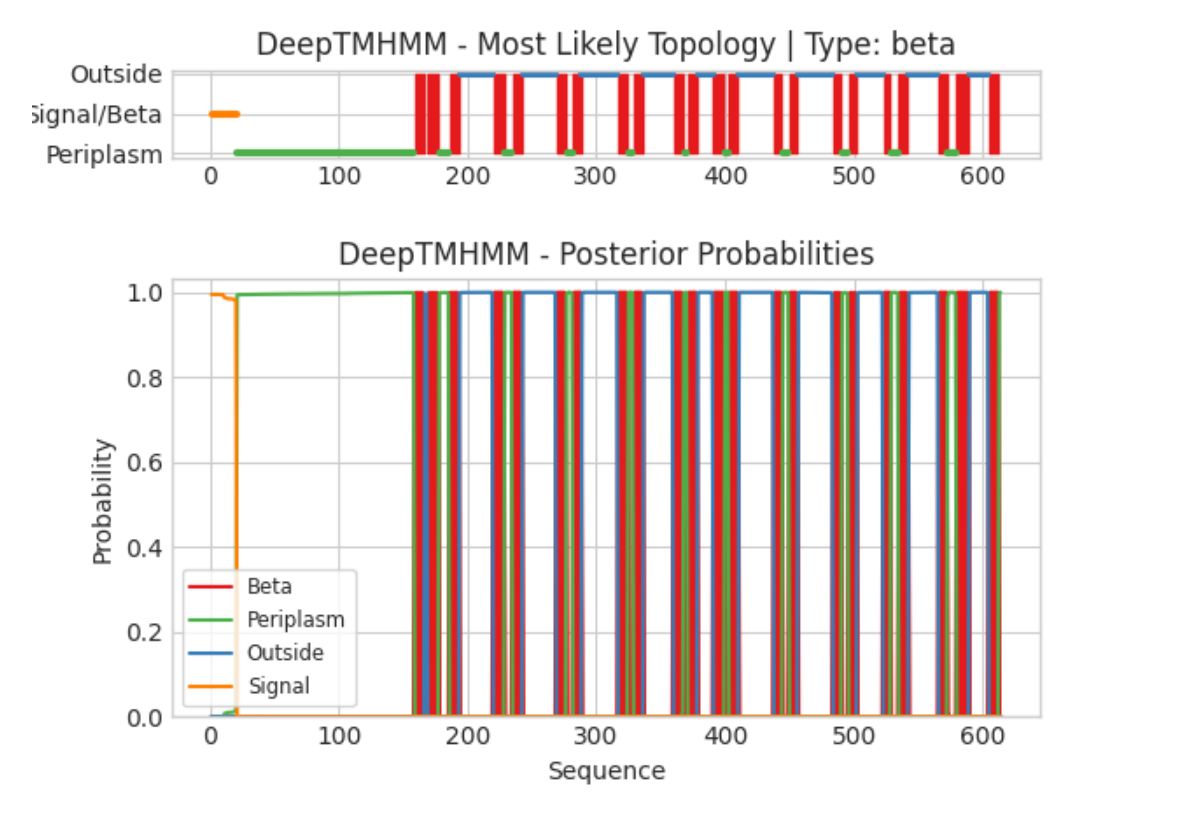
Рис. 2. Графическое представление предсказания DeepTMHMM для белка BtuB. На верхнем графике изображена наиболее вероятная топология белка. На нижнем — вероятности той или иной топологии. По оси X — последовательность белка. Красным отображены трансмембранные участки, синим - участки в наружной среде, зеленым - в периплазматическом пространстве, оранжевым - сигнальная последовательность.
Было предсказано 22 трансмембранных сегмента, что соответствует данным OPM. Но наблюдается смещение координат сегментов между предсказаниями (в среднем на 20 аминокислот). Это объясняется тем, что в OPM использовалась последовательность (594 АА) из PDB-структуры, а в DeepTMHMM анализировалась последовательность из UniProt (614 АА), включающая N-концевую сигнальную последовательность (= 20 АА). Только один трансмембранный участок полностью совпадает (371-378) и еще четыре участка перекрываются более чем на три аминокислоты.
Сравнение предсказаний трансмембранных участков в альфа-спиральном белке
Для выполнения этого задания был выдан белок Multidrug resistance protein pgp-1. Информация о белке представлена ниже:
Таблица 2. Информация о белке Multidrug resistance protein pgp-1.
Название белка (русский)
Белок pgp-1, обеспечивающий множественную лекарственную устойчивость
Название белка (английский)
Multidrug resistance protein pgp-1
Идентификатор PDB
4f4c
Идентификатор UniProt
PGP1_CAEEL
Организм
Caenorhabditis elegans
Мембрана
Эукариотическая плазматическая мембрана
Функция белка
Энергозависимый дренажный насос, ответственный за снижение накопления лекарственного средства в клетках с множественной лекарственной устойчивостью.
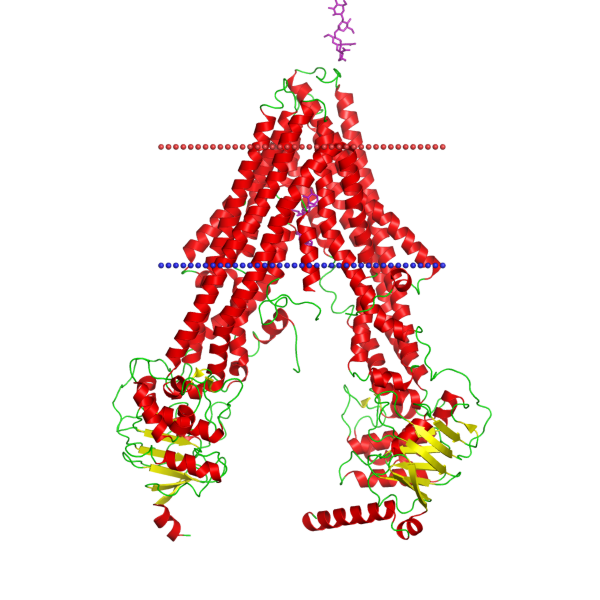
Рис. 3. Структура белка Multidrug resistance protein pgp-1 из базы данных OPM.
Координаты трансмембранных участков белка, приведенные в OPM: 1( 9- 17), 2( 23- 32), 3( 71- 99), 4( 138- 161), 5( 211- 234), 6( 238- 260), 7( 313- 338), 8 ( 356- 376), 9( 750- 776),10( 797- 821),11( 870- 894),12( 898- 917),13( 976-1000),14(1018-1038).
Для проведения сравнения была взята fasta последовательность трансмембранного белка с UniProt.
Координаты трансмембранных участков белка, предсказанные DeepTMHMM: 1(75- 79), 2( 143- 162), 3( 214- 233), 4( 240- 260), 5( 316- 340), 6( 359- 376), 7( 751- 775), 8( 798- 818), 9( 874- 894), 10( 897- 915), 11( 985-1006),12(1018-1038).
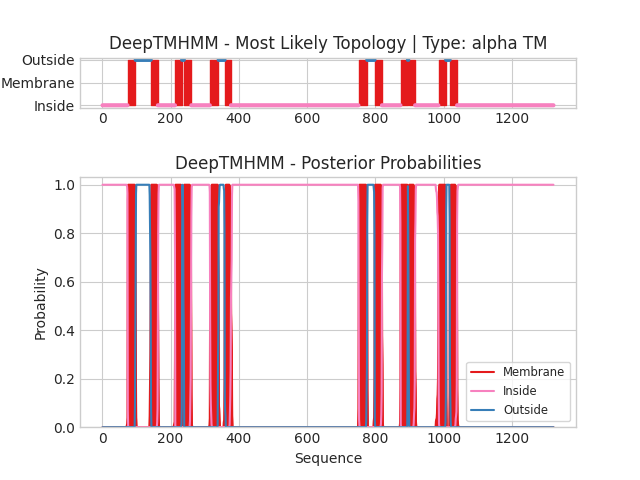
Рис. 3. Графическое представление предсказания DeepTMHMM для белка Multidrug resistance protein pgp-1. На верхнем графике изображена наиболее вероятная топология белка. На нижнем — вероятности той или иной топологии. По оси X — последовательность белка.
В C-концевой области различия находятся в пределах погрешности (5-10 АА). Только один трансмембранный участок полностью совпадает (1018-1038), что может говорить об особой структурной и функциональной важности этого домена для работы белка. Две α-спирали оказались полностью "не замечены". Наибольшие расхождения наблюдаются в N-концевых участках, можно предположить, что это первые два участка границ спиралей, предсказанных OPM, так как при сравнении координат трансмембранных участков белка, предсказанных DeepTMHMM и OPM (без учета этих участков), в границах спиралей наблюдается расхождение не больше 5-10 AA.
Практикум 7
Сравнение предсказаний трансмембранных участков в бета-листовом белке
Для выполнения данного задания был выбран белок BtuB. Ниже представлена информация о белке:
| Название белка (русский) | Транспортер кобаламина для наружной мембраны BtuB |
|---|---|
| Название белка (английский) | Outer membrane cobalamin transporter BtuB |
| Идентификатор PDB | 1NQE |
| Идентификатор UniProt | BTUB_ECOLI |
| Организм | Escherichia coli (strain K12) |
| Мембрана | Бактериальная грамотрицательная наружная мембрана |
| Функция белка | Белок является транспортером витамина B12 через наружную мембрану в периплазматическое пространство. |

Координаты трансмембранных участков белка, приведенные в OPM: 1(136-145), 2(149-158), 3(164-173), 4(201-209), 5(214-222), 6(249-257), 7(261-269), 8(297-306), 9(309-316), 10(339-348), 11(351-359), 12(371-378), 13(383-390), 14(418-426), 15(429-436), 16(464-472), 17(475-482), 18(503-510), 19(514-521), 20(547-554), 21(559-566), 22(586-593).
Для проведения сравнения была взята fasta последовательность трансмембранного белка с UniProt.
Координаты трансмембранных участков белка, предсказанные DeepTMHMM: 1(159-166), 2(169-177), 3(186-193), 4(220-228), 5(235-242), 6(269-276), 7(282-288), 8(317-324), 9(329-336), 10(360-367), 11(371-378), 12(391-399), 13(403-410), 14(438-444), 15(450-456), 16(484-490), 17(496-502), 18(523-528), 19(535-542), 20(566-572), 21(580-589), 22(605-612).

Было предсказано 22 трансмембранных сегмента, что соответствует данным OPM. Но наблюдается смещение координат сегментов между предсказаниями (в среднем на 20 аминокислот). Это объясняется тем, что в OPM использовалась последовательность (594 АА) из PDB-структуры, а в DeepTMHMM анализировалась последовательность из UniProt (614 АА), включающая N-концевую сигнальную последовательность (= 20 АА). Только один трансмембранный участок полностью совпадает (371-378) и еще четыре участка перекрываются более чем на три аминокислоты.
Сравнение предсказаний трансмембранных участков в альфа-спиральном белке
Для выполнения этого задания был выдан белок Multidrug resistance protein pgp-1. Информация о белке представлена ниже:
| Название белка (русский) | Белок pgp-1, обеспечивающий множественную лекарственную устойчивость |
|---|---|
| Название белка (английский) | Multidrug resistance protein pgp-1 |
| Идентификатор PDB | 4f4c |
| Идентификатор UniProt | PGP1_CAEEL |
| Организм | Caenorhabditis elegans |
| Мембрана | Эукариотическая плазматическая мембрана |
| Функция белка | Энергозависимый дренажный насос, ответственный за снижение накопления лекарственного средства в клетках с множественной лекарственной устойчивостью. |

Координаты трансмембранных участков белка, приведенные в OPM: 1( 9- 17), 2( 23- 32), 3( 71- 99), 4( 138- 161), 5( 211- 234), 6( 238- 260), 7( 313- 338), 8 ( 356- 376), 9( 750- 776),10( 797- 821),11( 870- 894),12( 898- 917),13( 976-1000),14(1018-1038).
Для проведения сравнения была взята fasta последовательность трансмембранного белка с UniProt.
Координаты трансмембранных участков белка, предсказанные DeepTMHMM: 1(75- 79), 2( 143- 162), 3( 214- 233), 4( 240- 260), 5( 316- 340), 6( 359- 376), 7( 751- 775), 8( 798- 818), 9( 874- 894), 10( 897- 915), 11( 985-1006),12(1018-1038).

В C-концевой области различия находятся в пределах погрешности (5-10 АА). Только один трансмембранный участок полностью совпадает (1018-1038), что может говорить об особой структурной и функциональной важности этого домена для работы белка. Две α-спирали оказались полностью "не замечены". Наибольшие расхождения наблюдаются в N-концевых участках, можно предположить, что это первые два участка границ спиралей, предсказанных OPM, так как при сравнении координат трансмембранных участков белка, предсказанных DeepTMHMM и OPM (без учета этих участков), в границах спиралей наблюдается расхождение не больше 5-10 AA.