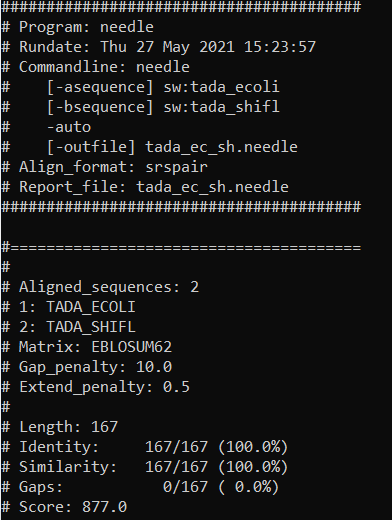
Рис. 1. Результаты выравнивания TADA_ECOLI и TADA_SHIFL, полученные с использованием команды needle.
Я создал два файла с белками кишечной и сенной палочек следующими командами:
infoseq sw:'*_ECOLI' -only -name -noheading -out ECOLI.txt
infoseq sw:'*_BACSU' -only -name -noheading -out BACSU.txt
Далее с помощью скрипта, написанного на Python, я получил список белков данных бактерий с одинаковой мнемоникой функции. Из полученных мнемоник для дальнейшей работы я выбрал 3: SSUB, TADA, SYS.
Результаты глобального парного выравнивания трёх пар белков представлены в таблице 1.
| Таблица 1. Характеристики глобального парного выравнивания трёх пар белков | |||||||
|---|---|---|---|---|---|---|---|
| Protein Name | ID 1 | ID 2 | Score | % Identity | % Similarity | Gaps | Indels |
| Aliphatic sulfonates import ATP-binding protein SsuB | SSUB_ECOLI | SSUB_BACSU | 349.0 | 33.3 | 53.7 | 11.1 | 11 |
| tRNA-specific adenosine deaminase | TADA_ECOLI | TADA_BACSU | 332.5 | 41.9 | 61.1 | 3.6 | 2 |
| Serine--tRNA ligase | SYS_ECOLI | SYS_BACSU | 1093.5 | 51.6 | 67.1 | 2.1 | 4 |
Результаты локального парного выравнивания трёх пар белков представлены в таблице 2.
| Таблица 2. Характеристики локального парного выравнивания трёх пар белков | |||||||||
|---|---|---|---|---|---|---|---|---|---|
| Protein Name | ID 1 | ID 2 | Score | % Identity | % Similarity | Gaps | Indels | % Coverage1 | % Coverage2 |
| Aliphatic sulfonates import ATP-binding protein SsuB | SSUB_ECOLI | SSUB_BACSU | 358.5 | 37.7 | 59.3 | 5.6 | 6 | 87.84 | 88.24 |
| tRNA-specific adenosine deaminase | TADA_ECOLI | TADA_BACSU | 337.0 | 44.4 | 64.1 | 0 | 0 | 91.62 | 95.03 |
| Serine--tRNA ligase | SYS_ECOLI | SYS_BACSU | 1095.5 | 52.1 | 67.5 | 1.9 | 3 | 99.30 | 99.06 |
Результаты глобального парного выравнивания неродственной пары белков представлены в таблице 3.
| Таблица 3. Характеристики глобального парного выравнивания неродственной пары белков | ||||||||
|---|---|---|---|---|---|---|---|---|
| Protein Name 1 | Protein Name 2 | ID 1 | ID 2 | Score | % Identity | % Similarity | Gaps | Indels |
| Trans-2-enoyl-CoA reductase [NADH] | Serine--tRNA ligase | FABV_TREDE | SYS_ECOLI | 45.0 | 6.2 | 11.1 | 77.8 | 13 |
Результаты глобального парного выравнивания неродственной пары белков представлены в таблице 4.
| Таблица 4. Характеристики глобального парного выравнивания неродственной пары белков | ||||||||||
|---|---|---|---|---|---|---|---|---|---|---|
| Protein Name 1 | Protein Name 2 | ID 1 | ID 2 | Score | % Identity | % Similarity | Gaps | Indels | % Coverage1 | % Coverage2 |
| Trans-2-enoyl-CoA reductase [NADH] | Serine--tRNA ligase | FABV_TREDE | SYS_ECOLI | 52.5 | 18.4 | 32.5 | 35.4 | 7 | 47.86 | 36.98 |
Для последующего выравнивания я выбрал мнемонику функции "TADA"(tRNA-specific adenosine deaminase). В Swiss-Prot нашлось 25 белков с такой мнемоникой. Кроме TADA_ECOLI и TADA_BACSU были выбраны следующие 5 белков: TADA_BUCAP, TADA_STRP6, TADA_HAEIN, TADA_STAAM и TADA_SHIFL.
Множественное выравнивание было проведено с помощью программы muscle, после чего полученный .fasta файл был импортирован в Jalview (проект доступен для скачивания по ссылке).
Разброс длины белков невелик - длины варьируются в диапазоне от 151 до 173 аминокислот. В выравнивании видны длинные консервативные блоки (в районе от 60 до 151 аминокислоты). Исходя из этого можно говорить о том, что все белки, учавствующие в выравнивании гомологичны. Отдельно стоит отметить последовательности TADA_ECOLI и TADA_SHIFL, принадлежащие, соответственно бактериям Escherichia coli и Shigella flexneri (обе бактерии входят в семейство Enterobacteriaceae) - эти последовательности идентичны на 100% согласно выравниванию, полученному с помощью программы needle.

Рис. 1. Результаты выравнивания TADA_ECOLI и TADA_SHIFL, полученные с использованием команды needle.