Транс-2-еноил-КоА-редуктаза относится к классу оксидоредуктаз, представляет собой мономер, связывает НАД. Этот фермент участвует в синтезе жирных кислот в цитоплазме - катализирует реакцию восстановления двойной связи в молекуле кротонил-КоА, в результате чего получается бутирил-КоА. Интересно, что эта изоформа фермента, несмотря на то, что обладает высокой активностью в отношении реакции восстановления, использует в качестве лиганда НАДН, а не НАДФН. Продукт гена fabV [1],[2].
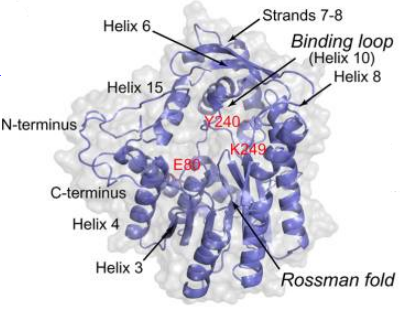
Данный белок состоит из 397 аминокислотных остатков, является мономерным и включает в себя 15 альфа-спиралей и 10 бета-слоев. Из структурных мотивов в нем можно выделить укладку Россмана (типична для НАД/НАДФ/ФАД-связывающих белков) и НАД-связывающую петлю (рис. 1) [2],[3].

Данный фермент был выделен из бактерии Treponema denticola, штамм ATCC 35405/CIP 103919/DSM 14222.
| Классификация организма | |
|---|---|
| Домен | Bacteria |
| Отдел | Spirochaetes |
| Класс | Spirochaetia |
| Порядок | Spirochaetales |
| Семейство | Spirochaetaceae |
| Род | Treponema |
| Вид | Treponema denticola |
Treponema denticola - спирохета, подвижная, грамотрицательная, протеолитическая бактерия. Является возбудителем пародонтита, а секреция ею пептидиларгинин-деиминазы может привести к раку поджелудочной железы, вероятно, это сделало бактерию объектом интереса учёных [4].
| Информация о данном белке, полученная из UniProt | |
|---|---|
| Рекомендуемое название | Trans-2-enoyl-CoA reductase [NADH] |
| Длина белка | 397 аминокислот |
| Молекулярная масса | 43759 Да |
| UniProtKB | Swiss-Prot |
| UniProt ID | FABV_TREDE |
| UniProt AC | Q73Q47 |
| EMBL AC | AE017226 |
| PDB ID | 4FBG, 4GGO, 4GGP |
Теперь воспользуюсь расширенным поиском
| Результаты расширенного поиска в UniProt | |||
|---|---|---|---|
| Запрос | Результаты | Swiss-Prot | TrEMBL |
| gene:fabv AND reviewed:yes | Все продукты гена fabV из разных организмов, записи о которых получены из Swiss-Prot | 109 | 0 |
| gene:fabv AND reviewed:no | Все продукты гена fabV из разных организмов, записи о которых получены из TrEMBL | 0 | 5131 |
| taxonomy:"Spirochaetes [203691]" gene:fabv NOT organism:"Treponema denticola (strain ATCC 35405 / CIP 103919 / DSM 14222) [243275]" existence:"Inferred from homology [3]" | Белки-продукты гена fabV бактерий из отдела Spirochaetes, кроме Treponema denticola, существование которых предсказано на основе гомологии | 1 | 140 |
| annotation:(type:disease "periodontal disease") | Белки, в аннотации которых присутствует упоминание пародонтита - болезни, которую вызывает Treponema denticola | 4 | 0 |
| NOT gene:fabv AND organism:"Treponema denticola (strain ATCC 35405 / CIP 103919 / DSM 14222) [243275]" | Белки-продукты генов Treponema denticola, кроме продукта гена fabV | 256 | 2497 |
| name:"trans-2-enoyl-coa reductase" NOT gene:fabv | Транс-2-еноил-КоА-редуктазы из разных организмов, не являющиеся продуктами гена под названием fabV | 7 | 1647 |
Переход к кластерам последовательностей UniRef я осуществил через поиск в верхней части страницы (UniRef> "fabv trede").
Помимо различных бактерий рода Treponema, кластер UniRef50 включает в себя такие организмы, как Myroides odoratus (отдел Bacteroidetes), Ferrimonas marina (отдел Proteobacteria, класс Gammaproteobacteria), Rickettsiales sp. (отдел Proteobacteria, класс Alphaproteobacteria), из чего можно сделать вывод, что данный белок весьма распространен (что логично, учитывая важность синтеза ЖК в метаболизме).
В кластерах присутствует самая длинная последовательность (seed) и наиболее изученная (representative) последовательность. Данный белок является репрезентативной последовательностью. Замечу, что последовательность seed больше репрезентативной на 8 аминокислотных остатков (причем последние 6 из этих аминокислот - гистидины), принадлежит к тому же организму и кодирует тот же белок; с ее страницы ссылки направляют на сайт PDB, где представлена одна из структур Транс-2-еноил-КоА-редуктазы, полученная в ходе кристаллографии. Таким образом, по сути seed и representative являются одной и той же последовательностью.
| Кластеры UniRef | |
|---|---|
| ID Кластера | Размер кластера |
| UniRef50_Q73Q47 | 722 |
| UniRef90_Q73Q47 | 14 |
| UniRef100_Q73Q47 | 3 |
Проведу сравнение протеомов Treponema denticola и Porphyromonas gingivalis. Последний вид был выбран, так как эта бактерия также является возбудителем пародонтита. Оба протеома являются референсными протеомами.
| Сравнение протеомов в UniProt | |||||
|---|---|---|---|---|---|
| Proteome ID | Organism | Protein count | Swiss-Prot | Complete Proteome Detector (CPD) | Genome representation (RefSeq) |
| UP000008212 | Treponema denticola (strain ATCC 35405 / CIP 103919 / DSM 14222) | 2753 | 257 | Close to standard (high) | full |
| UP000000588 | Porphyromonas gingivalis (strain ATCC BAA-308 / W83) | 1863 | 281 | Standard | full |
Рассмотрим функциональные группы белков этих двух видов бактерий.
| Информация о функциональных группах белков | |||
|---|---|---|---|
| Запрос | Результаты | Swiss-Prot | TrEMBL |
| annotation:(type:transmem) AND organism:"Treponema denticola (strain ATCC 35405 / CIP 103919 / DSM 14222) [243275]" AND proteome:up000008212 | Трансмембранные белки Treponema denticola | 9 | 585 |
| ec:* AND organism:"Treponema denticola (strain ATCC 35405 / CIP 103919 / DSM 14222) [243275]" AND proteome:up000008212 | Ферменты Treponema denticola | 146 | 207 |
| protein-arginine deiminase activity AND organism:"Treponema denticola (strain ATCC 35405 / CIP 103919 / DSM 14222) [243275]" AND proteome:up000008212 | Белки Treponema denticola c пептидиларгинин-деиминазной активностью | 0 | 0 |
| annotation:(type:transmem) AND organism:"Porphyromonas gingivalis (strain ATCC BAA-308 / W83) [242619]" AND proteome:up000000588 | Трансмембранные белки Porphyromonas gingivalis | 13 | 310 |
| ec:* AND organism:"Porphyromonas gingivalis (strain ATCC BAA-308 / W83) [242619]" AND proteome:up000000588 | Ферменты Porphyromonas gingivalis | 170 | 206 |
| protein-arginine deiminase activity AND organism:"Porphyromonas gingivalis (strain ATCC BAA-308 / W83) [242619]" AND proteome:up000000588 | Белки Porphyromonas gingivalis c пептидиларгинин-деиминазной активностью | 1 | 1 |
Как видно из полученных данных, белков с трансмембранной активностью у Treponema denticola больше (однако и белков в протеоме Treponema denticola больше, чем у Porphyromonas gingivalis), количество ферментов у этих двух видов примерно одинаково. Отмечу, что ферменты у обоих видов бактерий описаны вручную (то есть записи о них из Swiss-Prot) в процентном отношении больше, чем трансмембранные белки. В качестве третьей функциональной группы я выбрал белки с пептидиларгинин-деиминазной активностью, по причине описанной ранее - согласно литературным данным ([4]), секреция пептидиларгинин-деиминазы бактериями, вызывающими пародонтит, в том числе и Treponema denticola, возможно, провоцирует развитие рака поджелудочной железы. К сожалению, записей в UniProt о пептидиларгинин-деиминазе в протеоме Treponema denticola нет, в то время как есть 2 записи об этом ферменте у Porphyromonas gingivalis.
1. Hu K., Zhao M., Zhang T., Zha M., Zhong C., Jiang Y., Ding J. Structures of trans-2-enoyl-CoA reductases from Clostridium acetobutylicum and Treponema denticola: insights into the substrate specificity and the catalytic mechanism. Biochem. J. 449:79-89(2013)
2. Bond-Watts B.B., Weeks A.M., Chang M.C. Biochemical and structural characterization of the trans-enoyl-CoA reductase from Treponema denticola. Biochemistry 51:6827-6837(2012)
3. Israel Hanukoglu. Proteopedia: Rossmann fold: A beta-alpha-beta fold at dinucleotide binding sites. Multimedia in Biochemistry and Molecular Biology Education Volume43, Issue3 May/June 2015 Pages 206-209
4. Öğrendik M. Periodontal Pathogens in the Etiology of Pancreatic Cancer. Gastrointest Tumors. 2017 Mar;3(3-4):125-127