
В базе данных OPM я выполнил запрос "porin" и выбрал первый белок из результатов поиска. Им оказался порин F внешней мембраны (Outer membrane porin F) Грам-отрицательной бактерии Pseudomonas aeruginosa. PDB ID: 4rlc; Uniprot ID: PORF_PSEAE
| Таблица 1. Информация о порине F внешней мембраны из БД OPM | |
|---|---|
| Толщина гидрофобной части белка в мембране | 24.2 Å |
| Локализация | Внешняя мембрана Грам-(-) бактерии |
| Координаты трансмембранных участков | 1(5-14), 2(28-35), 3(43-48), 4(68-77), 5(84-94), 6(112-122), 7(129-137), 8(150-158) |
| Среднее количество остатков в одном β-тяже белка | 8-10 аминокислот |
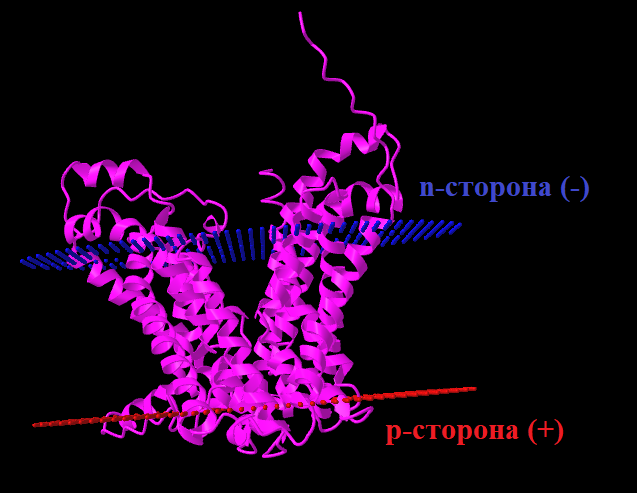
На рисунке снизу внешняя сторона мембраны (p-сторона) обращена вниз, хорошо виден β-цилиндр, образованный восемью β-листами.

Для дальнейшей работы я использовал выданный мне α-спиральный белок THIP_BRUA2 и β-цилиндрический белок PORF_PSEAE из прошлого задания этого практикума.
Перед обсуждением результатов вычислений DeepTMHMM приведу название выданного α-спирального белка - пермеаза системы транспорта тиамина ThiP (Thiamine transport system permease protein ThiP) Грам-отрицательной бактерии Brucella abortus. UniProt ID: THIP_BRUA2.
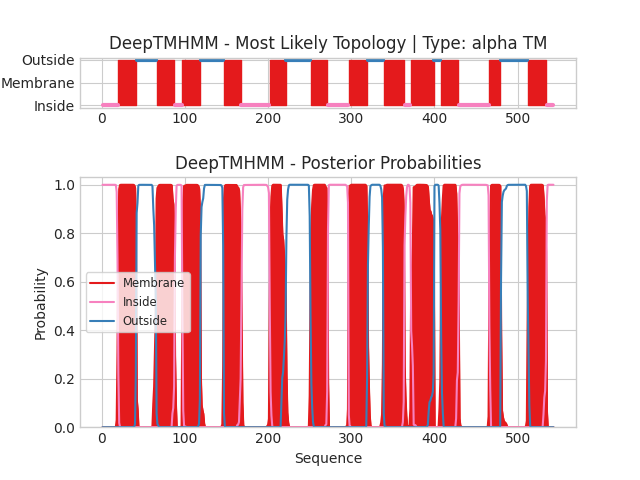
Изображение выше показывает выдачу DeepTMHMM для α-спирального белка ThiP. Верхний график показывает наиболее вероятное для каждого из остатков положение: с внешней, внутренней стороны клетки или в мембране. На нижнем графике показана вероятность нахождения остатка в одном из трех положений. Красным цветом показана вероятность нахождения остатка в мембране, розовым - внутри, синим - снаружи клетки. Отмечу, что алгоритм самостоятельно правильно определил, что мембранная часть белка состоит из α-спиралей.

Выше изображена выдача DeepTMHMM для β-цилиндрического белка порина F. Аналогично предыдущей картинке, Верхний график показывает наиболее вероятное для каждого из остатков положение: с внешней, внутренней стороны клетки или в мембране. На нижнем графике показана вероятность нахождения остатка в одном из трех положений. Красным цветом показана вероятность нахождения остатка во внешней мембране (в β-листе), зеленым - в периплазме (межмембранном пространстве), синим - снаружи клетки, светло-оранжевым - в сигнальном пептиде. Отмечу, что алгоритм самостоятельно правильно определил, что мембранная часть белка представлена структурой из β-листов.
Выдача в текстовом виде: пермеаза ThiP, порин F.
Название α-спирального белка и его идентификатор в Uniprot были указаны в прошлом задании. Согласно UniProt, данный белок расположен во внутренней мембране бактерии. По графической выдаче DeepTMHMM для данного белка (см. задание 2) видим, что аминокислоты на N-конце с высокой вероятностью располагаются внутри клетки (т.е. N-конец находится в цитоплазме). Параметры запуска приведены в таблице ниже.
| Таблица 2. Параметры запуска PPM для выданного белка | |
|---|---|
| Число мембран | 1 |
| Тип мембраны | Внутренняя мембрана Грам-(-) бактерии |
| Allow curvature | Нет |
| Топология (N-концевой домен) | Внутри |
| Включить гетероатомы (для нестандартных аминокислотных остатков) | Нет |
Выдача PMM представлена в таблице ниже.
| Таблица 3. Информация о пермеазе ThiP, вычисленная PPM | |
|---|---|
| Толщина гидрофобной части белка в мембране | 29.9 ± 0.8 Å |
| Локализация | Внутренняя мембрана Грам-(-) бактерии |
| Координаты трансмембранных участков | 1(18-41), 2(58-84), 3(95-123), 4(146-165), 5(202-224), 6(250-271), 7(297-324), 8(335-360), 9(375-399),10(407-429),11(466-489),12(512-533) |
| Среднее количество остатков в одном β-тяже белка | 23 аминокислоты |
Сравнение DeepTMHMM и PPM (α-спиральный белок ThiP) начнем с количества трансмембранных структур- в обоих случаях алгоритмы предсказали существование 12 α-спиральных трансмембранных элементов - это согласуется и с данными из UniProt.
В таблице ниже привожу позиции α-спиралей из DeepTMHMM, PPM и Uniprot.
| Таблица 4. Координаты трансмембранных участков пермеазы ThiP | |||
|---|---|---|---|
| № | DeepTMHMM | PPM | UniProt |
| 1 | 20-41 | 18-41 | 19-39 |
| 2 | 66-87 | 58-84 | 64-84 |
| 3 | 97-118 | 95-123 | 102-122 |
| 4 | 147-167 | 146-165 | 142-162 |
| 5 | 202-221 | 202-224 | 205-225 |
| 6 | 251-271 | 250-271 | 250-270 |
| 7 | 297-319 | 297-324 | 300-320 |
| 8 | 339-363 | 335-360 | 343-363 |
| 9 | 372-399 | 375-399 | 379-399 |
| 10 | 408-428 | 407-429 | 406-426 |
| 11 | 466-479 | 466-489 | 468-488 |
| 12 | 512-534 | 512-533 | 510-530 |
В целом оба метода предсказывают координаты трансмембранных участков достаточно точно (близко к данным из UniProt, сложно сказать, какой из методов предсказания точнее).
Сравнение DeepTMHMM и OPM (порин F) начнем с количества трансмембранных структур - DeepTMHMM правильно предсказал наличие 8 бета-листов.
| Таблица 4. Координаты трансмембранных участков пермеазы ThiP | ||
|---|---|---|
| № | DeepTMHMM | OPM |
| 1 | 30-38 | 5-14 |
| 2 | 52-61 | 28-35 |
| 3 | 65-72 | 43-48 |
| 4 | 93-100 | 68-77 |
| 5 | 109-117 | 84-94 |
| 6 | 137-145 | 112-122 |
| 7 | 152-159 | 129-137 |
| 8 | 175-182 | 150-158 |
Как видно из таблицы выше, DeepTMHMM сместил бета-листы - 1, похоже, соответствует 2 в OPM, такая же ситуация наблюдается с 7 и 8. Я предполагаю, что в OPM представлена последовательность зрелого белка без сигнального пептида, а в DeepTMHMM была загружена последовательность с сигнальным пептидом - это объясняет смещение координат бета-листов.

На рисунке выше представлена структура, предсказанная AlphaFold. Достоверно предсказаны трансмембранные участки α-спиралей, с низкой точностью предсказана структура N-концевого домена. В целом качество предсказания - хорошее.
Поскольку трансмембранные участки предсказаны AlphaFold достоверно (pLDDT > 90), а низкое качество имеют участки, лежащие вне мембраны, я полагаю, что наличие участков с низким качеством предсказания структуры не повлияло на работу сервера PPM.
UniProt AC белка ThiP - Q2YLW7; UniProt AC порина F - P13794. Первый белок не был найден в базе данных TCDB. Второй белок внесен в эту базу данных. Выдача внесена в таблицу ниже.
| Таблица 5. Параметры запуска PPM для выданного белка | |
|---|---|
| TC-код | 1.B.6.1.2 |
| Название белка | PORF aka OmpF aka OprF aka PA1777 |
| Длина | 350 аминокислот |
| Молекулярная масса | 37640.00 Да |
| Вид | Pseudomonas aeruginosa |
| Число трансмембранных сегментов | 1 |
| Локализация/Топология | Внешняя мембрана клетки/Пронизывает мембрану несколько раз(multi-pass membrane protein) |
Значение TC-кода выданного белка: 1. - каналы/поры; B. - β-цилиндрические порины; 6. - семейство OmpA-OmpF Porin (OOP); .1.2: индивидуальный код данного белка.