| Таблица 1. Выбранные бактерии | |
|---|---|
| Название | Мнемоника |
| Acidothermus cellulolyticus | ACIC1 |
| Arthrobacter sp. | ARTS2 |
| Bifidobacterium longum | BIFLO |
| Clavibacter michiganensis | CLAMS |
| Leifsonia xyli | LEIXX |
| Nocardioides sp. | NOCSJ |
| Streptomyces avermitilis | STRAW |
| Thermobifida fusca | THEFY |
Для начала работы я объединил протеомы бактерий из практикума 1 в файл proteomes.fasta. Далее с помощью blastp был проведен поиск гомологов белка CLPX_ECOLI - команды:
makeblastdb -dbtype prot -in proteomes.fasta -out bacdb
blastp -query CLPX_ECOLI.fasta -db bacdb -evalue 0.001 -out res.txt
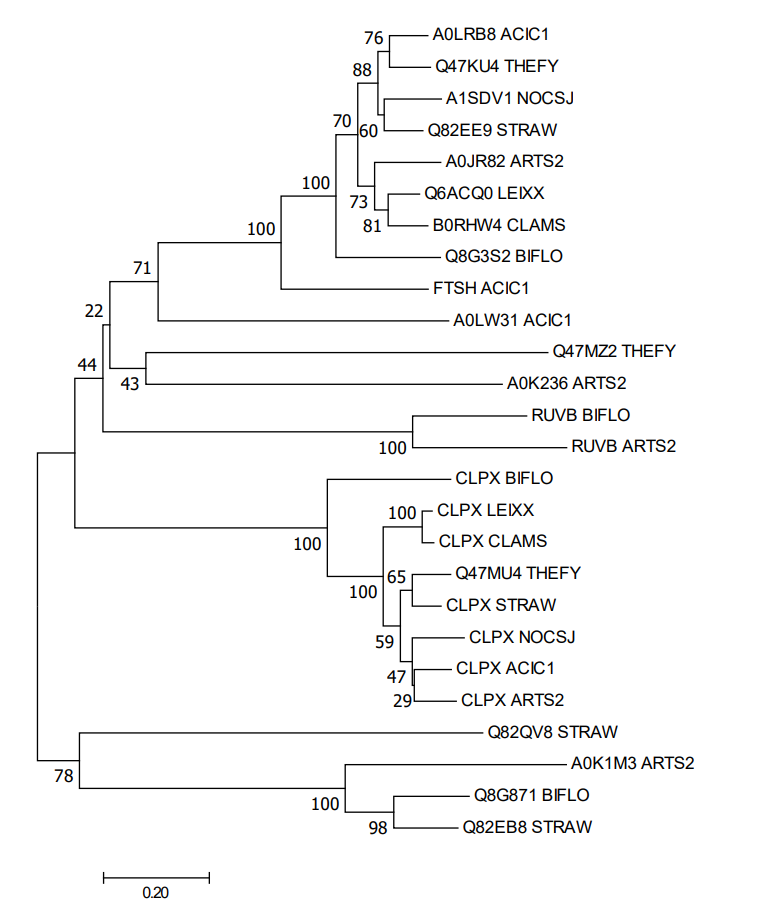
В итоге получен список находок. Из него перед построением выравнивания в MEGA7. Дерево(Newick) также было построено в программе MEGA7 методом Neighbor-Joining.



Дерево, ветви которого схлопнуты, было импортировано в MEGA11 из MEGA7, собственно, для схлопывания ветвей.
На деревьях сверху красным отмечена группа белков-ортологов CLPX - АТФ-связывающих субъединиц АТФ-зависимой протеазы Clp. С деревом из 1 практикума есть совпадающие поддеревья: (LEIXX,CLAMS); (THEFY,STRAW). В остальном топология деревьев отличается.
Зеленым отмечена группа цинк-зависимой металлопротеазы FtsH. С деревом из 1 практикума совпадает поддерево ((LEIXX, CLAMS), ARTS2). В остальном деревья отличаются.