Секвенирование ДНК по Сэнгеру и анализ хроматограмм.¶
Задание №1¶
Для выполнения работы мной были скачены прочтения прямой и обратной цепочки секвенируемой ДНК. forward_read
| Участок в начале | Участок в конце | |
| Прямая цепочка | проблем нет | 727-836 нуклеотид |
| Обратная цепочка | 1-179 нуклеотид | проблем нет |
Forward-прочтение¶
Высокий шум мешает определить примерно 100 нуклеотидов в сиквенсе.
Reverse-прочтение¶
Высокий шум мешает определить первые 20 нуклеотидов в сиквенсе, а до 179 нуклеотида он сохраняется достаточно высоким. Минимальный уровень шума наблюдается примерно с 150 по 500 нуклеотид, к 550 букве он повышается, но детектировать нуклеотиды все еще возможно. примерно после 800 нуклеотида детекция уже невозможна.
Уровень шума по отношению к уровню сигнала определялся на глаз. Уровень шума для нуклеотидов до 179 и после 800 позиций составляет более 40%, что мешает интерпретировать результаты секвенирования.В центральной области секвенирования уровень шума не превышает 10%.
Общая оценка¶
Визуальная оценка неравномерности сигнала: для прямого и обратного прочтений характерны соотносимые по высоте пики в середине и сильно разбросанные по высоте пики в начале и конце обоих последовательностей.
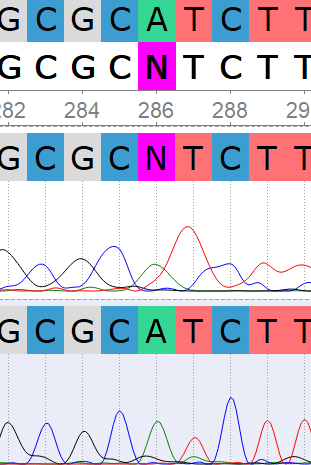
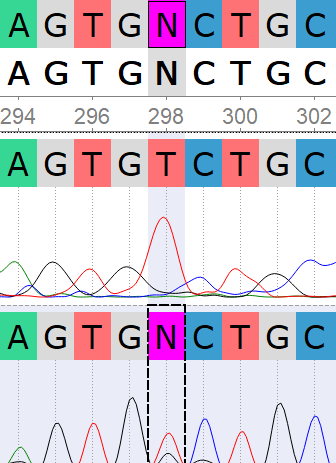
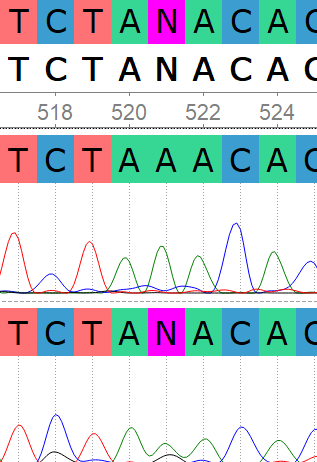
Визуальная оценка неравномерности шума: в начале и конце прямого и обратного прочтений пики имеют разнообразную,пересекаются или сливаются, в середине последовательности пики а хроматограмме хорошо различимы, сигналы небольшого фонового загрязнения не мешают детекции. Вырезанные проблемные участки каждого прочтения можно восстановить по другой цепи.
*За референсную было взято обратное прочтение для более полного результата, такое выравнивание выглядило лучше.
Итак, с помощью программы UGENE был получен консенсус.
Выравнивание¶
После выравнивания прямого и обратного прочтения, редактирования всех нечитаемых участков вручную, с помощью blastn NCBI была найдена последовательность генов идентичная консенсусной — KR920030.1 Scoloplos acutissimus voucher W.44175.001 18S ribosomal RNA gene, partial sequence . Reference sequence
Выравнивание на референс:
Решения по проблемным нуклеотидам¶