 Terms
Terms
|
 Other results
Other results
|
 Home page
Home page
|
| |
|
Бутстреп-анализ выдал вот
такое консенсусное дерево:
+---------------------------C
|
| (1) +------F
| (2) +-72.0-|
| +-69.0-| +------E
| | |
+------| +-------------D
|
| (3) +------B
+-------100.0-|
+------A
|
A B C D E F
(1) . . . . * *
(2) . . . * * *
(3) * * . . . .
_________________________
Разметка реальных деревьев:
A B C D E F
(1) . . * * * *
(2) . . . * * *
(3) . . . . * *
|
Если же рассматривать бутстреп значения внутренних ветвей реального дерева(на основе предыдущей выдачи) то получим такое изображение:
+--A
|
+---100.0--+
| |
+-+69.0 +--B
| |
| +-------------C
+-+72.0
| |
| +--------------D
+------+
| |
| +--------------E
-+
|
+--------------------F
Как видно, несмотря на внешнии различия деревьев топология их совпадает, что доказывает
полная запись их разбиений. Бутстреп значения ветки АВ 100 в то время как остальных значительно меньше: вероятно это связано,
с тем, что наши последовательности создавались на основе случайных мутаций, а расстояния между предками
АВС и АВСD было невелико, поэтому последовательности C, D, E, F сильно похожи друг на друга и следовательно в некоторых случаях они объединялись в разные ветки - что и дало низкие бутстреп значения.
Данные по выдачам программ в этом случае, вы можете достать по соответсвующим ссылкам.
Второе задание
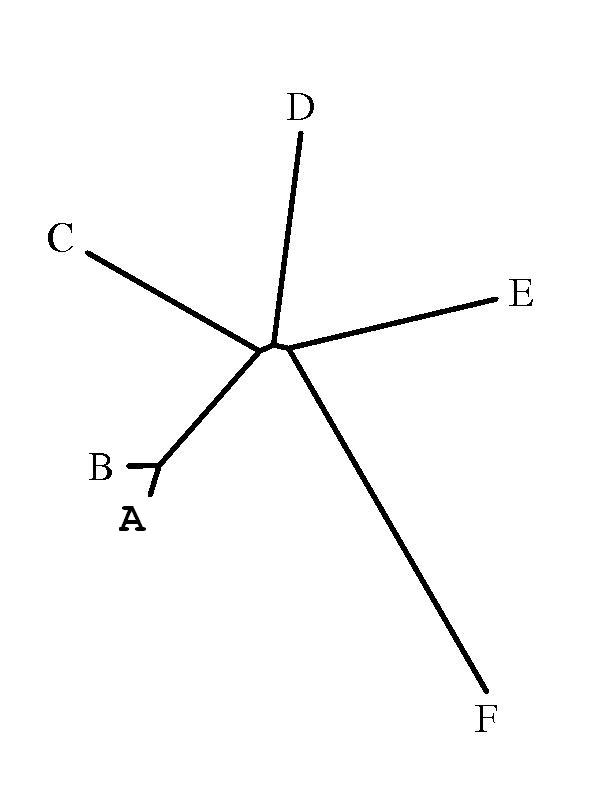
Данная программа хорошо и наглядно строит неукоренненые деревья по скобочным формулам, что сильно упрощает процесс визуализации полученных результатов.
*********************************
****Дополнительное задание*******
*********************************
1)Результатом алгоритма JackKnife оказалось такое же по топологии дерево, как и консенсус bootstrep, хотя значения
показывающие частоту появления определенных ветвей довольно сильно различаются. Это может говорить о
некоторой зависимости частоты замен от положения на последовательности. В алгоритме "случайных" замен
(К примеру в Delphi алгоритм случайных чисел - random смещен в сторону меньшей половины т.е выдает значения меньшие
0,5 с большей вероятностью, чем > 0,5 при интервале ответа [0,1]).
С другой стороны, неизменность топологии дерева может говорить о том, что эта зависимость невелика.
Если же не брать в расчет, что это дерево сгенерированно подобным путем, то по результату двух программ можно сказать,
что даная реконструкция соответвует модели.
2) С помощью программы fretree я переукоренил дерево в среднюю точку. Для этого после ее запуска с входным файлом мне потребовалось ввести символ. В интерактивном
help я нашел, что укоренение в среднюю точку задается символом M. После чего я сохранил полученное дерево обратно в тот же файл.
Надо отметить, что укоренение в среднюю точку не является в данном случае удачным алгоритмом. Т.к. он ставит корень
совсем не там, где он находился в первоначальном дереве.
3)
С помошью параметров программы fdrawgram :
-grows y - я получил горизонтальное дерево
-style p - филограмму
Полученный файл после прочтения GSView импортировался в формат JPEG:

Программа fdrawtree имеет большое параметров вывода направленных в первую очередь в удобстве просмотра полученного изображения без необходимости
переконвертациии часто вместе с форматом можно выбрать расширение. - Данная программа скорее предназначенна для печати полученных результатов сразу на принтере.
4) Помимо предложенной программы востановления первоначальной последовательности (fdnamlk) я нашел опцию
-hypstate - дающую возможность находить последовательность предка еще и программой fdnaml.
При этом самое интересное они выдают два разных дерева. И соответственно две разные предковые последовательности.
Файлы выдачи:
fdnaml
fdnamlk
C помощью needle, запущенной с высокими штрафами за открывание гэпа (т.к в этом выравнивании гэпов быть не может, и mismatch программа может интерпретировать как гэп), я построил два выравнивания.
Как видно первая программа выдает саму последовательность, строя консенсус(реальную последовательность из нуклеотидов) используя для этого неукорененое дерево.
А вторая, укоренняя, строит консенсус заменяя несогласованные в выравнивании нуклеотиды консенсусными буквами. Проценты схожести 47 и 56.
Вероятно, каждая программа удобна в своих случаях - т.к. иногда нам требуется реальная последовательность, которая могла быть
предковой, а иногда только консенсус, отражающий консервативность позиций в последовательностях потомков.
|