Составление репрезентативной выборки белка PLY_BACSU
При помощи алгоритма BLAST была осуществлена выборка гомологов. Сначала был проведен поиска гомологов среди прокариот, затем среди эукариот. В обоих случаях мы исключали филум Firmicutes, к которому относится Bacillus subtilis. Установили порог E-value равный 1е-05. Результаты поиска по данным параметрам представленны в таблице 1.
| Поиск | Алгоритм BLAST | Название базы данных | Ограничения по таксонам | Порог e-value | Максимальное количество хитов |
| По прокариотам | blastp | refseq | Не firmicutes; не Eukaryota | 1,00E-005 | 417 |
| По эукариотам | blastp | refseq | не firmicutes; Eukaryota | 1,00E-005 | 382 |
Табл.1
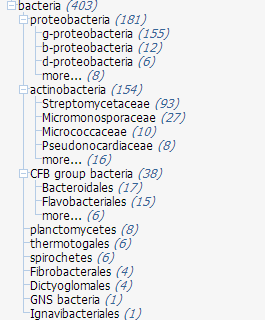
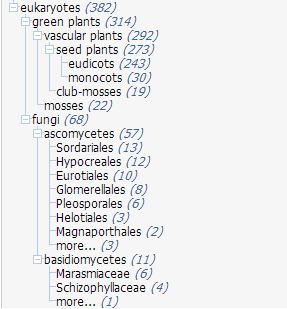
На рисунках 1 и 2 показыны деревья, которые наглядно демострируют степень схожести гомологов белка PLY_BACSU среди прокариот и эукариот соответственно.
Рис.1 Таксономические группы прокариот
Рис.2 Таксономические группы эукариот
В таблице 2 были преведены белки, которые были выбраны для множественного выравнивания.
| Домен | Царство/Филум | Название организма | Количество белков |
| Archaea | не найдено | ||
| Bacteria | proteobacteria | Alishewanella agri | 1 |
| Alishewanella jeotgali | 1 | ||
| Rheinheimera sp. A13L | 1 | ||
| Alishewanella aestuarii | 1 | ||
| actinobacteria | Streptomyces zinciresistens | 1 | |
| Streptomyces sp. W007 | 1 | ||
| Streptomyces coelicoflavus | 1 | ||
| Eucariot | green plants | Glycine max | 1 |
| Zea mays | 5 | ||
| Solanum lycopersicum | 1 | ||
| fungi | Zymoseptoria tritici IPO323 | 1 | |
| Leptosphaeria_maculans_JN3 | 1 | ||
| Schizophyllum_commune_H4-8 | 1 |
Табл.2 Выборка гомологов
Множественное выравнивание гомологов белка PLY_BACSU
Для множественного выравнивания была использованна программа muscle.
В работе применена следующая цветовая схема выравнивания:
Цветовую обработку имеют те участки, процент идентичности в которых привышает 55%. Так же на рисунке 3 показана дополнительная информация о функциях и структуре. На поле structure показаны альфа-спирали, обозначенные красным, и бета-листы, обозначенные зеленым. На поле BLOCKS символами В обозначены гомологичные блоки. На поле ligand символами L обозначены лиганды, присутствующие в белке.
Рис.3 Множественное выравнивание гомологов PLY_BACSU
Множественное выравнивание в формате fasta
Результаты анализа множественного выравнивания гомологов белка PLY_BACSU
В целом выравнивание достаточно консервативно. Самое плохое выравнивание на концах последовательностей, в ядре молекулы находятся наиболее консервативные участки.
На рисунке 4 выделены лиганты присутствующие в белке. На рисунке 5 лиганд окрашен в зеленый цвет, а связанные с ним аминокислотные остатки в соответствии с цветовой схемой выравнивания. Для этого задания установленный процент индентичности равен 20%. В результате мы видим, что Asp 223, Lys 247, Arg 284 b Ser 253 являются консервативными.
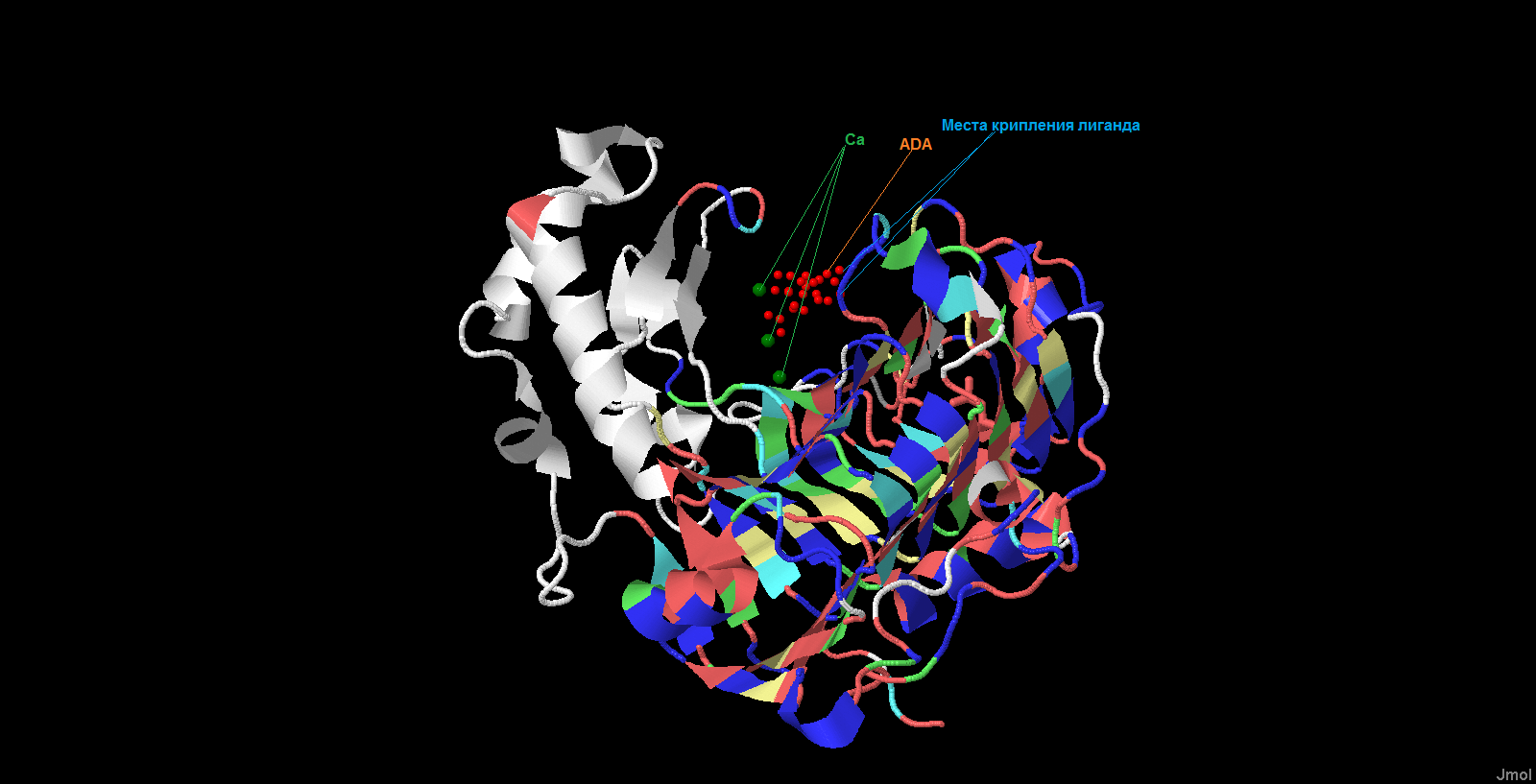
Рис. 4 Трехмерная структура белка PLY_BACSU, окрашенная в соответствии с консервативностью аминокислотных остатков
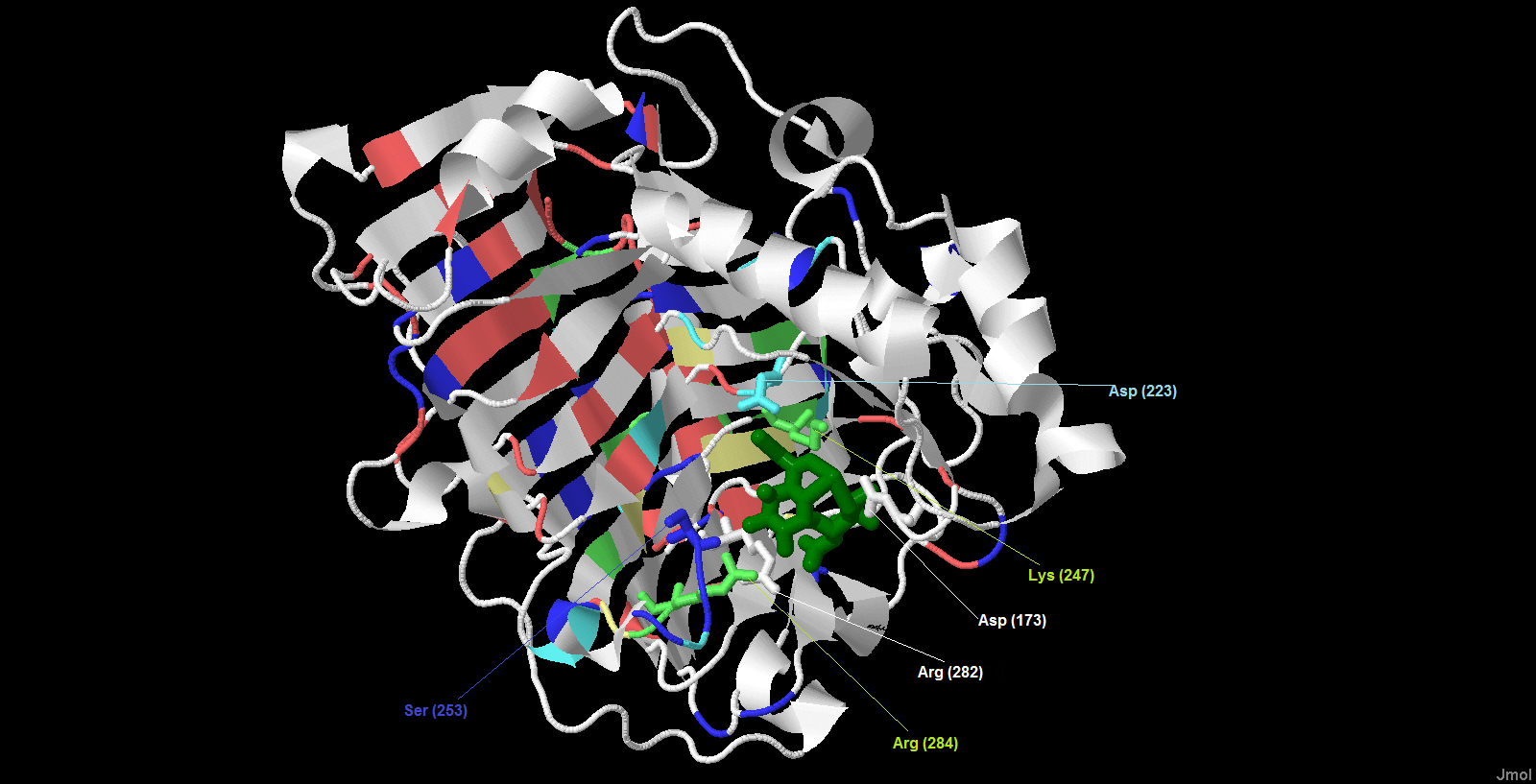
Рис. 5