Постороение дерева по нуклеотидным последовательностям
В данном задании были получены координаты генов 16S рРНК в геномах изучаемых бактерий. Полученные результаты были приведены в таблице ниже.
| Название | Мнемоника | AC записи EMBL | Координаты РНК | Цепь |
| Bacillus subtilis | BACSU | AL009126 | 30279..31832 | Прямая |
| Clostridium tetani | CLOTE | AE015927 | 176113..177621 | Прямая |
| Enterococcus faecalis | ENTFA | AE016830 | 1018187..1019708 | Прямая |
| Geobacillus kaustophilus | GEOKA | BA000043 | 30790..32343 | Прямая |
| Lactobacillus delbrueckii | LACDA | CR954253 | 45160..46720 | Прямая |
| Listeria monocytogenes | LISMO | AL591980 | 96266..97811 | Обратная |
| Staphylococcus epidermidis | STAES | AE015929 | 1598006..1599559 | Обратная |
Создадим общий fasta-файл с последовательностями, в котором отредактируем название последовательностей оставив только мнемонику.
При помощи програмы Mega построим дерево методом Maximum Likelihood.
Ниже представлено правильное дерево
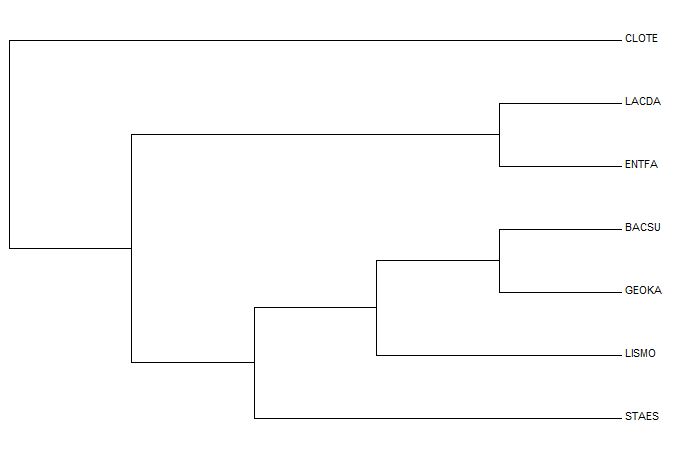
На дереве, построенном по нуклеотидным последовательностям появились такие ветви как:
А на правильном дереве в отличии от построенного присутствуют такие ветви как:
Как видно из изображений, представленных выше, деревья достаточно сильно различаются. В одном из предыдущих практикумов мы строили деревья по аминокислотным последовательностям 5 различными способами, и сравнив эти построения мы можем сказать, что дерево построенное по аминокислотным последовательностям является более точным. С одной стороны это могло произойти потому, что аминокислот у нас 20, а нуклеотидов всего 4. Но это компенсирутся тем, что длина нуклеотидной последовательности больше, чем аминокислотной. Поэтому причина различий не совсем ясна.
Постороение и анализ дерева, содержащего паралоги
В выбранных мною бактериях я нашла гомологи белка CLPX_BACSU. Для нахождения гомологов воспользовалась файлом proteo.fasta, который находится на диске Р. При помощи программы blastp был произведен поиск и отбор последовательностей по мнемонике, которые относятся к моим бактериям.
Далее был получен файл, содержащий находки, относящиеся к моим бактериям. Данный файл был импортирован в Mega и выровнен. Далее было постороено дерево представленное на рисунке ниже.

Если белки находятся в разных организмах и разделение их общего предка на линии, ведущие к ним, произошло в результате видообразования, то такие белки называются ортологами. А два гомологичных белка, находящиеся в одном организме, будут называться паралогами.
Ортологи: CLPX BACSU и CLPX GEOKA, HSLU ENTFA и HSLU LACDA
Паралоги: Q8YAB6 LISMO и Q8Y8B1 LISMO, CLPC BACSU и CLPE BACSU