Функциональная роль гена в подсистеме.
Мне был дан белок D2TIS8, который кодируется геном rcpA (ROD_41401). Белок пренадлежит бактерии Citrobacter rodentium (strain ICC168) (Citrobacter freundii biotype 4280). Предположиткльно, белок RcpA (Putative tight adherence secretin RcpA) нужен для образования плотных взаимодействий во время секреции бактерий с типом секреторной системы The Tad. Для экспорта Flp пилей (в данной системе) необходимо плотное взаимодействие. Предполагается, что RcpA образует наружний мультимерный секреторный мембранный канал (секретин) состоящий из Flp пилусного биогеного аппарата . Три Rcp белка (RcpA, RcpB, и RCPC) и TadD, предполагаемый липопротеин, локализуются в наружней мембране бактерий. RcpA, RcpC, и TadD также были обнаружены и во внутренней мембране.
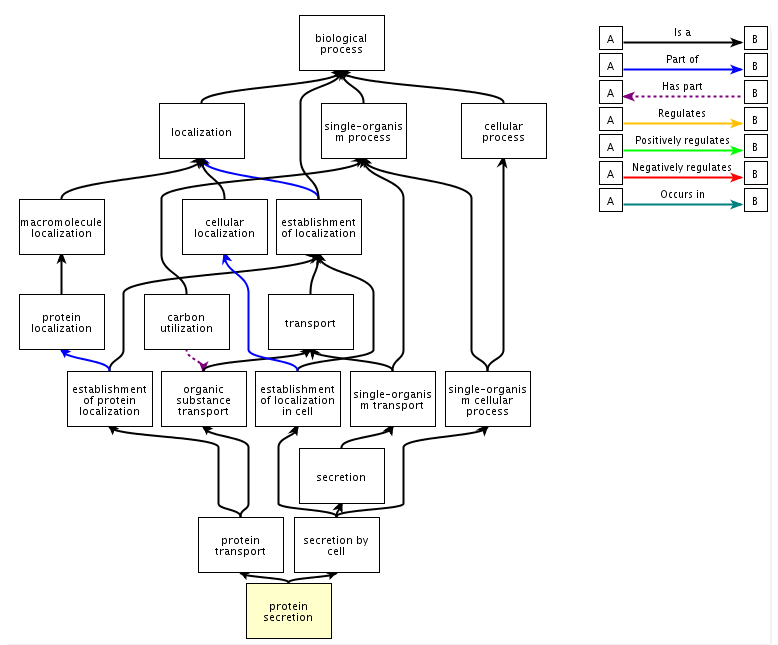
Был найден идентификатор GO. На изображении ниже показан результат поиска.

Так как функция белка предположительна, я решила посмотреть функции его гомологов. Гомологи были найдены по базе данных GO. В таблице ниже представленны несколько из возможных гомологов.
| GO ID | Название | Онтология | Определение |
| GO:0045166 | секреторный белок | биологический процесс | Контролирует выход белков из клетки |
| GO:0001593 | допамин нейротрансмостерная рецепторая активность посредствам соединения через Gi/Go | молекулярная функция | Объединение с нейромедиатором дофамином и активация аденилатциклазы, через соединения с Gi / Go. Это инициирует изменения клеточной активности. |
Информация о белке в базе данных KEGG . Для этого белка нет полного и точного метаболического пути. В представленной таблице(как раз по этому) отсутствует графа Pathway. Это объясняется тем, что все функции, приписанные к этому белку, являются лишь предположительными.
С помощью SEED Blast с параметрами Number of regions = 10, E-value cut off = 1e-25, Region Size (bp)=30000 (т.е. +-15000 оснований от гена) были найдены и отобраны 10 ортологов. Самого вида бактерии в списке не было, поэтому была выбрана бактерия из того же рода, но другого вида - Citrobacter koseri ATCC BAA-895. На изображении ниже представлена карта окрестностей отобранных ортологов. Полученное выравнивания приведено здесь.
Что мы можем видеть на представленном выше изображении: сиреневым цветом(цифра 14) обозначен белок General secretion pathway protein A. Предположительно, это тот самый белок, о котором шла речь ранее. Почему не нашелся наш белок? Мы смотрим у бактерии другого вида, и поэтому есть вероятность, что этот белок в одном штамме присутствует, а в другом, он представлен чем-то похожим. Можно посмотреть гомологов и сравнить. Может найдется этот белок. Стоит не забывать, что функция белка предположительная, так что можно сказать, что что-то похожее выполняют другие белки(General secretion pathway protein D(отмеченный красным цветом с цифрой 1)). Вероятно, что именно его сервис считает тем самым белком по которому велся поиск.
По ссылке Tabular Region information была получена таблица всех генов окрестностей. Далее была построенна сводная таблица. Ниже приведены результаты сводной таблицы.
| Встречаемость ( /11) | Set | |
| General secretion pathway protein D | 11 | 1 |
| General secretion pathway protein E | 11 | 2 |
| General secretion pathway protein F | 10 | 3 |
| General secretion pathway protein G | 10 | 4 |
| General secretion pathway protein K | 10 | 5 |
| General secretion pathway protein J | 8 | 6 |
| General secretion pathway protein L | 9 | 7 |
| General secretion pathway protein H | 7 | 8 |
| General secretion pathway protein C | 6 | 9 |
| General secretion pathway protein A | 3 | 14 |
Вывод: Как видно из рисунка, представленного выше, все генны из подсистемы колокализованы. Вероятно, колокализация происходит из-за того,что необходимо одновременно траскрибировать и транслировать гены системы секреции. Все это, по-видимому, так же способствует быстрой и скоординированной сборке секреторного комплекса.