Задание №0
В базе данных OPM были найдены три трансмембранных альфа-спиральных белка и три трансмембранных бета-баррельных белка. результаты поиска представленны в таблице ниже.
Таблица 1. Параметры для трансмембранных белков
| PDB код | Тип (спираль, баррель) | Какая мембрана | Толщина гидрофобной части мембраны в ангстремах | Медиана числа остатков в одном трансмембранном участке |
| 4J7C | спираль | Плазматическая мембрана, Bacillus subtilis | 31.8 ± 1 А | 24 |
| 2YEV | спираль | Внутренняя мембрана, Thermus thermophilus | 31.2 +- 0.8 А | 24 |
| 1L9B | спираль | Внутренняя мембрана, Rhodobacter sphaeroides | 31.6 ± 0.8 А | 24 |
| 3PIK | баррель | Внешняя мембрана, Escherichia coli | 24.3 ± 1.2 А | 10 |
| 4E1T | баррель | Внешняя мембрана, Yersinia pseudotuberculosis | 25.6 ± 1.4А | 9 |
| 3B07 | баррель | Клеточная мембрана, Pseudomonas fluorescens | 24.2 ± 1.2 А | 9 |
Из таблицы видно, среднее число остатков для трансмембранных альфа-спиральных белков равно 24, а для бета-баррелей - около 9 остатков. Средняя толщина мембраны для альфа-спиральных белков равна 31 А, а для бета-баррелей примерно 24А. По полученным данным можно сказать, что количество остатков увеличивается с увеличением мембраны.
Oтбор гомологов
При помощи BLAST была создана репрезентативная выборка гомологов выданного белка KtrAB. Так как в белке много цепей, была выбрана одна цепь (I), по которой производилась выборка. Бактерия (Bacillus subtilis), из которой взят данный белок, относится к филуму firmicutes. Поэтому при поике были исключены данный филум и эукариоты, порог e-value был установлен равный 1 и количество последовательностей, выдаваемых при поиске 1000. Поиск производился пр базе данных RefSeq.
Aнализ структуры выданного белка
В ходе работы была получена информация, которая находится в таблице 2.
Таблица 2. Описание структуры трансмембранного транспортера калия KtrAB (идентификатор PDB 4J7C, цепь I).
| PDB ID | Организм | Тип мембраны | TC-код | Угол наклона спиралей (бета-тяжей) к нормали | Количество трансмембранных спиралей (бета-тяжей в бочонке) |
| 4J7C | Bacillus subtilis | Плазматическая мембрана | 2.A.38 | 0 | 17 |
Расшифровка ТС-кода:
2. - потенциало-зависимые транспортеры
2.А. - портеры
2.А.38 - семейство калиевых транспортеров (Trk)
Aнализ множественного выравнивания трансмембранных белков
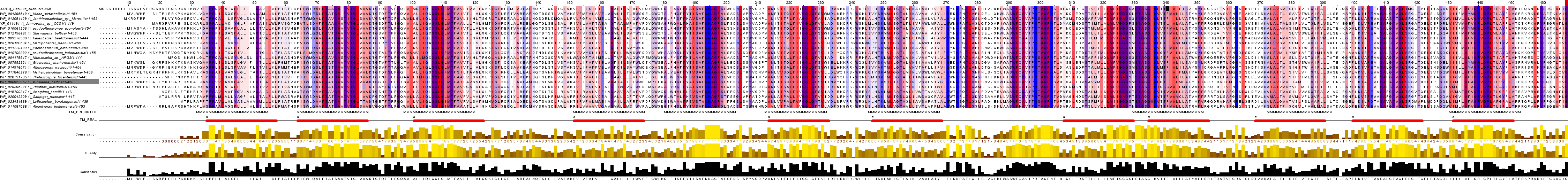
Построила множественное выравнивание отобранных гомологов с помощью программы Muscle. При помощи программы JalView было построенно выравнивание выбранных гомологов и данного белка. В новой строке с названием "TM_REAL" были помечены участки выравнивания, отвечающие трансмембранным спиралям в данном мне белке. В строке "TM_PREDICTED" отмечены альфа-спирали, выдаваемые программой TMHMM, принадлежащие гомологу WP_004395519.1 из Vibrio_metschnikovii. Выбрала цветовую схему, позволяющую визуально различать гидрофобные и гидрофильные остатки. Затем установила галочку на "By Conservation" (30%). Предсказание программы TMHMM представлены на рисунке 1. Полученное выравнивание показано на рисунке 2.
Рисунок 1.Предсказание из ТМНММ для гомолога WP_004395519.1
Рисунок 2.Выравнивание репрезентативной выборки. Цветовая схема по гидрофобности, порог консервативности 30%.
Часть участков, встречающихся в альфа-спиралях, консервативны, а чать - нет. У выбранного гомолога есть спирали, которые частично совпадают, а есть те которые не совпадают совсем. Из полученных результатов видно, что спирали белков чаще всего расположены в консервативной области, но бывают и исключения. В консервативных областях последовательности чаще всего присутствубт такие аминокислоты как : серин, аланин, лейцин(изолейцин), валин, пролин, трионин, лизин, глутаминовая кислота. Реже аспарогиновая кислота, аспарагин, аргинин.