Обзор
Мини-обзор генома Vibrio Cholerae
Малахов Георгий
1 Факультет биоинженерии и биоинформатики МГУ им. М.В. Ломоносова
Абстракт: В данном исследовании проведен некоторый анализ геномных молекул ДНК Vibrio cholerae, а именно анализ их нуклеотидного состава, анализ белок-кодирующих генов, РНК кодирующих генов, частоты встречаемости различных стоп-кодонов в геноме, GC-skew анализ каждой из геномных молекул ДНК. На основании некоторых результатов выдвинуты предположения.
Ключевые слова: Vibrio cholerae, GC-skew, анализ генома.
1. Введение
Vibrio cholerae — грамотрицательная факультативно анаэробная бактерия [1]. Геном V. cholera, представленный двумя кольцевыми хромосомами а также одной плазмидой, представляет интерес из-за патогенности бактерии: она вызывает холеру — заболевание ЖКТ, причем в последнее время бактерия стала резистентна к широкому спектру антибиотиков [2-4]. Бактерии V. cholera способны создавать биопленки, используя чувство кворума, что также усиливает патогенность и заразность бактерии [5-6]. Также на второй хромосоме V. cholerae лежит достаточно крупный (126 Kb) высоковариабельный генетический элемент — суперинтегрон, это позволяет бактерии быстро адаптироваться к изменяющимся условиям среды, в частности приобретать устойчивость к антибиотикам [2]. Это, как и разделение генома на две хромосомы, обеспечивает высокую скорость эволюции генома V. cholerae [2].
В этом исследовании я проанализировал некоторые данные о геноме Vibrio cholerae, и на их основе выдвинул некоторые предположения.
2. Материалы и методы
Для исследования генома V. cholerae были использованы написанные мною на Python 3.9 программы, GoogleTabs и сервис Webskew с открытым исходным кодом. Геном бактерии и информация о нем взята с сайта NCBI.
3. Результаты
3.1 Описание стандартных данных о геноме Vibrio Cholerae
Из данных о сборке генома можно понять, что как было сказано выше, геном V.cholerae состоит из двух крупных кольцевых хромосом и одной плазмиды, данные о длине каждой из молекул ДНК представлены в данных о сборке генома, но точность определения GC-состава невелика ,так что стоит уточнить имеющиеся данные, применив вычислительные методы к геномной последовательности. Для этого написана программа, результаты работы которой представлены в таблице 1.
| Молекула ДНК | Длина, п. н. | GC-состав, % |
|---|---|---|
| Хромосома 1 | 2948589 | 47.72 |
| Хромосома 2 | 1140710 | 46.92 |
| Плазмида | 49113 | 41.23 |
По GC-составу понятно, что бактерия живет в относительно умеренных температурных условиях. Две крупные хромосомы выгодно увеличивают скорость эволюции генома V. cholerae [2].
3.2 Статистические данные о белках протеома Vibrio cholerae
Исследование распределения белков по длинам было проведено на основе таблицы особенностей генома. Как видно из рисунка 1, для этого распределения наблюдаются характерные два пика в районе 150 и 300 аминокислотных остатков.
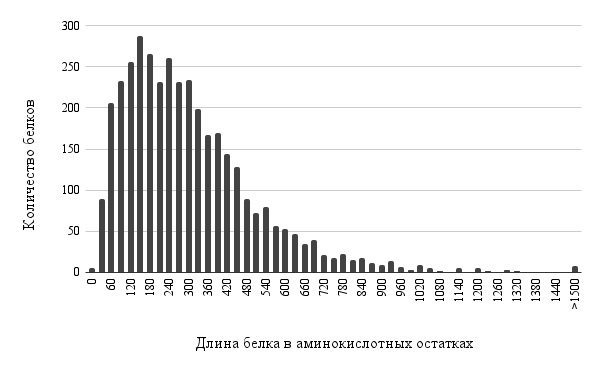
Могу предположить, что пик в районе 150 аминокислотных остатков соответствуют большинству однодоменных белков, пик в районе 300 аминокислотных остатков - большинству двухдоменных белков. Также можно заметить небольшие пики каждые примерно 120 - 150 аминокислотных остатков.
Проанализировав таблицу особенностей генома, я пришел к результатам представленным в таблице 2.
| Тип белков | Количество белков | Доля белков, % |
|---|---|---|
| Рибосомальные белки | 60 | 1.65 |
| Гипотетические белки | 317 | 8.71 |
| Транспортные белки | 367 | 10.09 |
| Все белки | 3639 | 100.00 |
Как видно из таблицы, всего 8.71% белок-кодирующих последовательностей - гипотетические. Причем из таблицы особенностей ясно, что каждая из этих последовательностей вероятно кодирует белки меньше 60 аминокислотных остатков. Это показывает, что протеом V. cholerae достаточно хорошо изучен.
Также получены данные о количествах генов белков на прямой и комплементарной цепях ДНК каждой из молекул, составляющих геном бактерии и рассчитаны p-value распределения генов белков по цепям ДНК. Результаты работы программы представленные в таблице 3.
| Молекула ДНК | Цепь | Количество генов | p-value распределения |
|---|---|---|---|
| Хромосома 1 | Прямая | 1305 | 0.13 |
| Обратная | 1228 | ||
| Хромосома 2 | Прямая | 566 | 6.41⋅10-3 |
| Обратная | 477 | ||
| Плазмида | Прямая | 57 | 1.64⋅10 -11 |
| Обратная | 6 |
Мы не можем отвергнуть утверждение о том, что на первой хромосоме гены распределены по цепям случайно, так как p-value > 0.05. Но вот для второй хромосомы уже есть отклонение от случайного распределения, для плазмиды отклонение от случайного распределения еще больше.
3.3 Статистические данные о генах РНК Vibrio cholerae
Проанализировав программой таблицу особенностей генома, я получил данные о генах РНК V.cholerae, представленные в таблице 4.
| Гены РНК | Количество генов |
|---|---|
| Все гены РНК | 137 |
| Гены тРНК | 102 |
| Гены рРНК | 31 |
Остальные четыре гена, приходятся на 6S РНК, SRP (частица распознавания сигнала от англ. signal recognition particle), Рибонуклеазу Р и транспортно-матричную РНК.
Мне стало интересно почему генов тРНК больше, чем вариантов антикодонов, я проанализировал таблицу особенностей генома и выписал антикодоны всех тРНК, закодированных в геноме V. cholerae, а также частоты их встречаемости в таблицу 5.
| Аминокислота | Антикодон | Количество встреч |
|---|---|---|
| Аланин | CGC | 1 |
| TGC | 5 | |
| Аргинин | ACG | 6 |
| GCG | 1 | |
| Аспарагин | GTT | 4 |
| Аспарагиновая кислота | GTC | 5 |
| Цистеин | GCA | 3 |
| Глутамин | TTG | 5 |
| Глутаминовая кислота | TCC | 5 |
| Глицин | GCC | 7 |
| TCC | 2 | |
| Гистидин | GTG | 2 |
| Изолейцин | GAT | 5 |
| Лейцин | CAA | 1 |
| TAG | 5 | |
| CAG | 4 | |
| TAA | 2 | |
| Лизин | TTT | 3 |
| Метионин | CAT | 7 |
| Фенилаланин | GAA | 3 |
| Пролин | TGG | 3 |
| GGG | 1 | |
| Серин | TGA | 2 |
| GCT | 2 | |
| GGA | 1 | |
| Треонин | GGT | 2 |
| TGT | 4 | |
| Триптофан | CCA | 1 |
| Тирозин | GTA | 4 |
| Валин | TAC | 3 |
| GAC | 2 |
Первое, что я заметил - то, что антикодонов в геноме V. cholerae всего 30 из 64 возможных сочетаний. Очевидно, в кодон-антикодоновом спаривании при трансляции у данной бактерии, часто имеет место wobble-взаимодействие. Также я обратил внимание на то, что среди антикодонов различных аминокислот, например, аланина и аргинина, существуют некие “минорные ” антикодоны. Из таблицы особенностей генома видно, что все тРНК для одной аминокислоты находятся крайне близко друг к другу, предположу, что даже транскрибируются эти тРНК вместе. Вероятно, это механизм тонкой регуляции трансляции, причем как возможности, так и скорости ее протекания для отдельных белков. На мой взгляд, это может быть полезно при трансляции большого белка, чтобы успевал происходить “здоровый” фолдинг новосинтезированной полипептидной цепи.
3.4 Нуклеотидный состав геномных ДНК
Программа, использованная для подсчета ГЦ-состава была модифицирована для оценки нуклеотидного состава геномных ДНК. Для оценки верности второго правила Чаргаффа найдем количества каждого нуклеотида в каждой паре в своей цепочке а затем рассчитаем p-value, считая данное распределение биномиальным, а второе правило Чаргаффа - нулевой гипотезой. Результаты представлены в таблице 6.
| Молекула ДНК | Нуклеотид | Доля нуклеотидов в цепочке, % | p-value биномиального распределения в паре |
|---|---|---|---|
| Хромосома 1 | A | 26.25 | 3.53⋅10-7 |
| C | 23.81 | 1.78⋅10-2 | |
| G | 23.91 | 1.78⋅10-2 | |
| T | 26.03 | 3.53⋅10-7 | |
| Хромосома 2 | A | 26.46 | 1.55⋅10-2 |
| C | 23.48 | 0.59 | |
| G | 23.44 | 0.59 | |
| T | 26.62 | 1.55⋅10-2 | |
| Плазмида | A | 28.52 | 5.47⋅10-7 |
| C | 17.77 | 5.47⋅10-86 | |
| G | 23.46 | 5.47⋅10-86 | |
| T | 30.25 | 5.47⋅10-7 |
По таким данным отвергнуть второе правило Чаргаффа нельзя только для распределения G и С во второй хромосоме. Тут, как и в исследовании протеома из общей картины сильно выбивается плазмида с p-value распределения G и С равным 5.47 ⋅ 10-86.
3.5 Поиск точек начала репликации и терминации репликации геномных молекул
Используя сервис Webskew, я получил графики GC-skew и кумулятивного GC-skew представленные на рисунке 2.
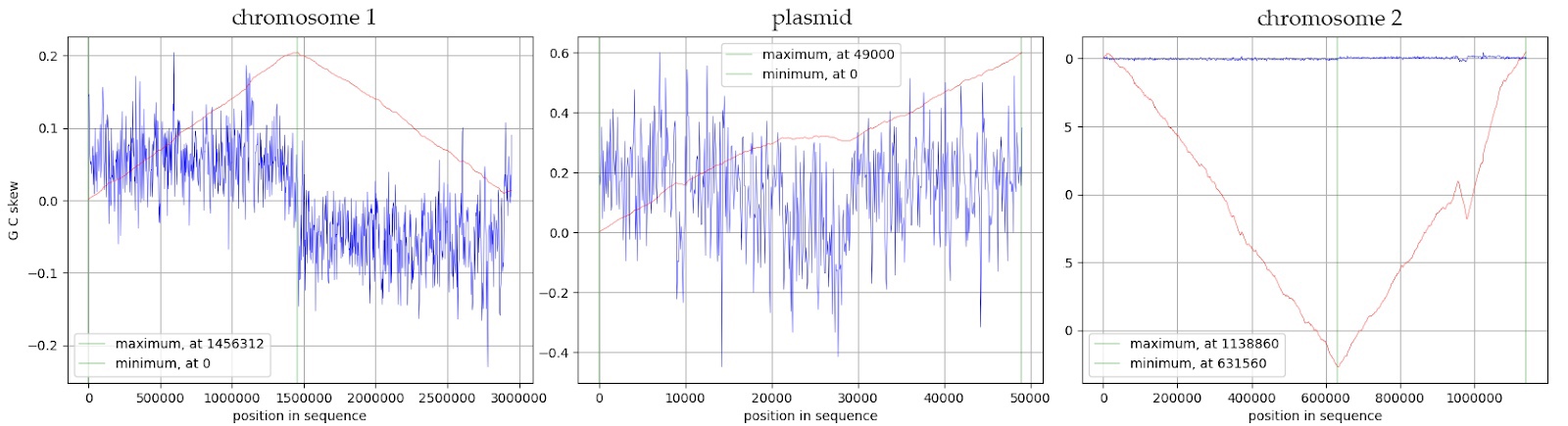
Очевидно различие между картинами графиков кумулятивного GC-skew для хромосом и плазмиды. Графики кумулятивного GC-skew хромосом V образны, и имеют небольшой излом на одном из концов (график для первой хромосомы имеет излом на правом конце, график для второй хромосомы - на левом). Очевидно, что точка ter в первой хромосоме располагается примерно на 1456312 нуклеотиде, точка oriC - в окрестности нулевого нуклеотида (излом на правом конце графика). Точка ter на второй хромосоме располагается в окрестности нулевого нуклеотида (излом на левом конце графика), точка oriC - примерно на 1138860 нуклеотиде. Интересен также излом на правом плече графика кумулятивного GC-skew для второй хромосомы. Из формулы GC-skew понятно, что на этом участке, наблюдается сильно больше С, чем G. Могу предположить, что это связано с наличием на данном участке суперинтегрона - высоковариабельного генетического элемента.
Для плазмиды картина совершенно другая, могу предположить, что это происходит из-за другого механизма репликации плазмиды (принцип катящегося колеса), наиболее перспективным участком для анализа, мне кажется интервал, где график выходит на плато с локальным максимумом (около 21000 нуклеотида), а затем минимумом (около 28000 нуклеотида), вероятно, это и есть точки ter и oriC соответственно.
3.6 Частота встречаемости стоп-кодонов
Написав программу, подсчитывающую количество встреч каждого из стоп кодонов кодирующих белок последовательностей, я получил данные, представленные в таблице 7.
| Молекула ДНК | Стоп-кодон | Количество встреч |
|---|---|---|
| Хромосома 1 | TAA | 1642 |
| TAG | 416 | |
| TGA | 475 | |
| Хромосома 1 | TAA | 661 |
| TAG | 189 | |
| TGA | 193 | |
| Плазмида | TAA | 37 |
| TAG | 13 | |
| TGA | 13 |
Действительно, TAG узнается фактором терминации трансляции RF1, TGA - фактором терминации трансляции RF2, а TAA узнается обоими [7]. Причем, логично, что мутации в стоп-кодонах чаще приводят к заменам на TAA, чем на TAG или TGA. Я думаю, именно поэтому, TAA лидирует по частоте встречаемости в белок-кодирующих генах.
4. Сопроводительные материалы
Литература
- Van Alst, Andrew J., и Victor J. DiRita. «Aerobic Metabolism in Vibrio Cholerae Is Required for Population Expansion during Infection». MBio 11, вып. 5 (1 сентябрь 2020 г.): e01989-20. https://doi.org/10.1128/mBio.01989-20 .
- Escudero, Jose Antonio, и Didier Mazel. «Genomic Plasticity of Vibrio Cholerae». International Microbiology: The Official Journal of the Spanish Society for Microbiology 20, вып. 3 (сентябрь 2017 г.): 138–48. https://doi.org/10.2436/20.1501.01.295 .
- Harris, Jason B., Regina C. LaRocque, Firdausi Qadri, Edward T. Ryan, и Stephen B. Calderwood. «Cholera». Lancet (London, England) 379, вып. 9835 (30 июнь 2012 г.): 2466–76. https://doi.org/10.1016/S0140-6736(12)60436-X .
- Zhou, Yifan, Shuwen Gu, Jie Li, Peng Ji, Yingjie Zhang, Congcong Wu, Qun Jiang, Xiaojian Gao, и Xiaojun Zhang. «Complete Genome Analysis of Highly Pathogenic Non-O1/O139 Vibrio Cholerae Isolated From Macrobrachium Rosenbergii Reveals Pathogenicity and Antibiotic Resistance-Related Genes». Frontiers in Veterinary Science 9 (2022 г.): 882885. https://doi.org/10.3389/fvets.2022.882885 .
- Kelly, Robert C., Megan E. Bolitho, Douglas A. Higgins, Wenyun Lu, Wai-Leung Ng, Philip D. Jeffrey, Joshua D. Rabinowitz, Martin F. Semmelhack, Frederick M. Hughson, и Bonnie L. Bassler. «The Vibrio Cholerae Quorum-Sensing Autoinducer CAI-1: Analysis of the Biosynthetic Enzyme CqsA». Nature Chemical Biology 5, вып. 12 (декабрь 2009 г.): 891–95. https://doi.org/10.1038/nchembio.237 .
- Silva, Anisia J., и Jorge A. Benitez. «Vibrio Cholerae Biofilms and Cholera Pathogenesis». PLoS Neglected Tropical Diseases 10, вып. 2 (февраль 2016 г.): e0004330. https://doi.org/10.1371/journal.pntd.0004330 .
- Ho, Alexander T, и Laurence D Hurst. «Variation in Release Factor Abundance Is Not Needed to Explain Trends in Bacterial Stop Codon Usage». Molecular Biology and Evolution 39, вып. 1 (1 январь 2022 г.): msab326. https://doi.org/10.1093/molbev/msab326 .
