Сравнение протеомов M. tuberculosis и M. bovis
Выбор протеомов для сравнения
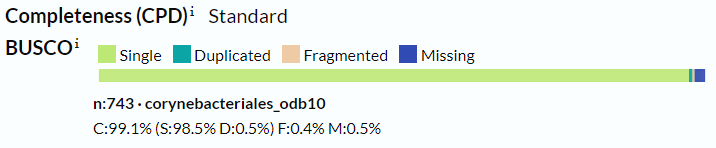
В прошлом практикуме я обозревал белок Mycobacterium tuberculosis, поэтому один из протемов для сравнения был определен заранее. Это протеом с ID UP000001584. Значение его Completness - Standard, а значит протеом по размеру близок к среднему по таксономической группе. BUSCO показывает, что в протеоме 99.1% покрытия ортологичных генов. 98.5% составляет значение Single. Мне кажется, что исследуемый протеом хорошего качества.
Вторым для сравнения я выбрал протеом Mycobacterium bovis. Эта бактерия - близкий родственник Mycobacterium tuberculosis, она также вызывает болезни дыхательной системы, но, обычно, у крупного рогатого скота. Болезнь может передаваться человеку или другим млекопитающим через неочищенное молоко и мясо, а также иногда и воздушно-капельным путем. ID этого протеома UP000001419 Значение CPD протеома - Close to standard (low value), а значит у протеома размер чуть меньше среднего по группе. BUSCO показывает, что протеом покрывает выбранные ортологичные гены на 99.3%, 98.8% соответствует значению Single.

Сравнение протеомов
Для сравнения протеомов я скачал их на kodomo и написал скрипт в Python. Ссылка на скрипт. Посчитать размер протеомов несложно, кроме того он представлен в UniProt. Размер протеома Mycobacterium tuberculosis - 3995 белковых записей, у Mycobacterium bovis размер чуть больше - 4088.
Дальше нужно было сравнить доли трансмембранных белков в протеомах. Для определения, трансмембранный ли белок, я использовал feature table (FT) из аннотации белка. Если в аннотации есть строчка, которая начинается на FT TRANSMEM, то я считал белок трансмембранным. Я считаю этот метод оптимальным, так как он не зависит от качества аннотаций белков, потому что feature table генерируется автоматически, так что даже у белковых записей из базы данных TrEMBL есть вразумительная feature table. Запустив программу, получаем доли трансмембранных белков в каждом из протеомов: 18.15% для Mycobacterium tuberculosis и 17.61% для Mycobacterium bovis. Значения близки, так как организмы близкородственны.
Затем, надо было сравнить доли ферментов в протеомах. Самый очевидный поиск ферментов на основе наличия каталитической активности меня совершенно не устроил, так как каталитическая активность есть только у хорошо аннотированных белков. Очевидно, нужно использовать какие-то характеристики, которые есть не только у хорошо аннотировных записей, иначе многие записи ускользнут от анализа. Постепенно я пришел к определению ферментов в три шага. Первый шаг - поиск в названии суперсемейства (оно определяется автоматически, так что есть не только у хорошо аннотированных белков) суффикса "аза", то есть поиск регулярного выражения r'^DR {3}SUPFAM.+ase'. Второй шаг - поиск среди биологических активностей символов "ase activity" (азная активность). Ну и последний третий шаг - поиск в названии фермента суффикса "ase" (aза). Причем поиск должен осуществвляться именно в конце названия, так как есть, например, некоторые белки, связывающие ферменты, тогда их название будет оканчиваться на "ase binding protein". Для такого поиска я использовал регулярное выражение r'^DE {3}RecName:.+ase \{'. Собственно, если хотя бы один из трех шагов успешно что-то обнаружил, я считал белок ферментом. Получились доли ферментов: 54.44% для Mycobacterium tuberculosis и 52.69% для Mycobacterium bovis. Цифры опять получились схожие, ничего неожиданного не произошло.
Я решил сравнить количества факторов вирулентности. К сожалению, я не придумал, как получить информацию о вирулентности из автоматически составляемой части аннотации, но выбранные протеомы вполне неплохо аннотированы (2288 из 3995 записей из Swiss-Prot для Mycobacterium tuberculosis и 1244 из 4088 для Mycobacterium bovis). Для этого я искал в аннотации регулярное выражение r'^KW.+Virulence'. Запустив программу, получил 154 фактора вирулентности для Mycobacterium tuberculosis и 18 для Mycobacterium bovis. Можно, конечно, предположить, что разница обусловлена более богатой аннотированностью протеома Mycobacterium tuberculosis, но различие между количествами аннотированных белков примерно в два раза, а количества факторов вирулентности различаются много сильнее. Вероятно активная борьба человечества с туберкулезом привела к увеличению количества механизмов защиты. Еще могу предположить, что различия вызваны различиями в иммунных системах хозяев той и иной бактерии-паразита, но мне, к сожалению, не хватает знаний для уверенных выводов.
В последнюю очередь я решил сравнить количество белков в протеомах, ответственных за резистентность к антибиотикам. У такого анализа есть та же проблема, что и у прошлого: поиск возможен только по аннотированным записям. Для сравнения я использовал реуглярное выражение r'^KW.+Antibiotic resistance'. Были получены количества: 49 для Mycobacterium tuberculosis и 13 для Mycobacterium bovis. Думаю, различие обусловлено тем, что для лечения человека используются более разнообразные антибиотики и в большем количестве, чем для лечения скота.
