Сборка хромосом de novo
Задание 1
Для тримминга адаптеров я объединил файлы с последовательностми адаптеров в один с помощью cat. Дальше я оттриммил скачанную библиотеку ридов с идентификатором SRR4240380 командой
TrimmomaticSE SRR4240380.fastq.gz output.fq.gz ILLUMINACLIP:adapters.fasta:2:7:7Триммер выдал вывод, откуда понятно, что 1.88% ридов действительно содержали адаптеры. Финальный тримминг был произведен командой
TrimmomaticSE SRR4240380.fastq.gz output.fq.gz ILLUMINACLIP:adapters.fasta:2:7:7 TRAILING:20 MINLEN:32На этот раз из исходного файла было удалено 6.75% ридов, размер fastq файла после тримминга уменьшился со 108 мегабайт до 99.
Задание 2
С помощью velveth я k-меризовал риды командой
velveth Assem 31 -short -fastq.gz output.fq.gzЗадание 3
Затем я собрал геном с помощью velvetg командой
velvetg ./Assem/Покрытие большинства контигов около двадцати, но у некоторых покрытие совсем небольшое - 2 - 3. Вот три из таких непокрытых контигов:
>NODE_235_length_62_cov_2.419355
CTATGGTAGATTAATATAATCAATTAATACAAAATCCTAACCTAACCTAACCGTACTAAA
ATCAGATGAATAAACGCAAACGCATACAAAAC
>NODE_301_length_63_cov_2.888889
TTCGCACGCGGTTCACGCGGTGTACCAGGTAAAGCCGGCGATCTCACTCATTTATAGTTT
GAGAATAGGTTGAGGTCGTTTCGGCCCCAATGT
>NODE_146_length_66_cov_3.045455
AGAACTAGGGCGGTATCTGATCGCCTTCGAACCTCTAACTTTCGTTCTTGATCATACGAG
AACGTACTTGGCAAATGCTTTCGCGTCAGTTCGTCTКонтиги довольно короткие, но не вижу в них ничего особенного. Наверное, их плохое покрытие можно списать на плохое качество секвенирования. N50 сборки - 12042.
Задание 4
Затем я разбил файл с контигами на много файлов формата fasta, и нашел три самых длинных контига длинами 25915, 23850 и 23807.
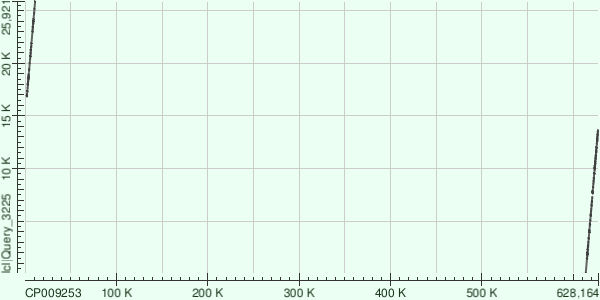
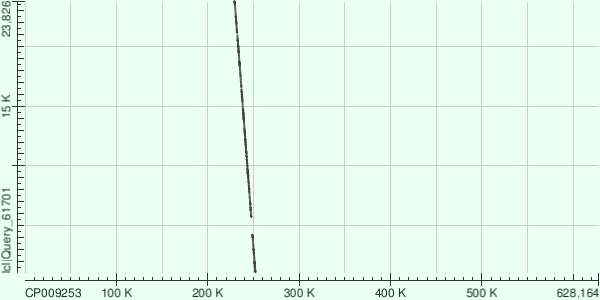
Как видно из графиков, контиги 3 и 20 (рисунки 1 и 2) очень неплохо отравнялись на геном бактерии, геном которой мы собирали. Единственное, в контиге 20 (рисунок 2) есть участок невыравненный на геном, предполагаю, что это связано либо с плохим качетством прочтения, либо это вариабельный участок генома. Контиг 23 (третий рисунок) откартировался хуже - есть небольшой участок несовпадения (примерно 10K по контигу) и очень большой (примерно 15K - примерно 20K по контигу),
Контиг 3 лег на координаты хроомосомы 613658 - 11103 (через 0 т.е. через начало секвенирования), контиг лежит на той же цепи по отношению к референсу.
Контиг 20 лег на координаты хроомосомы 229411 - 252164 с прерыванием 247596 - 248967, контиг лежит на обратной цепи по отношению к референсу.
Контиг 23 лег на координаты хроомосомы 573092 - 594099 с прерываниями 582686 - 584329, 587055 - 593743, контиг лежит на той же цепи по отношению к референсу.