Формы ДНК, структура РНК
Задание 1
С помощью программы fiber были получены структуры трех форм ДНК: Ссылка на форму А. Ссылка на форму B. Ссылка на форму Z.
Задание 2
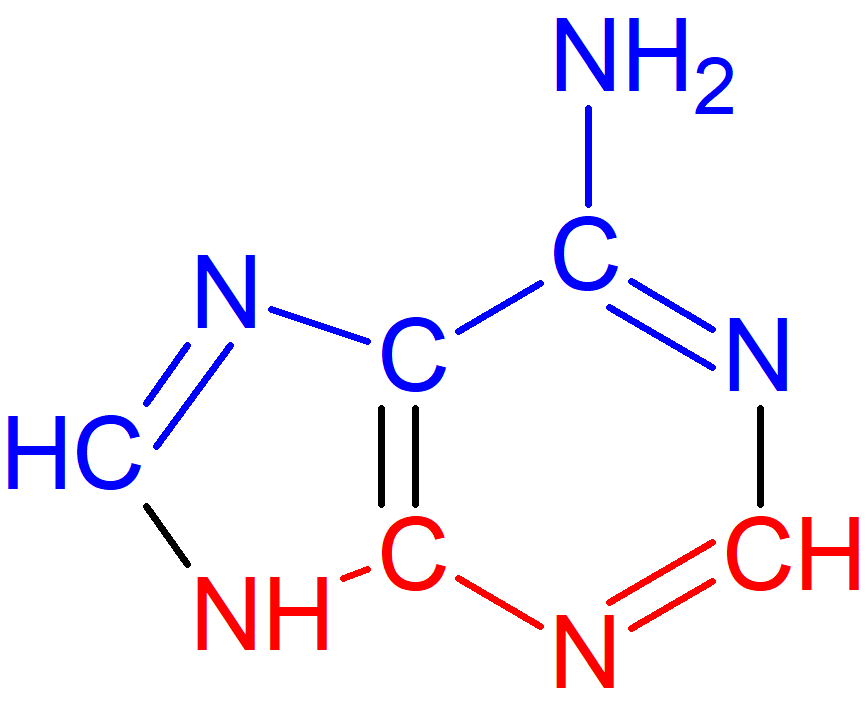
Для заданий мне был выдан аденин, из форм ДНК я выбрал форму А.
- В сторону большой бороздки обращены атомы C2, N3, C4, N9.
- В сторону большой бороздки обращены атомы N1, C5, N6, C6, N7, C8.
| Параметр | A-форма | B-форма | Z-форма |
|---|---|---|---|
| Тип спирали | правозакрученная | правозакрученная | левозакрученная |
| Шаг спирали (Å) | 28 | 33.8 | 43.5 |
| Число оснований на виток | 12 | 11 | 13 |
| Ширина большой бороздки (Å) | 16.8 (цепь А G9 - цепь B A34) |
17.2 (цепь А G9 - цепь B G39) |
18.3 (цепь А С2 - цепь B C30) |
| Ширина малой бороздки (Å) | 8 (цепь А G9 - цепь B A26) |
11.7 (цепь А G9 - цепь B C36) |
9.9 (цепь А C8 - цепь B G37) |
Задание 3.1
Программами find_pair и analyze были получены торсионные углы нуклеотидов в цепях ДНК и РНК. Торсионные углы РНК из результатов работы программы представлены в кодблоке.
Strand I
base alpha beta gamma delta epsilon zeta chi
1 G --- --- 174.2 89.3 -134.7 -66.8 175.2
2 G -71.9 179.9 53.2 81.6 -166.3 -63.7 -165.0
3 G -61.8 -177.5 53.8 81.1 -156.4 -69.1 -159.3
4 G 145.5 -164.3 -172.8 79.2 -139.8 -68.6 -174.8
5 U -60.2 178.6 47.8 81.0 175.8 -82.7 -158.0
6 A -172.6 -130.2 164.7 112.4 -69.3 -73.3 -157.0
7 C 19.4 -170.5 48.4 87.8 -144.1 -78.8 -166.8
8 G -49.6 169.2 46.1 84.8 -152.8 -63.9 -164.8
9 A -67.4 178.6 49.1 78.3 -175.7 -37.8 -149.7
10 G 178.4 148.9 160.4 84.0 -147.4 -68.6 -169.4
11 G -55.0 178.6 42.1 79.1 -155.7 -67.8 -168.1
12 U -63.9 -172.8 50.7 86.3 -140.0 -68.3 -165.1
13 U -59.3 170.3 49.0 83.1 -134.8 -81.5 -143.9
14 A -102.7 -158.0 54.1 81.8 -143.6 -62.4 -170.2
15 U -51.6 163.3 56.3 82.8 -128.5 -59.9 170.1
16 U -68.4 176.4 48.2 86.2 -153.3 -70.8 -172.3
17 C -62.4 163.9 54.9 84.7 -108.8 -147.6 174.1
18 C 105.8 -105.2 179.1 82.6 -129.9 -69.1 173.8
19 G -67.4 179.4 41.9 78.4 -156.9 -77.9 -177.4
20 G -56.7 176.5 50.9 70.3 83.5 54.7 -168.3
21 A 96.2 -176.4 162.9 92.2 -85.7 -135.7 -168.0
22 G 158.0 151.2 52.5 79.1 -155.8 -67.4 -173.9
23 C -65.4 -172.3 49.2 80.7 -158.5 -72.0 -154.3
24 C 148.2 -165.2 178.3 85.3 -129.8 -63.5 -170.6
25 A -53.1 161.1 56.7 82.2 -176.3 -82.7 -146.5
26 A 157.2 175.0 178.0 87.5 -138.1 -80.2 -172.6
27 G -39.0 159.7 46.3 87.4 -114.5 -31.3 -150.5
28 G -110.5 150.0 55.3 97.7 -160.4 178.6 -102.7
Далее я сравнил средние торсионные углы в каждой из форм ДНК с выданной мне РНК.
| Молекула | альфа | бета | гамма | дельта | эпсилон | дзета | хи |
|---|---|---|---|---|---|---|---|
| РНК | -12.2 | 47.0 | 68.8 | 84.4 | -120.9 | -59.7 | -124.1 |
| ДНК А-форма | -51.7 | 174.8 | 41.7 | 79.1 | -147.8 | -75.1 | -157.2 |
| ДНК B-форма | -29.9 | 136.3 | 31.1 | 143.3 | -140.8 | -160.5 | -98.0 |
| ДНК Z-форма | -43.8 | 21.1 | -61.5 | 116.2 | -100.0 | 8.6 | -47.8 |
Из таблицы можем сделать вывод, что тяжи выданной для задания РНК больше всего похожи на А-форму ДНК.
Задание 3.2
Strand I Strand II Helix
1 (0.010) ....>B:.902_:[..G]G-----C[..C]:.971_:B<.... (0.005) |
2 (0.012) ....>B:.903_:[..G]G-----C[..C]:.970_:B<.... (0.007) |
3 (0.011) ....>B:.904_:[..G]G-----C[..C]:.969_:B<.... (0.006) |
4 (0.012) ....>B:.905_:[..G]G-----C[..C]:.968_:B<.... (0.010) |
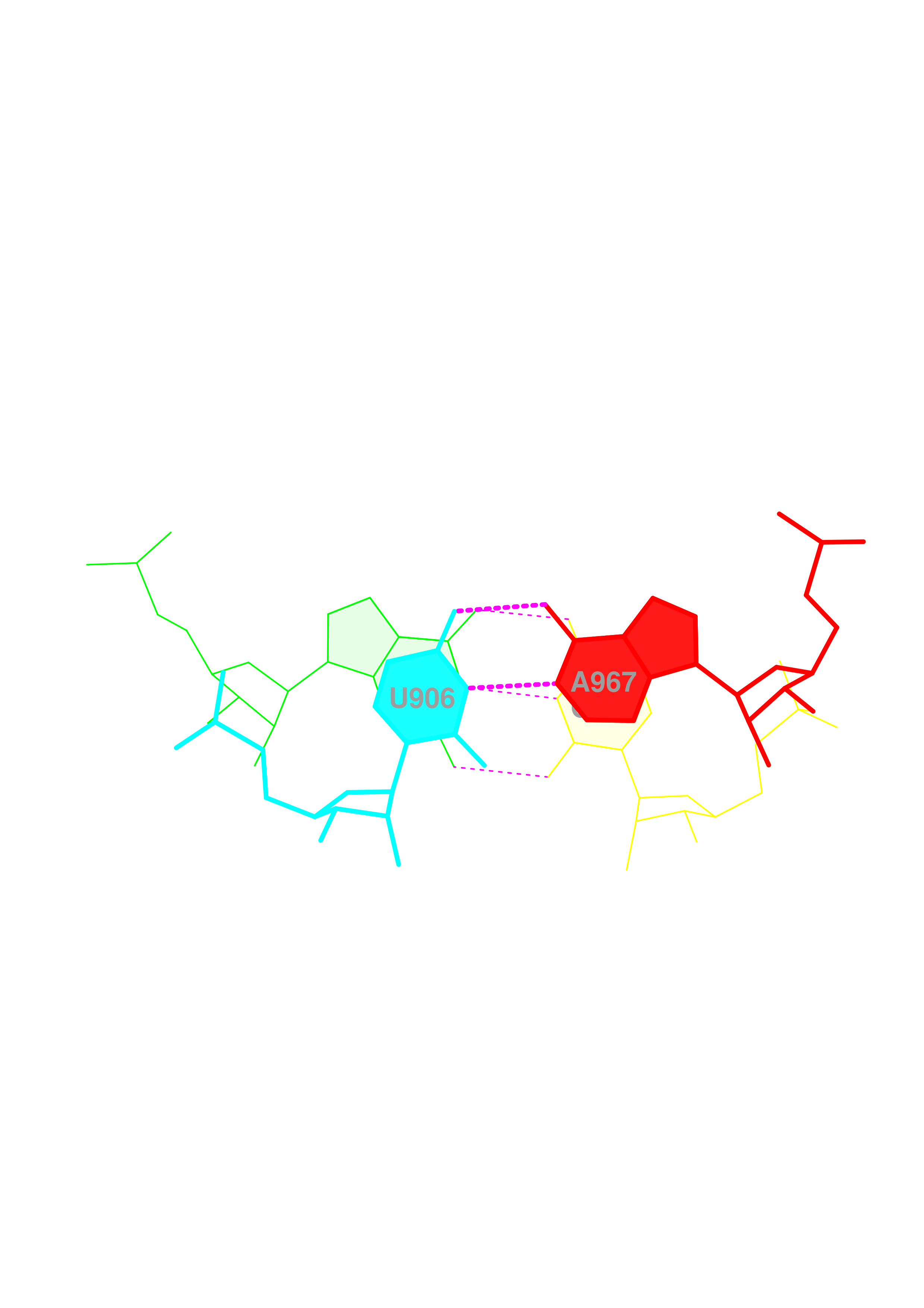
5 (0.010) ....>B:.906_:[..U]U-----A[..A]:.967_:B<.... (0.010) |
6 (0.013) ....>B:.907_:[..A]A-----U[..U]:.966_:B<.... (0.004) |
7 (0.006) ....>B:.949_:[..C]C-----G[..G]:.965_:B<.... (0.011) |
8 (0.008) ....>B:.950_:[..G]G-----C[..C]:.964_:B<.... (0.004) |
9 (0.005) ....>B:.951_:[..A]A-----U[..U]:.963_:B<.... (0.004) |
10 (0.014) ....>B:.952_:[..G]G-----C[..C]:.962_:B<.... (0.005) |
11 (0.006) ....>B:.953_:[..G]G-----C[..C]:.961_:B<.... (0.005) |
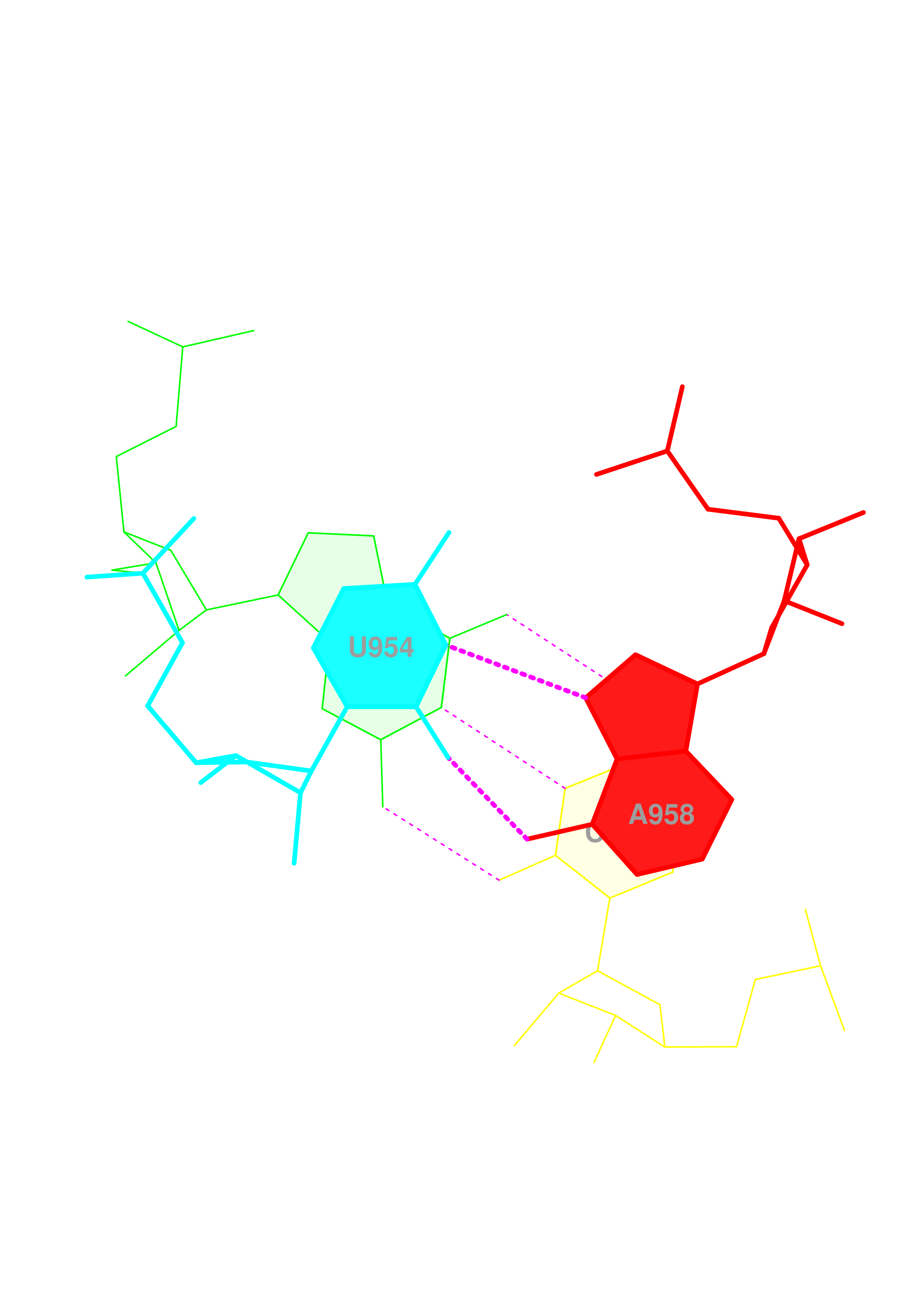
12 (0.002) ....>B:.954_:[..U]U-**--A[..A]:.958_:B<.... (0.008) |
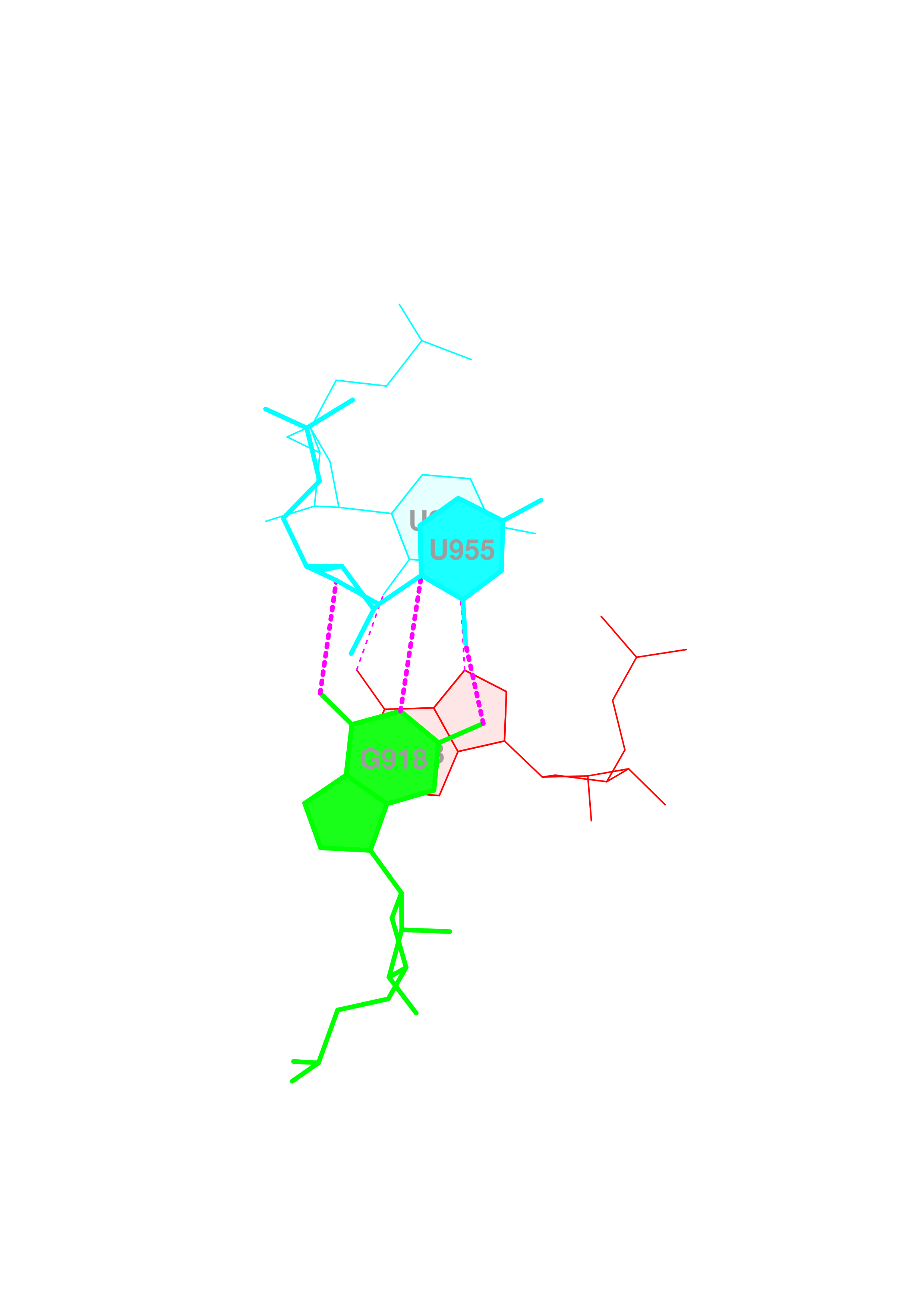
13 (0.005) ....>B:.955_:[..U]U-**+-G[..G]:.918_:B<.... (0.009) x
14 (0.007) ....>B:.937_:[..A]A-**--U[..U]:.933_:B<.... (0.003) |
15 (0.004) ....>B:.938_:[..U]U-**--U[..U]:.932_:B<.... (0.006) |
16 (0.006) ....>B:.939_:[..U]U-----A[..A]:.931_:B<.... (0.006) |
17 (0.005) ....>B:.940_:[..C]C-*---G[..G]:.930_:B<.... (0.012) |
18 (0.005) ....>B:.941_:[..C]C-----G[..G]:.929_:B<.... (0.007) |
19 (0.012) ....>B:.942_:[..G]G-----C[..C]:.928_:B<.... (0.010) |
20 (0.007) ....>B:.943_:[..G]G-----C[..C]:.927_:B<.... (0.003) |
21 (0.008) ....>B:.944_:[..A]A-**--A[..A]:.926_:B<.... (0.015) |
22 (0.013) ....>B:.910_:[..G]G-----C[..C]:.925_:B<.... (0.006) |
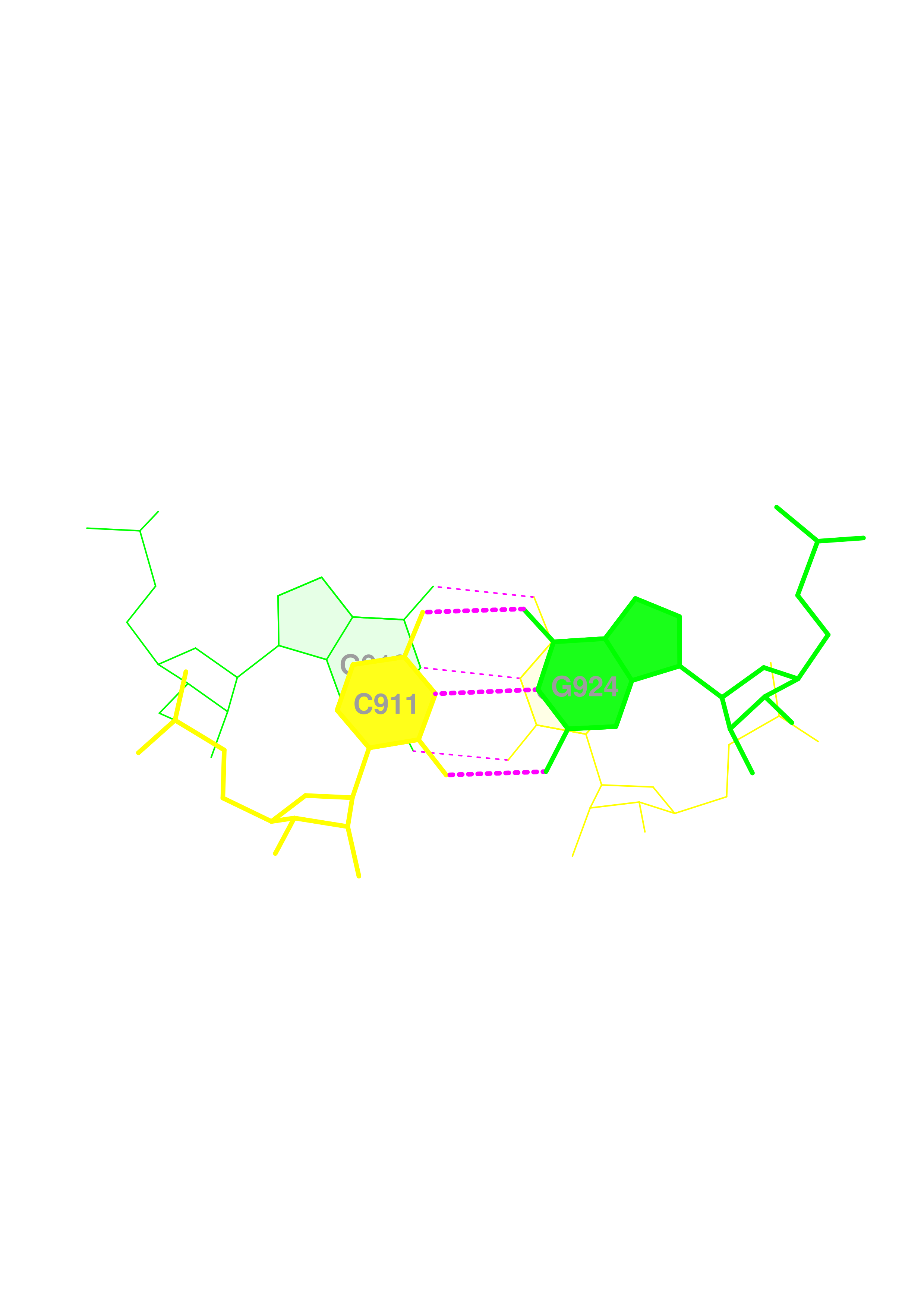
23 (0.013) ....>B:.911_:[..C]C-----G[..G]:.924_:B<.... (0.006) |
24 (0.008) ....>B:.912_:[..C]C-----G[..G]:.923_:B<.... (0.007) |
25 (0.012) ....>B:.913_:[..A]A-**+-G[..G]:.946_:B<.... (0.008) |
26 (0.006) ....>B:.914_:[..A]A-**--A[..A]:.921_:B<.... (0.008) |
27 (0.015) ....>B:.915_:[..G]G-**+-U[..U]:.948_:B<.... (0.012) x
28 (0.029) ....>B:.919_:[..G]G-----C[..C]:.956_:B<.... (0.012) +
Среди водородных связей явно видны четыре стебля:
- G902(1) - A907(6) и С971(70) - U966(65)
- C949(48) - G953(52) и G965(64) - C961(60)
- A937(36) - A944(43) и U933(32) - A926(25)
- G910(9) - C912(11) и C925(24) - G923(22)
Также из таблицы видно несколько некаконических пар оснований:
- U-G
- U-U
- A-A
- A-G
- G-U
Ну и стабилизирующие водородные связи, как я понимаю, это те связи которые не состоят в стеблях, а именно:
Strand I Strand II Helix
12 (0.002) ....>B:.954_:[..U]U-**--A[..A]:.958_:B<.... (0.008) |
13 (0.005) ....>B:.955_:[..U]U-**+-G[..G]:.918_:B<.... (0.009) x
25 (0.012) ....>B:.913_:[..A]A-**+-G[..G]:.946_:B<.... (0.008) |
26 (0.006) ....>B:.914_:[..A]A-**--A[..A]:.921_:B<.... (0.008) |
27 (0.015) ....>B:.915_:[..G]G-**+-U[..U]:.948_:B<.... (0.012) x
28 (0.029) ....>B:.919_:[..G]G-----C[..C]:.956_:B<.... (0.012) +
Интересно, что среди таких связей велика концентрация неканонических взаимодействий.
Задание 3.3
Ниже представлена информация о перекрывании азотистых оснований в данной для анализа РНК.
step i1-i2 i1-j2 j1-i2 j1-j2 sum
1 GG/CC 3.66( 2.18) 0.00( 0.00) 0.39( 0.00) 1.01( 0.21) 5.06( 2.38)
2 GG/CC 3.51( 2.12) 0.00( 0.00) 0.80( 0.00) 0.00( 0.00) 4.31( 2.12)
3 GG/CC 4.30( 2.56) 0.00( 0.00) 0.83( 0.00) 0.00( 0.00) 5.13( 2.56)
4 GU/AC 6.59( 3.65) 0.00( 0.00) 0.00( 0.00) 4.43( 2.87) 11.02( 6.52)
5 UA/UA 0.56( 0.00) 0.00( 0.00) 2.46( 1.75) 0.00( 0.00) 3.02( 1.75)
6 AC/GU 2.36( 0.94) 0.00( 0.00) 0.00( 0.00) 7.15( 4.52) 9.51( 5.46)
7 CG/CG 0.00( 0.00) 0.00( 0.00) 5.78( 2.91) 0.00( 0.00) 5.78( 2.91)
8 GA/UC 4.36( 3.00) 0.00( 0.00) 0.00( 0.00) 0.24( 0.00) 4.60( 3.00)
9 AG/CU 4.26( 3.40) 0.00( 0.00) 0.45( 0.00) 0.00( 0.00) 4.71( 3.40)
10 GG/CC 3.05( 1.56) 0.00( 0.00) 1.34( 0.00) 0.00( 0.00) 4.40( 1.56)
11 GU/AC 7.23( 4.23) 0.00( 0.00) 0.00( 0.00) 5.31( 2.58) 12.53( 6.81)
12 UU/GA 4.75( 2.36) 0.00( 0.00) 0.00( 0.00) 5.39( 3.28) 10.14( 5.65)
13 UA/UG 0.00( 0.00) 0.00( 0.00) 0.00( 0.00) 0.00( 0.00) 0.00( 0.00)
14 AU/UU 4.13( 0.55) 0.00( 0.00) 0.00( 0.00) 1.61( 0.50) 5.74( 1.04)
15 UU/AU 2.04( 0.73) 0.00( 0.00) 0.00( 0.00) 5.94( 4.50) 7.98( 5.23)
16 UC/GA 0.35( 0.08) 0.00( 0.00) 0.33( 0.00) 2.29( 0.88) 2.98( 0.96)
17 CC/GG 0.00( 0.00) 0.00( 0.00) 1.39( 0.02) 1.82( 0.45) 3.21( 0.47)
18 CG/CG 0.00( 0.00) 0.00( 0.00) 5.44( 2.67) 0.00( 0.00) 5.44( 2.67)
19 GG/CC 2.61( 1.11) 0.00( 0.00) 1.17( 0.00) 0.00( 0.00) 3.77( 1.11)
20 GA/AC 3.60( 1.16) 0.00( 0.00) 0.00( 0.00) 3.40( 1.70) 7.01( 2.86)
21 AG/CA 0.00( 0.00) 0.00( 0.00) 1.56( 0.39) 0.00( 0.00) 1.56( 0.39)
22 GC/GC 4.95( 2.09) 0.00( 0.00) 0.00( 0.00) 6.86( 3.65) 11.81( 5.74)
23 CC/GG 0.56( 0.16) 0.00( 0.00) 0.13( 0.00) 1.75( 0.45) 2.44( 0.62)
24 CA/GG 4.52( 2.04) 0.00( 0.00) 0.00( 0.00) 0.00( 0.00) 4.52( 2.04)
25 AA/AG 0.00( 0.00) 0.00( 0.00) 0.00( 0.00) 6.56( 4.29) 6.56( 4.29)
26 AG/UA 4.33( 3.52) 0.00( 0.00) 0.00( 0.00) 4.42( 3.38) 8.75( 6.90)
27 GG/CU 0.00( 0.00) 0.00( 0.00) 0.00( 0.00) 0.00( 0.00) 0.00( 0.00)
Для рассмотрения стекинг-взаимодействий я выбрал шаги с наибольшим перекрыванием - 4, 11, 12 и 22. Ниже представлены их общепринятые изображения.