Использование программы BLAST
Выбор участка сборки и информация о нем
Для тестирования фукционала BLAST я выбрал участок восемнадцатой хромосомы Bombus terrestris, с координатами 3907200..3916100, схема которого показана на рисунке 1. Хромосома 18 выбранного организма доступна по идентификатору RefSeq NC_063286.1. Ссылка на скачивание контига в FASTA формате.
Как видно из рисунка 1 на данном участке есть три CDS, обозначенных зеленым цветом, причем у одной из них есть два варианта траскрипта. Экзоны показаны более толстыми полосками, интроны более тонкими, стрелочки - направление от 5` к 3` в транскрипте. Светлозеленым цветом показаны нетранслируемые области транскрипта.

Использование различных видов BLAST
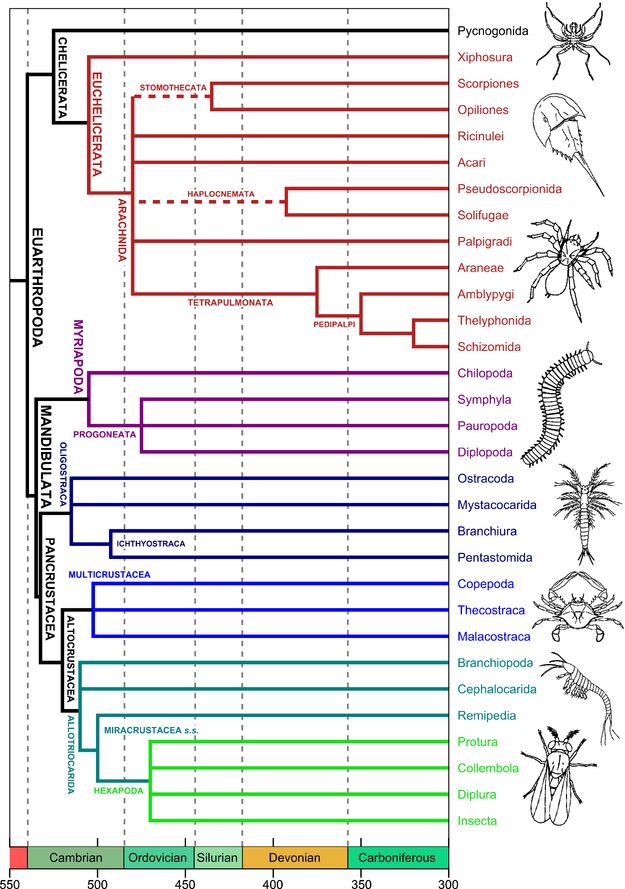
Для дальнейших манипуляций я выбрал CDS KAT8-гистонацетилтрансферазы (CDS LOC100642823 на картинке), и таксон Xiphosura - отряд реликтовых хелицеровых, филогенетические отношения этого таксона с отрядом Insecta, которому принадлежит Bombus terrestris, можно проследить на рисунке 2. Оба эти отряда относятся к типу Arthropoda.
Получив выдачу blastn, я обнаружил в ней 21 находку, среди которых содержатся в основном гистонацетилтрансферазы, и сверху списка находок располагаются именно мРНК KAT8-подобных гистонацетилтрансфераз. Снизу спска располагаются совершенно странные находки - мРНК ионного канала, АТФ-аз и фосфоглицерамутазоподобного фермента. Я не менял настройки по умолчанию, так что word size осталось равным 11. Задача, для которой я бы предложил использовать blastn, - установить гомологию бактерий по эволюции некодирующих участков.
В выдаче megablast ожидаемо получилось меньше находок - всего три. Первые две - мРНК KAT8-подобных гистонацетилтрансфераз, но вот третья - мРНК фосфоглицератмутазоподобного фермента. Интересный результат, учитывая что megablast находит крайне схожие с запросом результаты. Вероятно, получилось это, потому что я выбрал минимальный для megablast word size - 16. Обусловлен мой выбор был тем, что идентичность находок blastn запросу была низкой. Задача которую я могу придумать, чтобы решать ее с помощью megablast - проверить на консервативность участок ДНК, соответсвующая интересной вторичной стуктуре в РНК, например, самосплайсирующийся интрон.
Выдача blastx содержит 19 белков, все они - гистонацетилтрансферазы. word size, как и по умолчанию - 5. Задача, под которую я бы использовал blastx - аннотация генов.
tblastx выдал 24 находки, все из которых гистонацетилтрансферазы. Никаких странных находок не нашлось. Длина слова осталась по умолчанию - 3. Задача, для которой я бы использовал tblastx - поиск гомологичных участков в геноме малоизученных, быстро эколвюционирующих организмов, например, поиск протеаз в геноме новосеквенированного вируса.
Поиск генов рРНК в геноме Bombus terrestris
В качестве запроса я использовал данные мне последовательности 16S рРНК и 23S рРНК Escherichia coli штамма К-12. 16S рРНК является частью малой (30S) субъединицы бактериальной рибосомы, отвечает за узнавание последовательности Шайна-Дальгарно и стабилизирует правильное кодон-антикодоновое взаимодействие в А-сайте рибосомы. 23S рРНК - составляющая большой (50S) субъединицы бактериальной рибосомы - отвечает за транспептидацию и в целом играет важную роль в формировании P-сайта.
Для поиска гомологичных генов я пользовался blastn, так как организмы запроса и поиска дальнородственные, также Bombus terrestris - эукариот, а значит в генах помимо экзонов, встречаются экзоны, по этим двум причинам megablast отпадает, а остальные виды BLAST, не подходят, так как каким-либо образом связаны с белками, здесь же мы имеем дело с генами РНК. Я увеличивал параметр word size пока поиск не перестал выдавать результаты для 23S рРНК, последнее значение, при котором выдача была использовал для окончательного результата, это значение - 17. Ограничение по e-value я выбрал - 0.001
Сколько бы я не понижал word size, оба гена упорно не хотели выравниваться на хромосомы с хорошим e-value, зато они отлично выравнивались на неразмещенные скаффолды сборки, которых в сборке 231. Ссылка на выдачу blastn. Еще я заметил, что каждый из генов рРНК выравнялся ровно на четыре скаффолда с одинаковым весом и e-value. Предположу, что эти бактериальные гены рРНК отравнялись на митохондриальные гены рРНК, а разница между эукариотическими генами рРНК и генами в запросе слишком велика, чтобы появились хиты на хромосомах. Собственно, эта гипотеза базируется на предположении, что некоторые неразмещенные скаффолды - митохондриальные геномы (или может их k-меры).
Посмотрев на выдачу повнимательнее, я заметил, что выравниваются одни и те же участки рРНК E. coli на различные участки в неразмещенных скаффолдах, предположу, что эти участки рРНК - консервативнее всего, а неразмещенные скаффолды в таком случае - k-меры какой то крупной последовательности, может как раз митохондриального генома. С другой стороны, автоматическая аннотация аннотировала в этих скаффолдах 5.8S рРНК, не характерную для митохондрий, так что версии, почему я получил такую выдачу иссякли. По автоматической аннотации неразмещенных скаффолдов я понял, что действительно 16S рРНК отравнялась в места с аннотацией - small subunit ribosomal RNA, а вот участки, куда отравнялась 23S рРНК никак не аннотирована.
