Работа с HMM профилями
Создание HMM профиля и поиск по нему
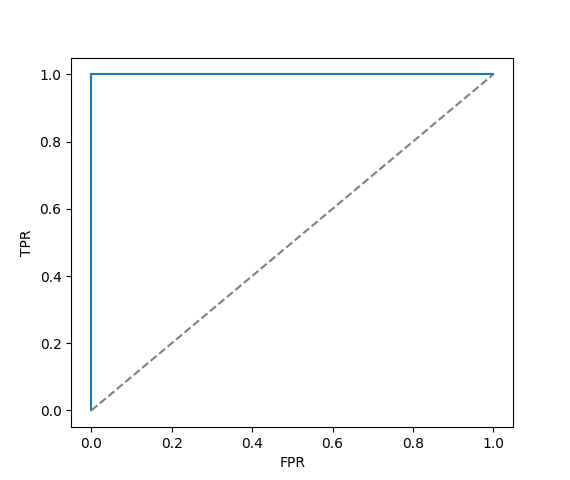
Для изучения я выбрал трипсиновый домен (PF00089), и его архитектуру PF00089 - PF03797. Я скачал все белки с такой архитектурой, выровнял их с помощью mafft и сохранил в формате sto. Провел ревизию выравнивания - удалил фрагменты с крупными делециями (файл на кодомо ~/term4/pr11/domain.sto) и выделил группу с redundancy больше 90% в отдельную выборку - впоследствии она станет положительным контролем (файл на кодомо ~/term4/pr11/positive.fasta). Далее на kodomo составил из выравнивания, над которым провел ревизию (файл на кодомо ~/term4/pr11/domain.sto), профиль HMM с помощью HMM3(файл на кодомо ~/term4/pr11/my_alignment-hmm3.hmm). Далее я скачал все 889 белков, содержащих трипсиновый домен и имеющих аннотацию (файл на кодомо ~/term4/pr11/proteins.fasta). Загрузил их на кодомо и провел поиск по HMM профилю среди этих 889 белков и 42 белков (файл на кодомо ~/term4/pr11/all.fasta), ранее удаленных из выравнивания по redundancy, не задавая порог. Мне было интересно получить все находки и их score, чтобы впоследствии проанализировать данные и выбрать нужный порог (файл на кодомо ~/term4/pr11/domains.tsv). Получив результаты, с помощью скрипта распарсил выдачу и построил ROC-кривую, которая изображена на рисунке 1. AUC ROC такого классификатора составила ровно единицу. Таким образом, классификация белков с трипсиновым доменом на содержащие данную архитектуру и не содержащие таковую по скору HMM-профиля идеальна для данной выборки, а значит существует такой порог, скор выше которого получают только белки, содержащие данную архитектуру - этот скор равен 747. Не вижу смысла в дополнительном подборе порога, так как классификатор и так идеален.