Трансмембранные белки
Задание 1
Для изучения я выбрал белок гасдермин B, относящийся к семейству гасдерминов. Его английское название: gasdermin B, индентификатор PDB: 8GTN, идентификатор UniProt: GSDMB_HUMAN. Исследуемый белок принадлежит человеку (Homo sapiens) и располагается на клеточной мембране. Белок задействован в таком процеесе, как пироптоз - обусловленная воспалением клеточная смерть. В процессе пироптоза с помощью длинного сигнального каскада от субъединиц гасдерминов отрезаются C-концевые домены, которые впоследствии активируют следущие стадии пироптоза, а субъединицы без C-концевых доменов оказываются сродными мембране, заходят в нее и гомоолигомеризуются с образованием достаточно крупных пор (10-15 нм, зависит от количества субъединиц). Через эти поры содержимое клетки покидает ее, и клетка погибает.
Координаты трансмембранных участков одинаковы у всех субъединиц: 1( 94- 98), 2( 105- 110)
Найдя в UniProt последовательность и место сайта вышеупомянутого разрезания, я обрезал последовательность и подал ее на вход DeepTMHMM. Допуская, ошибочность предположения о необходимости обрезать белок, подал и полную последовательность. Результаты представлены ниже.
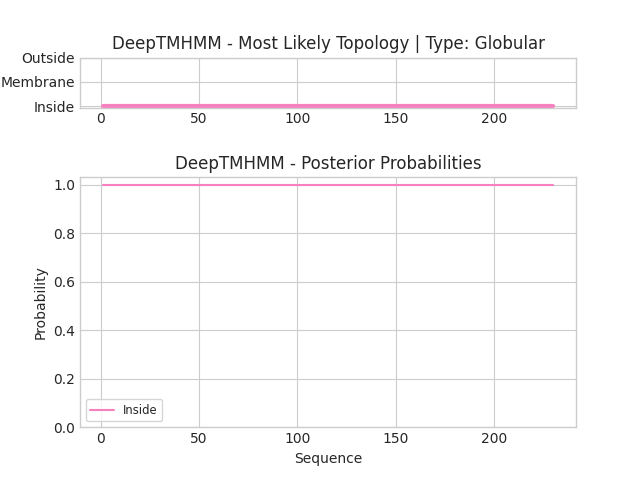
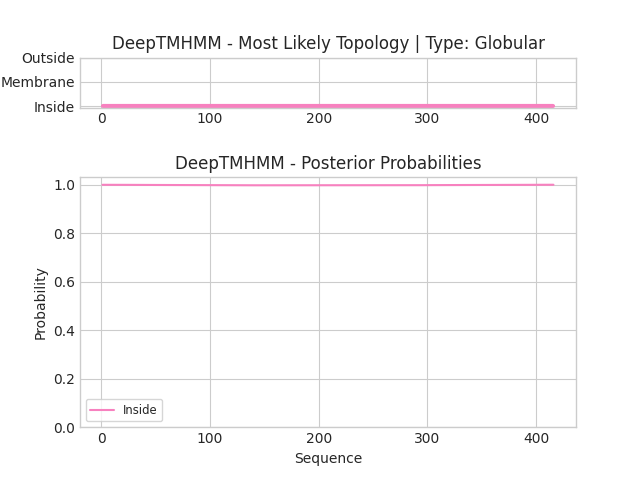
Результат огорчил: по мнению программы никаких трансмембранных доменов в белке нет. Cссылка на скачивание gff3 выдачи для обрезанного белка. Очевидно, для полной последовательности текстовая выдача аналогична. Расшифровать ее несложно: весь белок лежит внутри мембраны. DeepTMHMM абсолютно не справилась с предсказанием трансмембранного домена. Вероятно, это связано с тем, что на вход я подавал последовательность одной субъединицы.
Задание 2
Для изучения мне выдали траспортер D-арабинитола из Klebsiella pneumoniae. Его английское название: D-arabinitol transporter, идентификатор SwissProt: DALT_KLEPN. Белок располагается во внутренней мембране клетки (бактерия грамотрицательна) и транспортирует внутрь клетки D-арабинитол.
Найдя в UniProt последовательность белка, я подал ее на вход DeepTMHMM. Результаты представлены ниже.
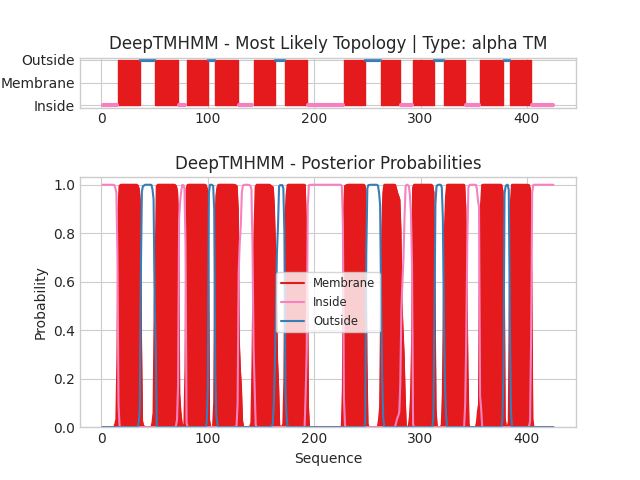
Тут уже сильно лучше. Cссылка на скачивание gff3 выдачи для выданного белка. На графике видно 12 трансмембранных доменов, предложенный класс топологии: альфа-трасмембранный белок. В целом, это соотносится с его изображением с помощью AlphaFold, а также с другими родственными белками. В выдаче программы в gff3 формате легко увидеть координаты найденных трансмембранных доменов (строчки с меткой TMhelix):
##gff-version 3
# sp|O52718|DALT_KLEPN Length: 425
# sp|O52718|DALT_KLEPN Number of predicted TMRs: 12
sp|O52718|DALT_KLEPN inside 1 15
sp|O52718|DALT_KLEPN TMhelix 16 36
sp|O52718|DALT_KLEPN outside 37 49
sp|O52718|DALT_KLEPN TMhelix 50 72
sp|O52718|DALT_KLEPN inside 73 79
sp|O52718|DALT_KLEPN TMhelix 80 100
sp|O52718|DALT_KLEPN outside 101 106
sp|O52718|DALT_KLEPN TMhelix 107 128
sp|O52718|DALT_KLEPN inside 129 142
sp|O52718|DALT_KLEPN TMhelix 143 163
sp|O52718|DALT_KLEPN outside 164 172
sp|O52718|DALT_KLEPN TMhelix 173 193
sp|O52718|DALT_KLEPN inside 194 227
sp|O52718|DALT_KLEPN TMhelix 228 248
sp|O52718|DALT_KLEPN outside 249 262
sp|O52718|DALT_KLEPN TMhelix 263 281
sp|O52718|DALT_KLEPN inside 282 292
sp|O52718|DALT_KLEPN TMhelix 293 313
sp|O52718|DALT_KLEPN outside 314 321
sp|O52718|DALT_KLEPN TMhelix 322 342
sp|O52718|DALT_KLEPN inside 343 355
sp|O52718|DALT_KLEPN TMhelix 356 378
sp|O52718|DALT_KLEPN outside 379 383
sp|O52718|DALT_KLEPN TMhelix 384 404
sp|O52718|DALT_KLEPN inside 405 425
Дальше предсказанную AlphaFold структуру я подал на вход PPM 3.0, указав внутреннюю мембрану грам-отрицательных бактерий в качестве мембраны, внешнюю локализацию N-конца (наложил построенную AlphaFold структуру на структуру родственных белков в базе OPM), остальные параметры оставил по умолчанию. Выдача порадовала: 1( 16- 40), 2( 47- 69), 3( 77- 100), 4( 105- 127), 5( 144- 166), 6( 171- 194), 7( 228- 252), 8( 260- 282), 9( 291- 314),10( 320- 340),11( 357- 382),12( 383- 403). PPM также нашел 12 трансмембранных доменов, сильно похожих по координатам на те, что нашла DeepTMHMM (отличия в координатах начала и конца на 1-2 аминокислоты). Считаю результаты сходящимися.
