Базы данных
Введение
Для анализа мне дан набор из 29 белков (ссылка на скачивание), состоящий из различных цитохромов Р450 - белков окисляющих различые субстраты и фосфолипаз - белков, отщепляюих фосфат от фосфолипидов, но я не знал этого до проведения аанализа по функциям в базах данных (это исправленная версия), так что примем, что первой целью моего анализа стало выяснить функции этой группы белков, и установить, что это за белки (как будто я этого не знаю).
База данных Reactome
Я решил что лучше всего сверхпредставленность по функциям покажет интерфейс базы данных Reactome. Уж очень я люблю диаграммы Вороного. Собственно, путем несложных махинаций, я отправил запрос и достаточно быстро получил красивую интерактивную диаграмму Вороного, представленную на рисунке 1. На рисунках 2, 3, 4, 5 и 6 представлены отдельные интересные ее фрагменты.
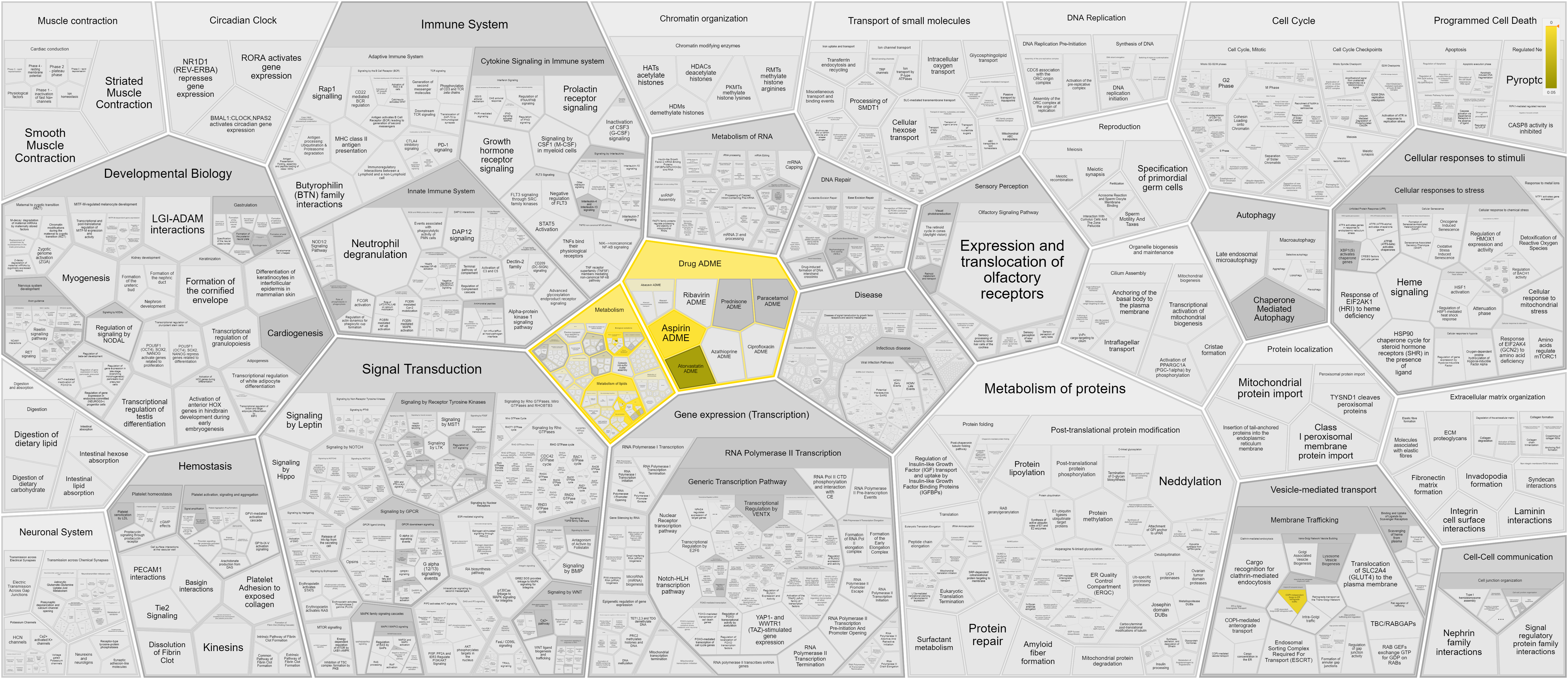
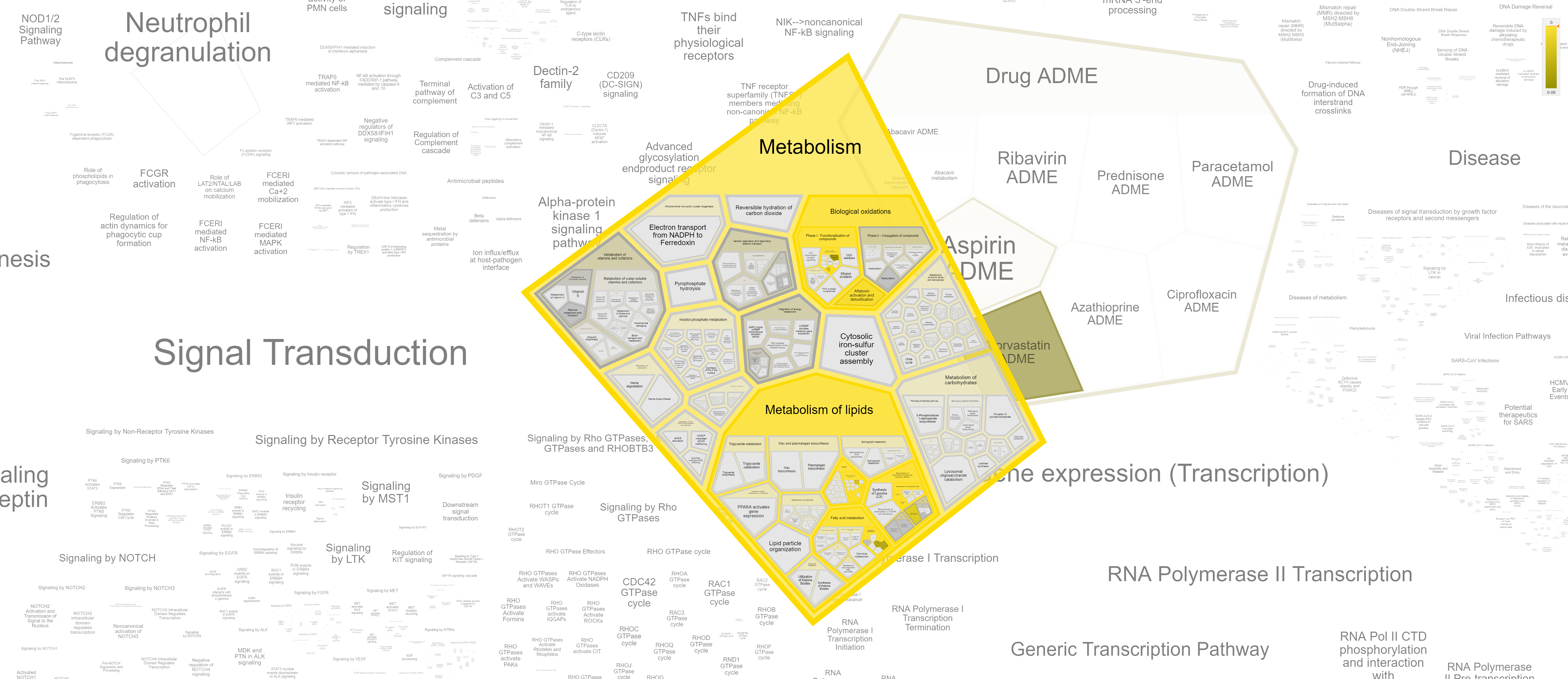
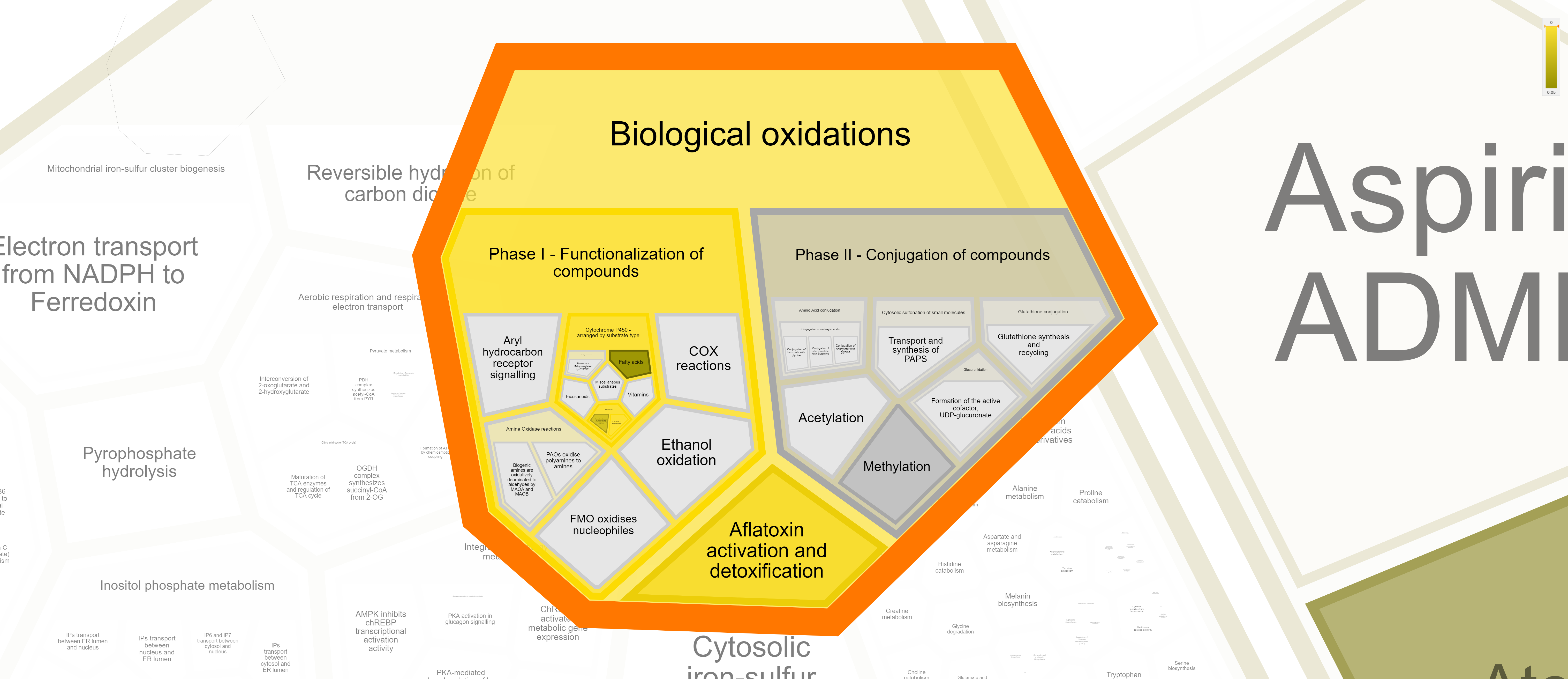
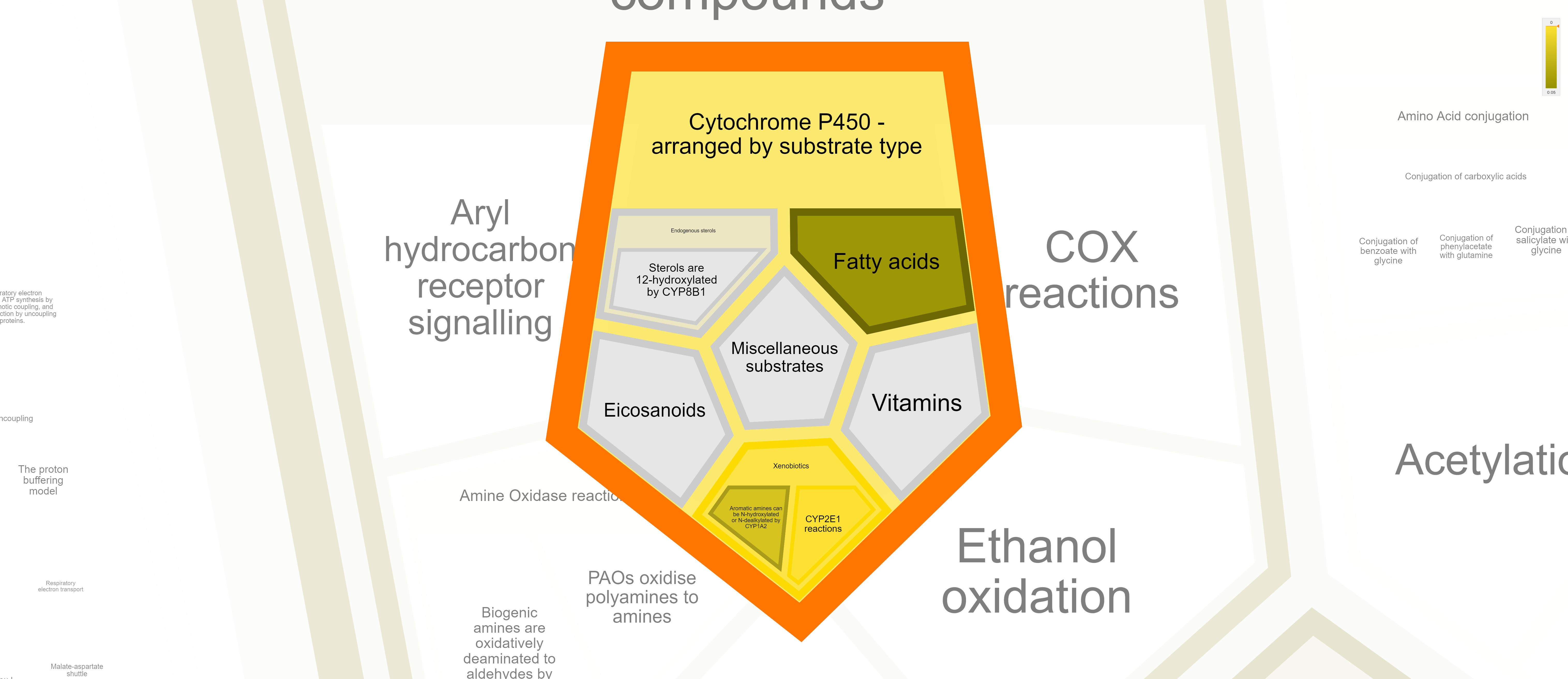
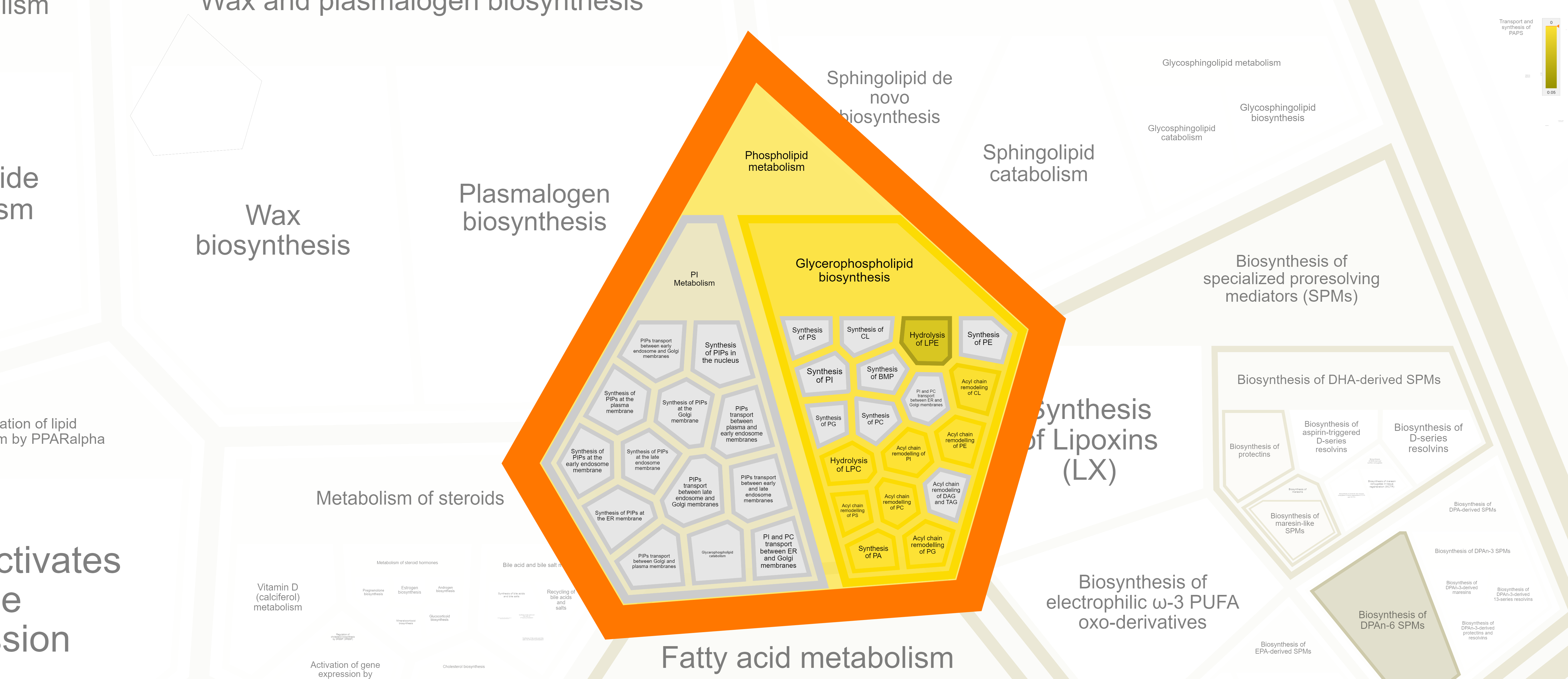
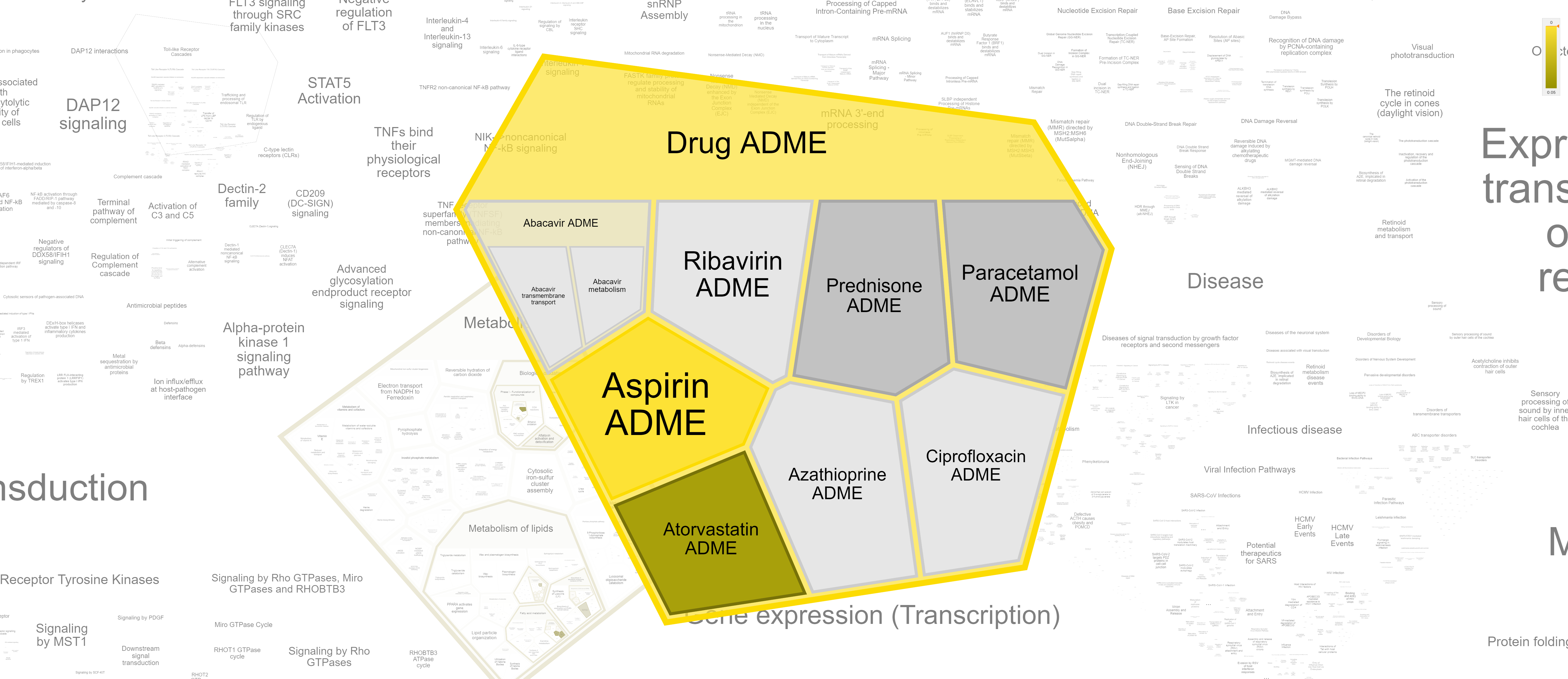
Как можно видеть из рисунков, основные сверхпредставленные функции связаны с метаболизмом лекарств (в основном аспирина), метаболизмом фосфолипидов и биоокислением, причем с помощью цитохрома P450. В таком случае, предположу, что белки, название которых, начинается с CYP - и есть цитохромы P450, тогда не удивительно, что все эти белки отвечают термину биоокисление - ведь это основная функкция цитохромов P450. Этим же объясняется и сверхпредставленность метаболизма лекарств - цитохром P450 разлагает их.
Остальные белки, начинающиеся с PL, видимо, как-то задействованы в метаболизме фосфолипидов - предполагаю, что это фосфолипазы A (PLA...) и фосфолипазы B (PLB...)
Остаются два белка с непохожими ни на что названиями. ALOX... задействован в окислении жирных кислот с помощью молекулярного кислорода - пусть будет липоксигеназа. Для белка JMJD7-PLA2G4B сверхпредставленности не нашлось.
Все гипотезы подтвердились после заглядывания в UniProt.
Таким образом базу Reactome удобно использовать для анализа и красивого представления функциональной сверхпредставленности различных GO-терминов.
База STRINGS
Я знаю что у базы STRINGS есть функционал кластеризации и показательной ее визуализации на графе, который я хотел бы попробовать. Ожидаемые результаты - два кластера: цитохромы + липооксигеназа (оба используют молекулярный кислород для окисления), фосфолипазы (включая странную JMJD7-PLA2G4B). Запустил поиск и кластеризовал с помощью k-средних на два кластера. Результаты представлены на рисунке 7.

Результаты получились несколько неожиданные: в кластер к цитохромам и липоксигеназе (которые, как я и предсказывал оказались вместе) попали некоторые фосфолипазы, включая фосфолипазу со странным названием. Посмотрев итоговую выдачу в текстовом формате, стало понятно, что в кластер к цитохромам попали фосфолипаза со странным названием, которая на самом деле дуплекс из двух белков, поэтому не имеет фосфолипазной активности, неактивная фосфолипаза, две фосфолипазы А2 - дзета и эпсилон (наверное, то, что это две последние буквы встречающиеся в названиях фосфолипаз А2, что-то значит) и кальций-независимая фосфолипаза А2 - единственная таковая среди представленных белков. Таким образом, кластеры разделились неплохо, но тогда возникает подозрение, что если попробовать кластеризовать на три кластера - в цитохромном кластере останутся только оксигеназы (цитохромы и липоксигеназа). Результаты кластеризации на три кластера представлены на рисунке 8.

И действительно: третий кластер собрал функциональные "выбросы" первого и второго кластеров.
Таким образом, помимо прочего, база STRINGS способна осмысленно кластеризовать объекты, что бывает полезно.
Выводы
Проанализировав свой набор белков, я выяснил, что обогащение терминами GO, вполне можно использовать для выявления функций, преобладающих в наборе белков. Также, я попользовался базами данных Reactome и STRINGS и наглядно оценил их способоность представлять данные. Reactome удобно использовать для анализа и красивого представления функциональной сверхпредставленности различных GO-терминов. Это может быть полезно на ранних этапах иссследования набора белков. STRINGS способна демонстрировать различные виды связи между элементами набора и помимо этого осмысленно кластеризовать объекты, что бывает полезно.
