Для исследования была выбрана цепь C, представляющая Glu тРНК со следующей последовательностью:
[1] 5'G G C C C C A U C G U C U A G C G G U U A G G A C G C G G C C C U C U C A A G G C C G A A A C G G G G G U U C G A U U C C C C C U G G G G U C A C C A 3'[75], Где 1 и 75 - номера первого и последнего нуклеотида соответственно.
В конце последовательности на 3' конце есть ССА триплет, координаты его атомов приведены.
Акцепторный стебель состоит из участка 501-507 и комплиментарного ему участка 572-566
Т-стебель состоит из участка 549-553 и комплиментарного участка 565-561
D-стебель состоит из участка 510-513 и остатка 515 (514 не образует водородных связей) и комплиментарного ему участка 525-521
Антикодоновый стебель состоит из участка 526-531 и комплиментарного ему участка 539-544
Антикодоном является триплет 534-536 и кодирует аминокислоту лейцин
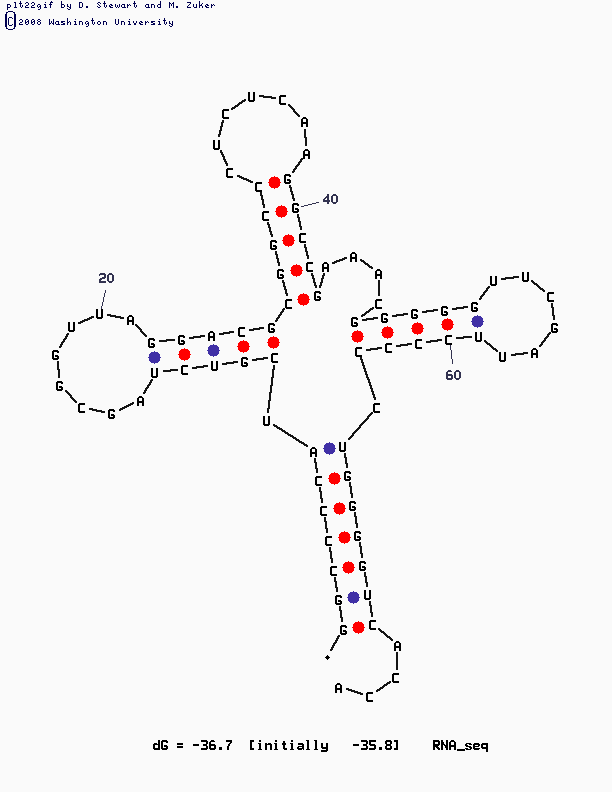
| Рис.1. Вторичная структура лейциновой тРНК из 2cv1.pdb | Скрипт для получения изображения |
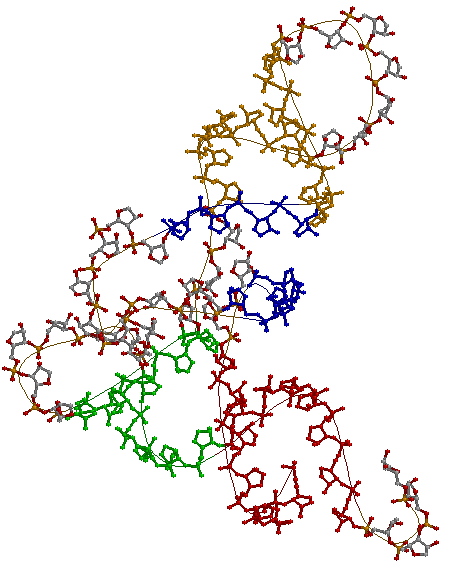 |
Хочу заметить, что РНК была заранее вырезана мною из файла 2cv1.pdb background white restrict backbone select (501-507 or 566-572) and backbone color red select (549-553 or 561-565) and backbone color green select (510-513 or 522-525) and backbone color blue select (526-531 or 539-544) and backbone color orange |
Неканонические пары: 2 пары G-U и две пары G-A.
| Неканоническая пара G-A (526 - 544) | Неканоническая пара G-U (502 - 571) |
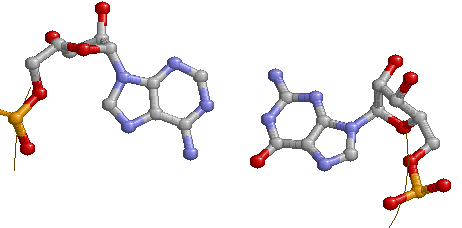 | 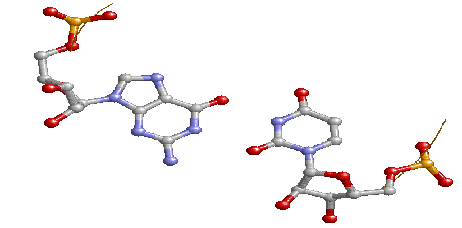 |
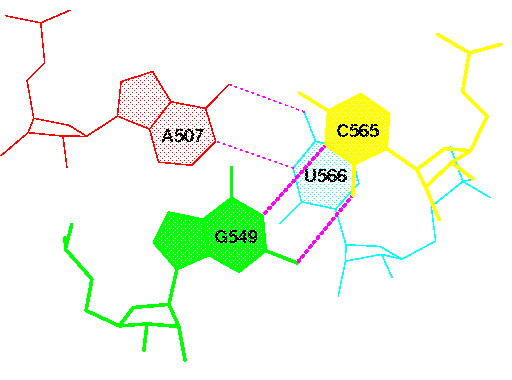
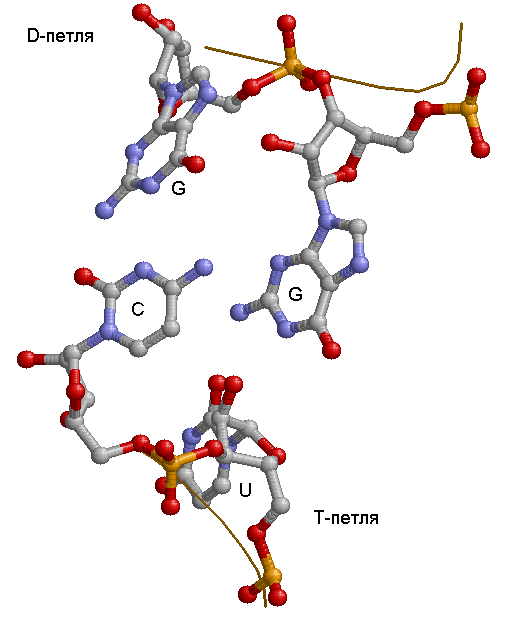
D и T петли находятся близко друг к другу, что обуславливает установление водородных связей между остатками 519-556 и 518-555, схема связей приведена ниже. Первая пара - G-C, вторая - неканоническая G-U.
| Участок структуры | Позиции в структуре (по результатам find_pair) |
Результаты предсказания с помощью einverted |
Результаты предсказания по алгоритму Зукера |
| Акцепторный стебель | 5' 501-507 3' 5' 566-572 3' |
предсказано 2 пары из 7 реальных | предсказано 7 из 7 |
| D-стебель | 5' 510-513, 515 3' 5' 521-525 3' |
предсказано 5 из 5 | |
| T-стебель | 5' 549-553 3' 5' 561-565 3' |
предсказано 5 из 5 | |
| Антикодоновый стебель | 5' 526-531 3' 5' 539-544 3' |
предсказано 5 из 6 реальных | |
| Общее число канонических пар нуклеотидов | 19 | 17 |