Настоящий мини-обзор представляет из себя работу с протеомом и геномом анаэробной почвенной бактерии Clostridium sporogenes с использованием методов программирования на языке Python, функционала электронных таблиц. Полученные данные объясняются различными биоинформатиками закономерностями.
Keywords: Clostridium sporogenes, анализ генома, анализ протеома
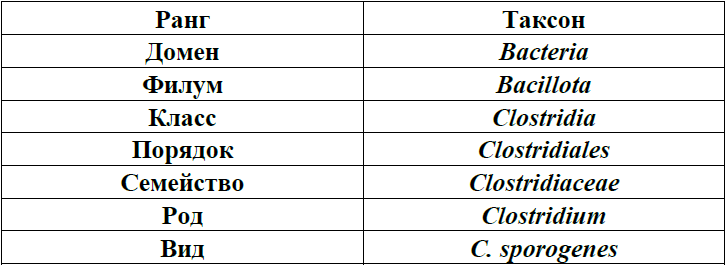
Бактерия Clostridium sporogenes открыта И. И. Мечниковым в 1908 году. Это грамположительная бактерия рода Clostridium (Таблица 1).
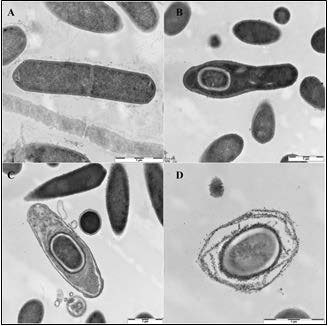
Это почвенная анаэробная палочковидная бактерия, производящая овальные субтерминальные эндоспоры (Рис. 1). Clostridium sporogenes является нетоксигенной бактерией по сравнению с Clostridium botulinum, которая производит нейротоксины. Но C. Sporogenes тоже является важным агентом порчи пищевых продуктов (Brown et al., 2012). У животных и человека бактерия носит не патогенный, а мутуалистический характер.
C. sporogenes используется в качестве нетоксигенного заменителя C. botulinum при тестировании эффективности коммерческой стерилизации. Так как C. sporogenes образует споры с очень высокой термостойкостью (Taylor et al., 2013).
C. sporogenes может быть способом доставки противораковых лекарств к опухолям пациентов (Nuyts et al., 2002).
Также было выявлено синтезирование этой бактерией биоактивных индолсодержащих соединений, полученных из триптофана, таких как индоксилсульфат и антиоксидант индол-3-пропионовая кислота (Wikoff et al, 2009).
В настоящем мини-обзоре рассматриваются и анализируются базовые особенности генома и протеома бактерии с помощью биоинформатических методов. Исследуются кодирующие белок последовательности и составляющие их нуклеотиды, особенности распределения последовательностей по кольцевой ДНК бактерии и распределение белков.

Геномная таблица (feature_table.txt.gz), Последователь-ность генома (genomic.fna. gz) и другая информация о бактерии была скачена с сайта Национального Центра Биотехнологической информации (NCBI).
Для анализа данных использовались программы, написанные на языке программирования Python, и также применялись электронные таблицы Google Sheets. Ссылки на программы Python и электронные таблицы можно найти в разделе “Сопроводительные материалы”.
Раздел 3.1: Первая программа Python.
Раздел 3.2: Вторая программа Python.
Раздел 3.3: Лист «Распределение генов» в ЭТ.
Раздел
3.4: Лист «Гистограмма» и «CDS».
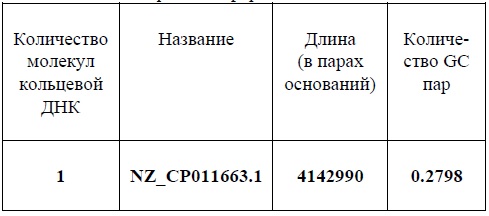
С помощью первой программы и файла genomic.fna.gz можно узнать количество последовательностей, назва-ние, длину и количество GC-пар в составе генома Clostridium sporogenes (Таблица 2). Последовательность в геноме одна – это кольцевая хромосома NC_022571.1. 5107814 пар нуклеотидов составляют длину хромосомы C. sporogenes. Количество GC-пар от общего количества составляет 27,98%. Именно относительно низкое со-держание GC-пар в геноме служит отличимым призна-ком для всей группы Firmicutes (Briggs et al., 2012), ча-стью которой и является Clostridium sporogenes.
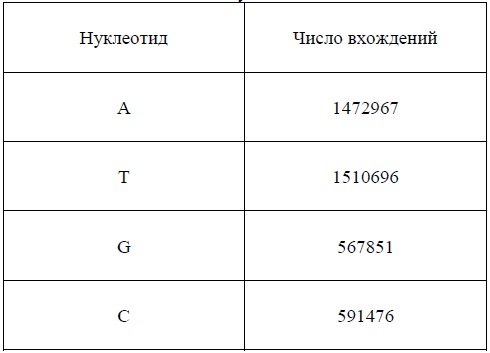
Воспользовавшись второй программой и тем же файлом, можно найти нуклеодидный состав всей ДНК (Таблица 3). Данные таблицы показывают, что для ДНК Clostridium sporogenes выполняется второе правило Чаргаффа – содержание гуанина приблизительно равно содержанию цитозина, а содержание аденина - содержанию тимина (Rudner et al, 1968).
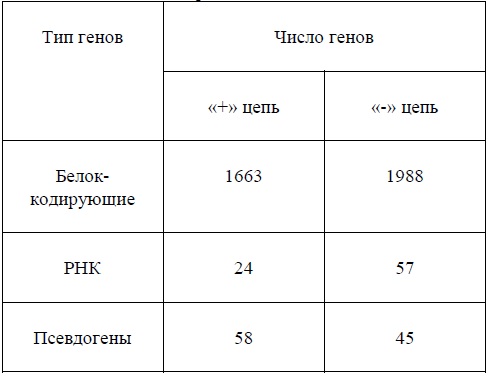
Используя ЭТ Google Sheets и файла feature_table.txt.gz получилась таблица по распределению генов на «+» и «-» цепях (Таблица 4)
По полученным данным можно увидеть небольшие раз-личия между распределением белок-кодирующих генов, Высокую разницу между РНК и незначительную между псевдогенами.
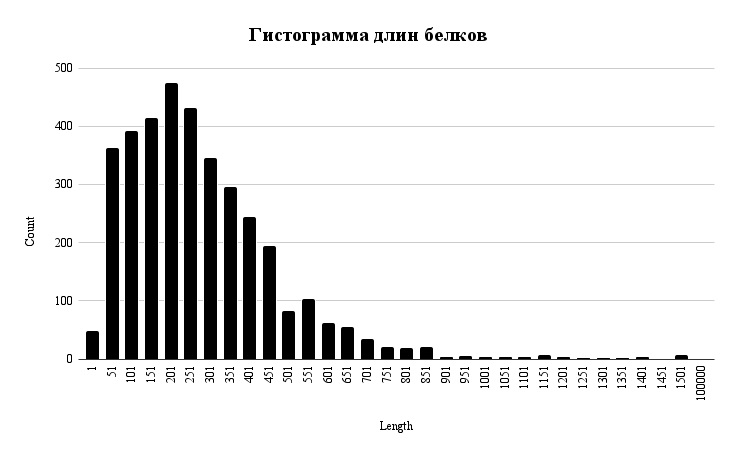
С помощью электронных таблиц и функции COUNTIFS мож-но построить гистограмму длин белков, кодируемых геномом бактерии (График 1).
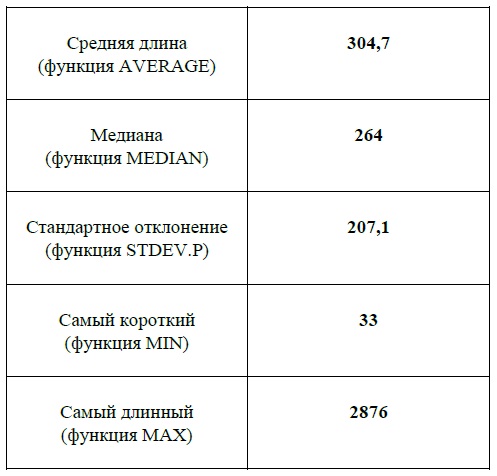
Также с помощью электронных таблиц (Таблица 5) найдены некоторые статистические параметры для распределения длин белков, такие как:
• Средняя длина
• Медиана
• Стандартное отклонение
• Самый короткий фрагмент
• Самый длинный фрагмент
Ссылка на Google Colaboratory (Colab)
Ссылка на Google Sheets
Brown, J. L., Tran-Dinh, N., and Chapman, B. (2012). Clostridium sporogenes PA 3679 and its uses in the derivation of thermal processing schedules for low-acid shelf-stable foods and as a research model for proteolytic Clostridium botulinum. J. Food Prot. 75, 779–792. doi: 10.4315/0362-028X.JFP-11-391
Taylor, R. H., Dunn, M. L., Ogden, L. V., Jefferies, L. K., Eggett, D. L., and Steele, F. M. (2013). Conditions associated with Clostridium sporogenes growth as a surro-gate for Clostridium botulinum in nonthermally processed canned butter. J. Dairy Sci. 96, 2754–2764. doi: 10.3168/jds.2012-6209
Nuyts, S., Van Mellaert, L., Theys, J., Landuyt, W., Lambin, P., and Anné, J. (2002). Clostridium spores for tumor-specific drug delivery. Anticancer Drugs 13, 115–125. doi: 10.1097/00001813-200202000-00002
Wikoff WR, Anfora AT, Liu J, Schultz PG, Lesley SA, Peters EC, Siuzdak G (March 2009). "Metabolomics analysis reveals large effects of gut microflora on mamma-lian blood metabolites". Proc. Natl. Acad. Sci. U.S.A. 106 (10): 3698–3703. Bib-code:2009 PNAS.106.3698W. doi:10.1073/pnas.0812874106. PMC 2656143.
Briggs GS, Smits WK, Soultanas P. Chromosomal replication initiation machinery of low-G+C-content Firmicutes. J Bacteriol. 2012 Oct;194(19):5162-70.
Rudner R, Karkas JD, Chargaff E. Separation of B. subtilis DNA into complementary strands. 3. Direct analysis. Proc Natl Acad Sci U S A. 1968 Jul;60(3):921-2.