Код доступа проекта по секвенированию бактерии Buchnera aphidicola - SRR4240359.
С сайта ENA, я скачал соответствующий файл в формате fastq.
Команда:
wget ftp://ftp.sra.ebi.ac.uk/vol1/fastq/SRR424/009/SRR4240359/SRR4240359.fastq.gz
Затем я создал файл в формате fasta со всеми возможными адаптерами, которые нужно вырезать из прочтений.
Команда:
cat /mnt/scratch/NGS/adapters/* > adapters.fasta
Далее я удалил адаптеры (в adapters.fasta) из чтений в файле SRR4240359.fastq.gz с помощью программы Trimmomatic.
Команда:
java -jar /usr/share/java/trimmomatic.jar SE -threads 10 -phred33 -trimlog trim.log SRR4240359.fastq.gz SRR4240359_trimmed_adapt.fastq.gz ILLUMINACLIP:adapters.fasta:2:7:7
| Изначально было | 13557938 чтений |
| Осталось | 13502066 (99.59%) чтений |
| Удалилось | 55872 (0.41%) чтений |
Потом с 3'-конца чтений я удалил нуклеотиды, которые не прошли порог качества (имели качество меньше 20), сохраняя при этом минимальную длину в 32 нуклеотидов.
Команда:
java -jar /usr/share/java/trimmomatic.jar SE -threads 10 -phred33 -trimlog trim2.log SRR4240359_trimmed_adapt.fastq.gz SRR4240359_full_trimmed.fastq.gz TRAILING:20 MINLEN:32
| Изначально было | 13502066 чтений |
| Осталось | 12184080 (90.24%) чтений |
| Удалилось | 1317986 (9.76%) чтений |
С помощью программы velveth я сформировал директорию (Assembly_SRR4240359) с k-мерами (k-меры длиной 31) из обработанных чтений.
Команда:
velveth Assembly_SRR4240359 31 -short -fastq.gz SRR4240359_full_trimmed.fastq.gz
В конечном итоге с помощью программы velvetg я сделал сборку.
Команда:
velvetg Assembly_SRR4240359
N50 и его длина
| N50 | 70607 |
Длины трёх самых длинных контигов
| Номер узла (контига) | Длина контигов | Покрытие |
| 11 | 125674 | 44.550949 |
| 1 | 108447 | 42.009184 |
| 14 | 71403 | 39.411551 |
Контиги с аномально большим или аномально малым покрытием
| Номер узла (контига) | Длина | Покрытие |
| 609 | 31 | 2.032258 |
| 80 | 40 | 109.500000 |
Нужную информацию я получил с помощью слудующих команд:
tail -1 Log (Отсюда я узнал N50)
sort -n -r -k 2 stats.txt | cut -f 2,6| head -n 3 (Длина и покрытие самых длинных контигов)
grep "125674" contigs.fa (Искал по длинам в contigs.fa)
grep "108447" contigs.fa (Аналогично)
grep "71403" contigs.fa (Аналогично)
(Причечательно, что покрытие в stats.txt и contigs.fa отлючаются на одну миллионную, в таблицу записал из stats.txt)
sort -n -k 6 stats.txt | less (Просматривал контиги с длиной больше 31)
grep '2.032258' contigs.fa (И искал по их покрытиям в contigs.fa)
sort -n -r -k 6 stats.txt | less (Аналогично)
grep '109.500000' contigs.fa (Аналогично)
grep -A 2100 '>NODE_11_length_125674_cov_44.550949' contigs.fa (Для удобного копирования последовательности из терминала)
grep -A 1810 '>NODE_1_length_108447_cov_42.009186' contigs.fa (Аналогично)
grep -A 1200 '>NODE_14_length_71403_cov_39.411552' contigs.fa (Аналогично)
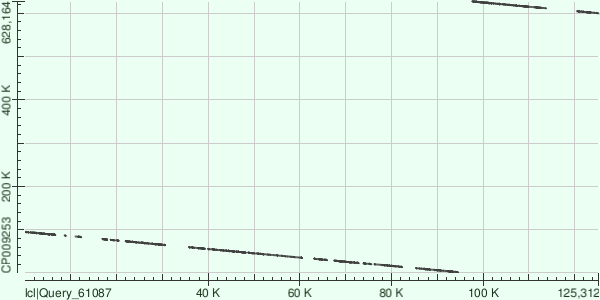
Ниже представлены карты локального сходства, полученные в результате выравнивания алгоритмом megablast с длиной слова 28 по отдельности трёх самых длинных контигов на геном Buchnera aphidicola (AC: CP009253). Каждые контиги картировались по 25, 15 и 14 участкам соответственно. Между ними участки, не имеющие гомологов в геноме. Их характеристики также представлены ниже.
| Фрагмент генома | Длина | Покрытие |
| 35124 to 44693 | 7981/9633(83%) | 130/9633(1%) |
| 2004 to 11103 | 7229/9223(78%) | 256/9223(2%) |
| 613658 to 620926 | 5845/7379(79%) | 184/7379(2%) |
| 47158 to 55420 | 6440/8436(76%) | 301/8436(3%) |
| 64632 to 70621 | 4703/6151(76%) | 274/6151(4%) |
| 599832 to 604795 | 3946/5046(78%) | 170/5046(3%) |
| 621055 to 627104 | 4678/6173(76%) | 248/6173(4%) |
| 23067 to 28363 | 4159/5433(77%) | 219/5433(4%) |
| 88200 to 93683 | 4223/5607(75%) | 243/5607(4%) |
| 17962 to 20182 | 1902/2231(85%) | 30/2231(1%) |
| 56071 to 59462 | 2717/3453(79%) | 122/3453(3%) |
| 14727 to 17919 | 2451/3226(76%) | 88/3226(2%) |
| 30013 to 32745 | 2150/2777(77%) | 84/2777(3%) |
| 20358 to 22183 | 1509/1851(82%) | 51/1851(2%) |
| 44768 to 46776 | 1619/2044(79%) | 64/2044(3%) |
| 611633 to 613671 | 1625/2086(78%) | 66/2086(3%) |
| 70970 to 73310 | 1774/2411(74%) | 102/2411(4%) |
| 83021 to 84409 | 1086/1409(77%) | 32/1409(2%) |
| 93821 to 94696 | 707/885(80%) | 18/885(2%) |
| 75528 to 76468 | 736/953(77%) | 20/953(2%) |
| 77117 to 78277 | 869/1182(74%) | 44/1182(3%) |
| 13994 to 14465 | 393/478(82%) | 9/478(1%) |
| 74833 to 75264 | 340/442(77%) | 33/442(7%) |
| 611229 to 611524 | 236/297(79%) | 2/297(0%) |
| 86174 to 86404 | 184/234(79%) | 5/234(2%) |
| Фрагмент генома | Длина | Покрытие |
| 127825 to 140555 | 9751/13010(75%) | 548/13010(4%) |
| 153752 to 161738 | 6355/8168(78%) | 264/8168(3%) |
| 144368 to 151796 | 5859/7536(78%) | 243/7536(3%) |
| 101712 to 108876 | 5567/7274(77%) | 215/7274(2%) |
| 187938 to 192665 | 3840/4801(80%) | 99/4801(2%) |
| 161898 to 166752 | 3911/4914(80%) | 112/4914(2%) |
| 166750 to 173180 | 4967/6517(76%) | 159/6517(2%) |
| 181712 to 185289 | 2778/3652(76%) | 110/3652(3%) |
| 194042 to 196061 | 1640/2070(79%) | 78/2070(3%) |
| 126623 to 127815 | 1004/1199(84%) | 11/1199(0%) |
| 192777 to 193984 | 985/1209(81%) | 4/1209(0%) |
| 196373 to 198260 | 1461/1910(76%) | 73/1910(3%) |
| 98408 to 99303 | 731/901(81%) | 9/901(0%) |
| 198467 to 199381 | 724/922(79%) | 17/922(1%) |
| 199545 to 200246 | 551/730(75%) | 52/730(7%) |
| Фрагмент генома | Длина | Покрытие |
| 266073 to 273028 | 5664/7060(80%) | 197/7060(2%) |
| 236918 to 247596 | 8178/10884(75%) | 389/10884(3%) |
| 202390 to 207661 | 4183/5329(78%) | 137/5329(2%) |
| 219625 to 223720 | 3342/4130(81%) | 61/4130(1%) |
| 224057 to 228137 | 3218/4178(77%) | 163/4178(3%) |
| 232358 to 236859 | 3468/4583(76%) | 134/4583(2%) |
| 228944 to 232057 | 2499/3165(79%) | 95/3165(3%) |
| 260224 to 263784 | 2788/3617(77%) | 101/3617(2%) |
| 248967 to 252161 | 2523/3245(78%) | 92/3245(2%) |
| 215717 to 218384 | 2145/2713(79%) | 72/2713(2%) |
| 209294 to 212243 | 2302/3007(77%) | 104/3007(3%) |
| 253223 to 257546 | 3245/4421(73%) | 195/4421(4%) |
| 208017 to 208904 | 692/902(77%) | 25/902(2%) |
| 218821 to 219491 | 515/676(76%) | 20/676(2%) |
Контиги картировались практически друг за другом, покрывая почти половину генома бактерии. Но они картировались с достаточного большим количеством разрывов. Контиги 11 и 14 оказались инвертированными по отношению к геному бактерии. Также 11 контиг наложился на точку начала последовательности генома бактерии.