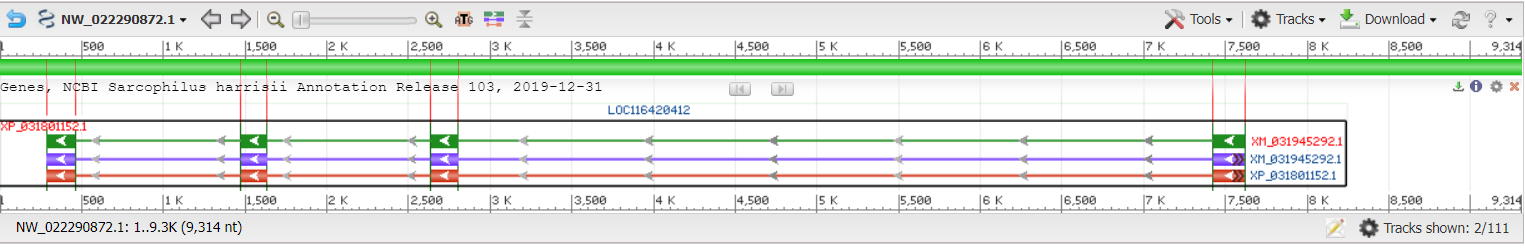
В геноме Sarcophilus harrisii я нашёл достаточно короткий контиг (NW_022290872.1, его длина: 9.314 nt), на которым есть CDS. Контиг содержит ген (LOC116420412, докализация: complement(284..7,619), длина: 7.336 nt) neurofilament medium polypeptide-like. Ген содержит CDS (XP_031801152.1, локализация: complement(284..7.619), длина: 701 nt). На рис 1 приведена схема контига и его описание.
Ссылка на последовательность контига в FASTA-формате
Затем я осуществил поиск BLAST по эукариотическим последовательностям для мой последовательности, исключив таксон Mammalia (taxid:40674) - млекопитающие.
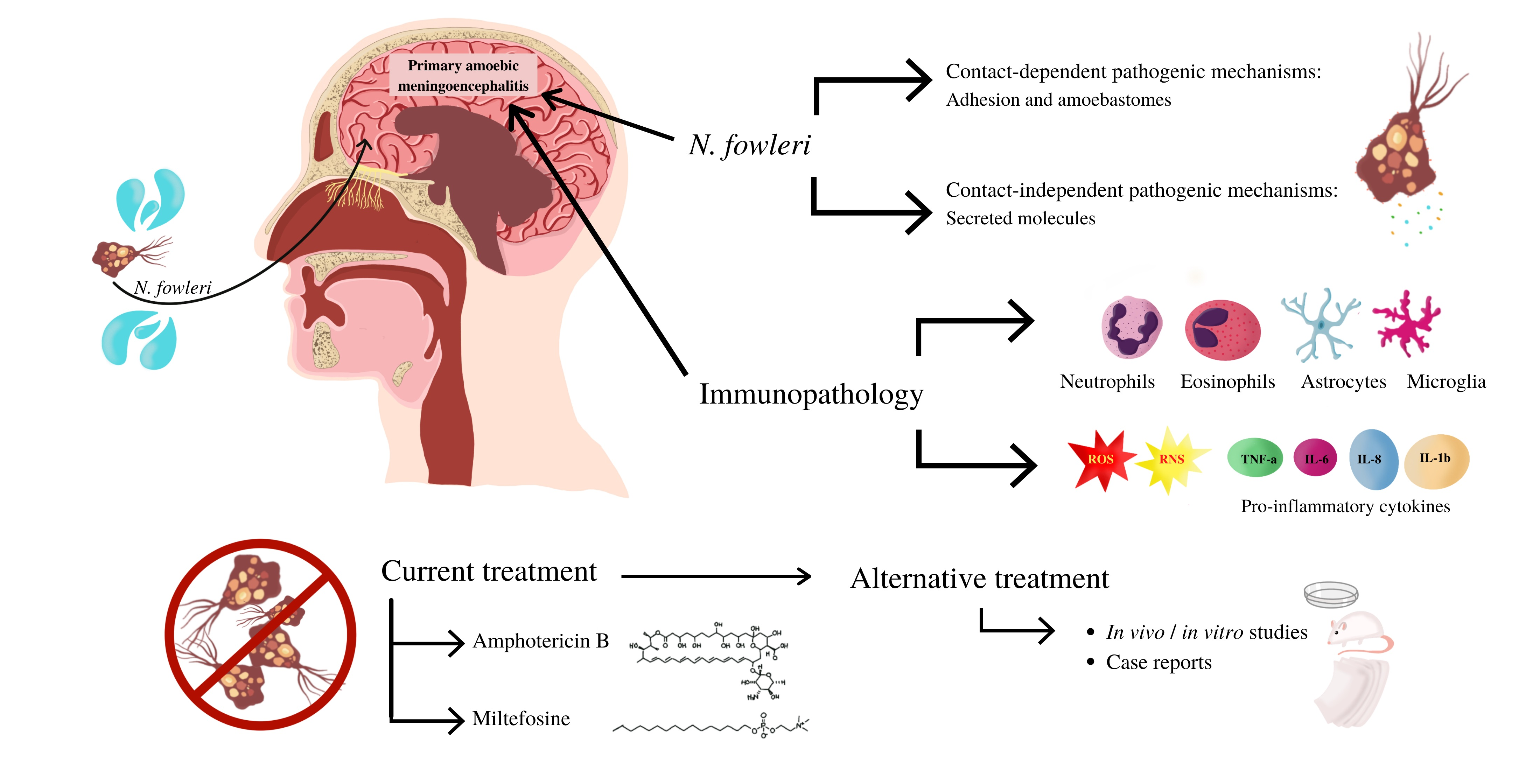
1. blastn: Найденo 131 находка. Максимальное число находок поставил 5000. Большинство это синтетические конструкции. Но также есть отдельные хромосомы других сборок. Например, Naegleria fowleri. Это чудовище вызывает первичный амёбный менингоэнцефалит. При купании оно через обонятельную луковицу и обонятельные пути проходит в мозг и приводит летальному исходу (рис 2).
2. megablast: Тут я исключил таксон ниже - marsupials (taxid:9263) - сумчатые, так как при исключении млекопитающих алгоритм ничего не выдавал. Найдено 86 находок. Максимальное число находое также 5000. Все записи являются отдельными хромосомами из других сборок. Первая в списке шла водяная полёвка (рис 3).
3. blastx: Найдено 2882 находки. Почти все они предпологаемые или неохарактеризованные белки. в начале нашёл метионинсульфоксидредуктазу B3 дикого кролика (рис 4).
4. tblastx совсем отказывается работать.
Blastn принимает на вход нуклеотидную последовательность и ищет схожие последовательности из некоторой нуклеотидной базы данных. Нужен, когда необходимо найти возможные гомологи некоторой нуклеотидной последовательности. Megablast применяется для поиска почти идентичных последовательностей (например, проверить, есть ли уже отсеквенированный геном в базе данных). Blastx принимает на вход нуклеотидную последовательность, транслирует её в белок в 6 рамках считывания и ищет в белковой базе данных схожие, гомологичные белки. Алгоритм tblastx принимает на вход нуклеотидную последовательность, транслирует её в белок в 6 рамках считывания и ищет схожие в нуклеотидной базе данных, транслированной в шести рамках считывания. Его можно использовать для того, чтобы удостовериться что искомые белки были корректно предсказаны и занесены в белковую БД.


Ещё в предыдущем практикуме я скачал геном своего организма. Теперь я сделал из него базу данных с помощью команды:
makeblastdb -in GCF_902635505.1_mSarHar1.11_genomic.fna -dbtype nucl
Далее я скачал файл с последовательностями рРНК Escherichia coli и провёл локальный поиск BLAST отдельно с 16sRNA и 23sRNA. Ниже приведены команды:
blastn -task "blastn" -query "16S_rRNA.fasta" -db "GCF_902635505.1_mSarHar1.11_genomic.fna" -out "16S_rRNA_output.tsv" -outfmt 7
blastn -task "blastn" -query "23S_rRNA.fasta" -db "GCF_902635505.1_mSarHar1.11_genomic.fna" -out "23S_rRNA_output.tsv" -outfmt 7
Выводы команд: 16S_rRNA_output.tsv и 23S_rRNA_output.tsv.
Для 16S рРНК нашлось пять находок:
Один раз легла на 2 хромосому, три раза на 4 и один раз на 1.
Для 23S рРНК нашлось 69 находок:
15 раз легла на 3, 9 раз на 2, 19 раз на 1, 10 раз на 5, 1 раз на митохондриальный геном, 1 раз на неопределённый скаффолд (NW_022290855.1), 9 раз на 4, 4 раза на 6 и 1 раз на X хромосому.
Длины выравниваний в основном коллебались в районе 50 - эта цифра не очень воодушевляет, так как эти последовательности достаточно длинные (1556 и 2932 п.н для 16S и 23S рРНК соответственно) и консервативные. Единственное, что порадовало так это единственное выравнивание 23S рРНК на митохондриальный геном, правда на 16S рРНК (Координаты на митохондриальном геноме: 1.658-3.225, длина: 1.568 п.н.). Длина этого выравнивания: 46, координаты запроса: 1067-1112, координаты выдачи: 2158-2202. Ничего примечательного в этом нет, так как выравнивание очень короткое. Я ожидал более вразумительных результатов (например, длинное выравнивание с хорошим процентом идентичности с митохондриальными 16S и 23S рРНК)
Я использовал blastn, так как на вход подаю нуклеотидную последовательность РНК и хочу найти гомологичные последовательности в нуклеотидной базе данных (сформированной из fasta-формата генома выбранного эукариота). Значения параметров не изменял.