Для выполнения практикума мне был выдан белок с идентификатором Y1337_MYCTO. Это мембранный белок Mycobacterium tuberculosis, обладающий активность эндопептидазы серинового типа.
Последовательность белка в fasta-формате: Y1337_MYCTO.fasta.
PDB-файл с предсказанной с помощью AlphaFold структурой: AF-P9WM20-F1-model_v4.pdb.
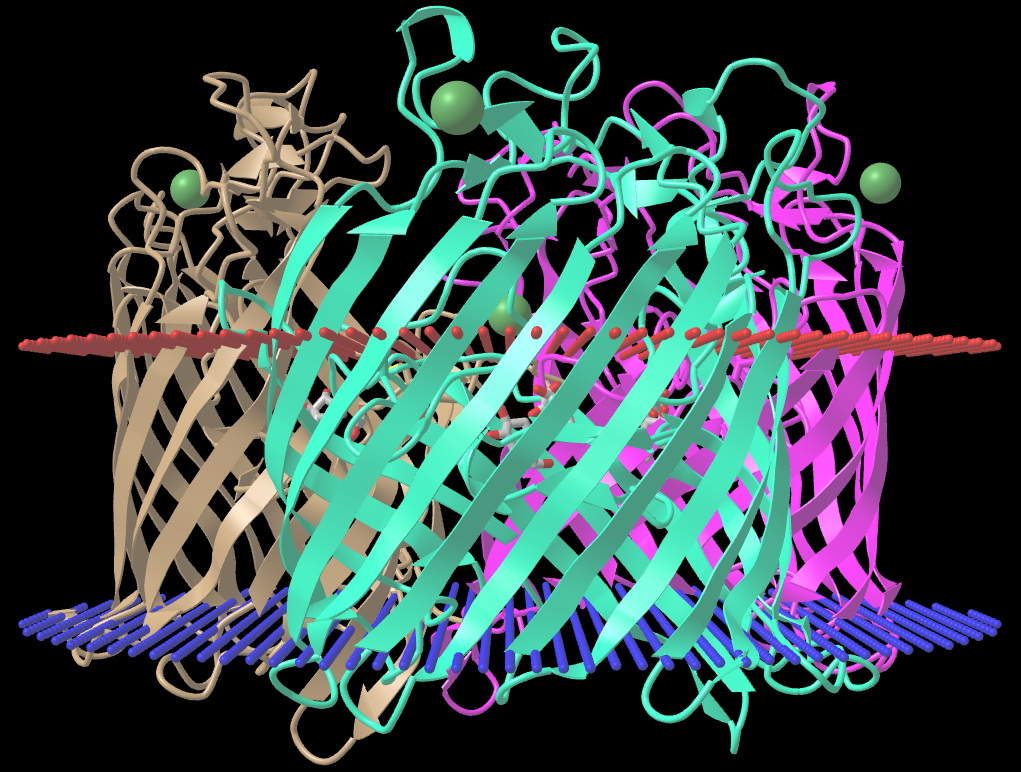
В базе данных OPM был найден белок Мальтопорин (рис. 1). Это гомотримерный белок внешней мембраны бактерий, облегчающий диффузию мальтодекстринов через внешнюю мембрану грамотрицательных бактерий. Мембранный канал образован антипараллельными листами β-бочонка. Каждая субъединица содержит 18 антипараллельных бета-листов
Идентификатор PDB: 1af6
Идентификатор Uniprot: LAMB_ECOLI

| Толщина гидрофобной части белка в мембране | Координаты трансмембранных участков (1) | Среднее количество остатков в одном β-тяже белка | Мембрана, в которую встроен белок |
| 25.1 Å | 1(2-13),2(39-48),3(58-68),4(75-88),5(98-103),6(125-132),7(138-146),8(170-179),9(185-194),10(213-221),11(227-235),12(269-278),13(284-293),14(305-314),15(320-329),16(343-352),17(361-370),18(411-420) | 10 | Внешняя мембрана Грам-отрицательной бактерии |
Текстовая выдача предсказания трансмембранных элементов альфа-спирального белка.
На рисунке 2 представлен график предсказания трансмембранных элементов белка Y1337_MYCTO: по оси ''X'' отмечены координаты - номера аминокислотных остатков, а по оси ''Y'' отмечена вероятность предсказания локализации. На небольшом горизонтальном рисунке сверху изображён профиль предсказаний: красным отмечены вероятные трансмембранные элементы, розовым - участки, располагающиеся вне мембраны с внутренней её стороны, а синим - с внешней её стороны. Можно заметить, что и N-конец, и C-конец располагаются с внутренней относительно мембраны стороны. Таким обрзом, предсказано 6 трансмембранных участков.
На рисунке 3 представлен график предсказания трансмембранных структур β-бочоночного белка LAMB_ECOLI. Оранжевым отмечен сигнал локализации. Было предсказано 18 трансмембранных бета-листа, что соответствует PDB-визуализации.
С помощью сервиса PPM, версии PPM 3.0, по pdb-файлу белка Y1337_MYCTO было получено предсказание положения данного в мембране.
Были выбраны следующие настройки алгоритма:
| Type of membrane | Allow curvature | Topology (N-ter) |
| Gram-positive bacteria inner membrane | no | in |
Тип мембраны выбран в соответствии с таксономическим положением данной бактерии. Mycobacterium tuberculosis - Грам-положительная бактерия с одной мембраной.
Предположение о локализации N-конца было взято из предсказания, полученного с помощью DeepTMHMM во втором задании.
| Толщина гидрофобной части белка в мембране | Координаты трансмембранных участков | Среднее количество остатков в одной трансмембранной альфа-спирали белка | Мембрана, в которую встроен белок |
| 31.1 ± 1.4 Å | 1( 36- 59), 2( 74- 81), 3( 92- 113), 4( 114- 134), 5( 149- 166), 6( 170- 189), 7( 201- 219) | 19 | Внутренняя мембрана Грам-положительной бактерии |
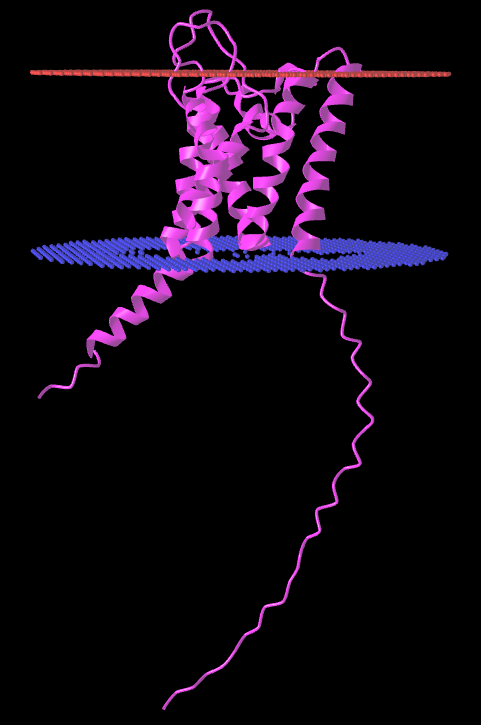
Сравним результаты двух предсказаний (DeepTMHMM и PPM) локализации белка Y1337_MYCTO:
Результаты достаточно похожи, но имеют значительные отличия. DeepTMHMM показал 6 трансмембранных альфа-спиралей, N- и C-концы имеют внутриклеточную локализацию. PPM аоказал 6 трансмембранных альфа-спиралей и одну внутримембранную альфа-спиралей, сами спирали длиннее, а между 3 и 4 спиралями нет внешних/внутренних аминокислот, хотя все эти спирали имеют очень высокую ("Very high") достоверность предсказания AlphaFold, N- и C-концы также имеют внутриклеточную локализацию. Тем не менее стоит отметить, что N-конец (вплоть до 30 аминокислотного остатка) имеет низкую (''Low''), а в паре аминокислот и очень низкую ("Very low"), достоверность в предсказании модели AlphaFold (''Model Confidence''), и это могло бы повлиять на результаты предскания PPM.