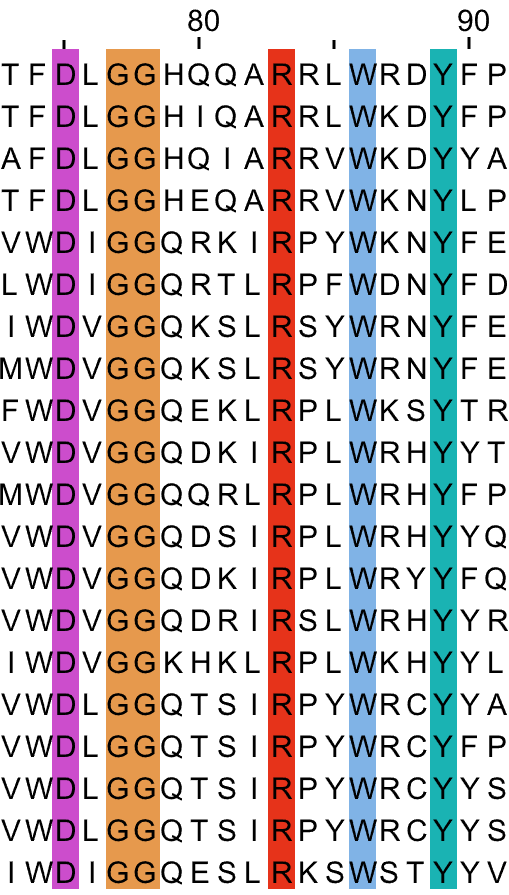
Для работы было выбрано семейство факторов рибозилирования АДФ (PF00025). Выравнивание seed для этого семейства содержит 20 последовательностей. В выравнивании на уровне 100% идентичности есть отдельные консервативные колонки и несколько мотивов. Для работы был взят второй мотив (рис. 1).
Для описания мотива был составлен следующий паттерн:
D.GG.{4}R.{2}W.{2}Y или D-x-G-G-x(4)-R-X(2)-W-x(2)-Y
С помощью функции Find в Jalview я нашёл данный паттерн во всех последовательностях по одному разу. Данный паттерн имеет высокое значение IC.
Далее я выполнил поиск по этому мотиву в базе данных SwissProt на сайте MyHits. Было найдено 206 находок. Большинство белков является белками семейства PF00025, кроме белков Guanine nucleotide-binding protein alpha, ubiquitin-protein ligase и их гомологов. Поэтому можно сказать сказать, что данный паттерн хорошо описывает белки из выбранного семейства.
На основе выравнивания было построено дерево с помощью алгоритма neighbor-joining, представленное на рисунке 2.
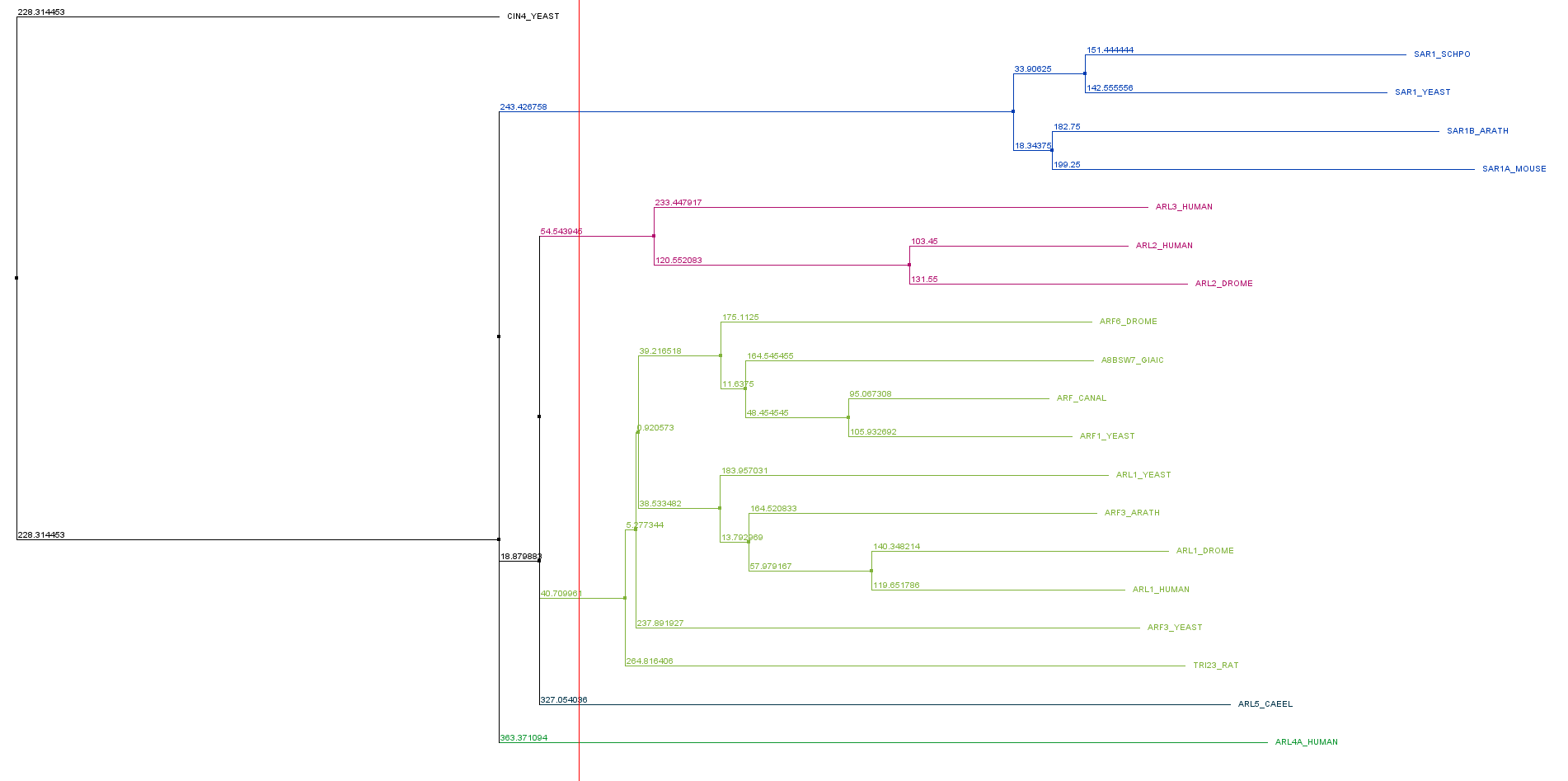
Было выделено 6 групп, три из них тривиальные. Далее я выбрал синию группу из 4 последовательностей, состоящую из SAR1 белков. В этом выравнивание был найден мотив:
PT.HPTSEEL.I
Этот мотив встречается по одному разу во всех 4 последовательностях данной клады. Это говорит о том, что мотив специфичен для данной клады.
Был выбран белок P0AD49. Во время стационарной фазы роста этот белок предотвращает образование рибосомы 70S, вероятно, для того, чтобы регулировать эффективность трансляции во время перехода между экспоненциальной и стационарной фазами роста. Таблица итераций PSI-BLAST представлена ниже.
Как видно из таблицы, после второй итерации перестало меняться число находок, удовлетворяющее порогу e-value в 0,005. Разница e-value между худшей надпороговой находки и лучшей подпороговой составляет примерно 1 порядок, что говорит о плохой обособленности надпороговых находок и малой вероятности составления из них отдельного семейства гомологичных белков.
Для проверки гипотезы был взят геном чумной палочки yersinia pestis. Число нуклеотидов: 4658550. Число слов AT: 351356. Ожидаемое число слов: 319382.6. Для проверки гипотезы использовался тест хи-квадрат. Статичтика хи-квадрата равна 1642.4283, p-value меньше 0.00001. Следовательно, мы отвергаем гипотезу о равенстве ожидаемого и наблюдаемого числа слов AT в геноме бактерии.