В базе данных Pfam был найден домен Carbohydrate binding module 77, удовлетворяющий критериям:
1. Число последовательностей в full - 96. Это больше 40, но меньше пары сотен.
2. Средняя длина домена - 108.1. Это менее 150.
3. Среднее сходство (identity) - 42%. Это более 40%.
4. Средний процент покрытия последовательности белка доменом (coverage) составляет 11.82%, то есть есть место для второго домена.
5. Число доменных архитектур - 22. Это больше двух.
Ниже показано описание домена Carbohydrate binding module 77.
| ID | AC | Число белков в seed | Число белков в full | Число белков в UniProt | Длина профиля HMM |
| CBM77 | PF18283 | 13 | 96 | 412 | 109 |
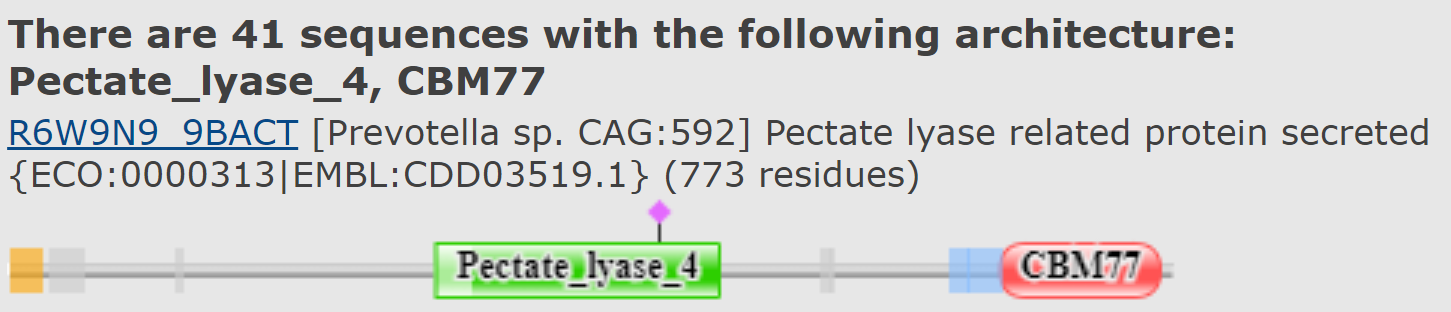
Была найдена единственная архитектура с доменами Pectate_lyase_4 и CBM77, удовлетворяющая критериям:
1. В ней есть ещё один домен, кроме выбранного. Рекомендуется взять именно двухдоменную архитектуру.
2. Встречается у 41 белка. Это более 20 и менее 58 (половины белков с выбранным доменом).
Описание и изображение архитектуры (Рис. 1) показано ниже:
| ID 2 домена | AC 2 домена | Число белков с архитектурой | Длина профиля HMM |
| CBM77 | PF18283 | 31 | 372 |
Далее скачиваем последовательности нужного семейства (prot_96.fasta). Затем удаляем дубликаты (prot_93.fasta). Теперь копируем из Pfam АС белков одной архитектуры и создаём файл с последовательностями этих белков (prot_41.fasta). Команды приведены в colab. Последовательности с нужной архитектурой были выровнены в Jalview с помощью Muscle с параметрами по умолчанию (prot_41_align.fasta). После этого была произведена ревизия полученного выравнивания: удаление N-концевого (до 284 аминокислоты) и C-концевого (после 767 аминокислоты) участков по координатам последовательности R6W9N9_9BACT (координаты первого домена (Pectate_lyase_4) - 284:472, а второго (CBM77) - 661:767), исключение из выравнивания некоторых наиболее отличающихся последовательностей. Также была проведена процедура удаление избыточных последовательностей на уровне идентичности 99-100%. В итоге получилось выравнивание (prot_31_align_red.fasta), состоящее из 31 последовательности.
Построим HMM-профиль нашей двух-доменной архитектуры с помощью hmm2build:
hmm2build HMM_profile new_aln.fa
Откалибруем (нормируем вес):
hmm2calibrate HMM_profile
Проведём поиск профиля по последовательностям из prot_96.fasta, файл hmm.txt используем для таблицы:
hmm2search -E 0.1 --cpu=1 HMM_profile full.fasta > hmm.txt
Таблица и графики показаны в Google-таблице.
Далее была построена гистограмма распределения длин белков, входящих в выбранное CBM77 (Рис. 2). Можно сделать вывод, что для белков выбранного семейства характерна длина в диапазоне 555 - 833. Затем был построен график падения весов последовательностей (Рис. 3). При этом красной линеей отмечен порог, полученный с помощью RОС-кривой (Рис. 4): для этого измерим наибольшее расстояние от ROC-кривой до диагонали, соединяющей начало и конец. Здесь эта точка, примерно, с координатами (-0.865384615;0.853658537) и весом 552,9. График F1 (рис.5): при миниальных значениях чувствительности линия плавная, затем растёт, достигает пика и убывает; локальный минимум в точке (618.90;0.820512821).