Задание 1. Предсказание вторичной структуры заданной тРНК
Нужная последовательность тРНК была скачена с
сайта PDB в формате fasta(после в скачанном файле была удалена аминокислотная последовательность и I
внутри последовательности был заменен на G).
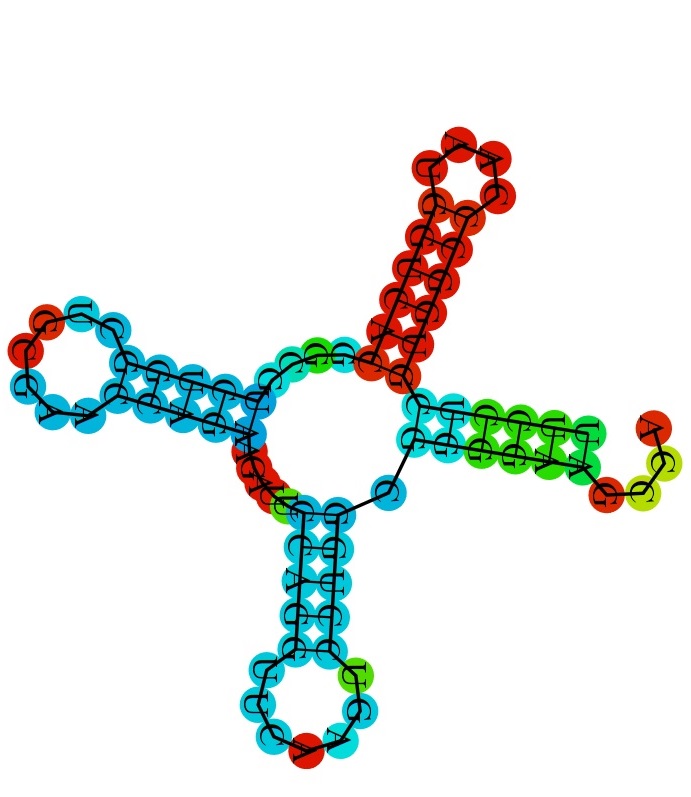
Привер типичной структуры тРНК:
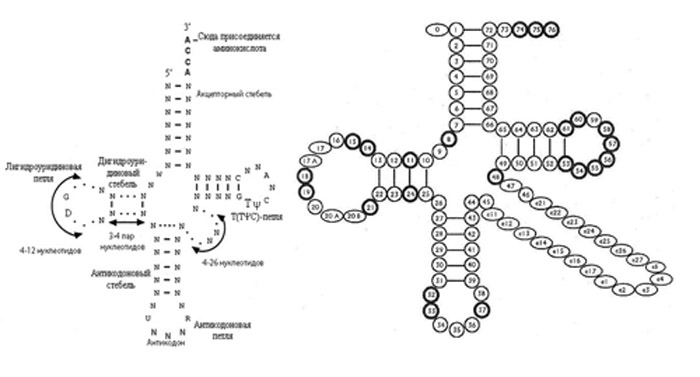
Само предсказание вторичной структуры:
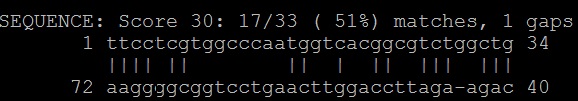
º Einverted
При стандартных значениях программа выдает пустой файл.
Что получается, если изменить параметры:
Gap penalty [12]: 5
Minimum score threshold [50]: 20
Match score [3]: 3
Mismatch score [-4]: -1
º Алгоритм Зукера
Для выполнения этого упражнения я воспользовалась
онлайн версией RNAfold.
Изменяя параметры, вот чего мне удалось добиться:
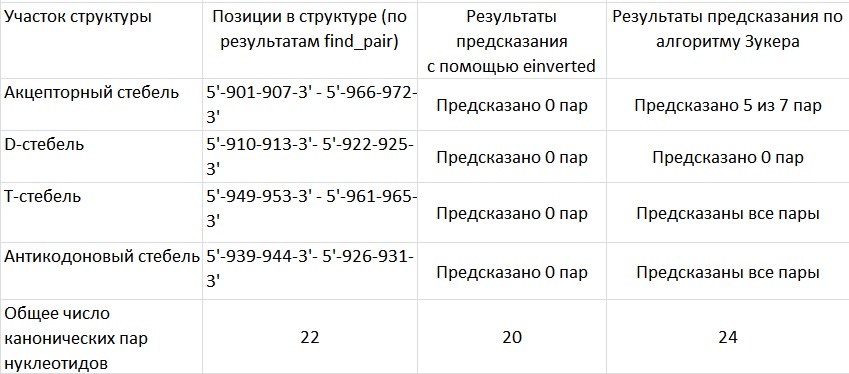
Таблица 1. Реальная и предсказанная вторичная структура тРНК из файла
1f7v.pdb Задание 2. Поиск ДНК-белковых контактов в заданной структуре
Переключайте кнопки "Start1" и "Start2" после нажатия на апплет выше.
После этого можете переключать между изображениями при помощи кнопки "Resume".
Таблица 2. Контакты разного типа в комплексе
1rio.pdb
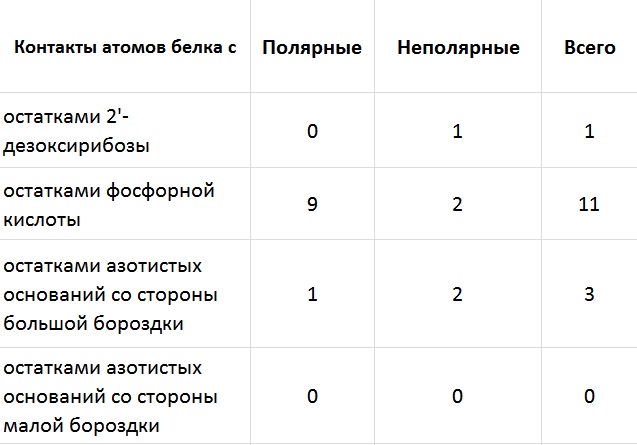
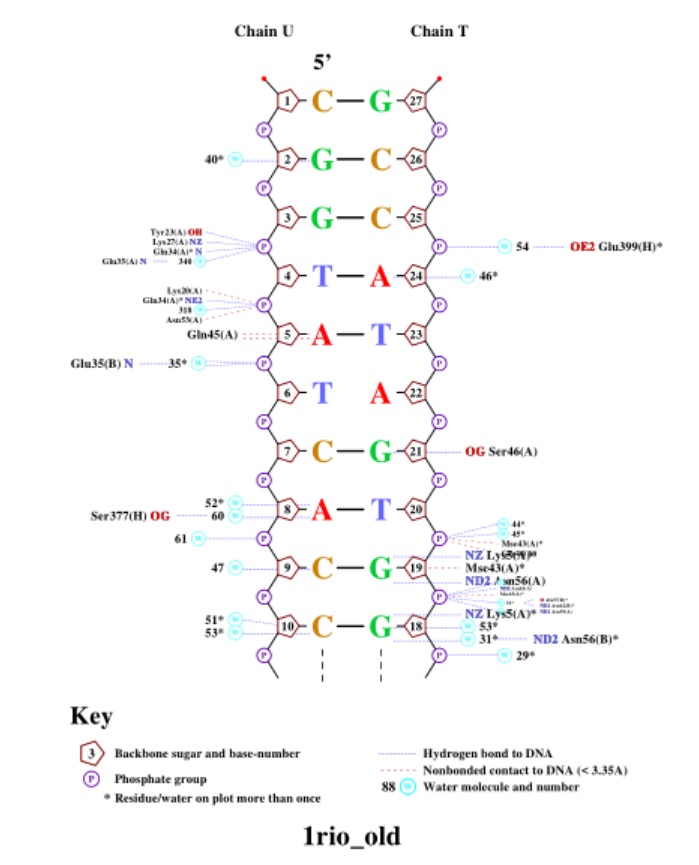
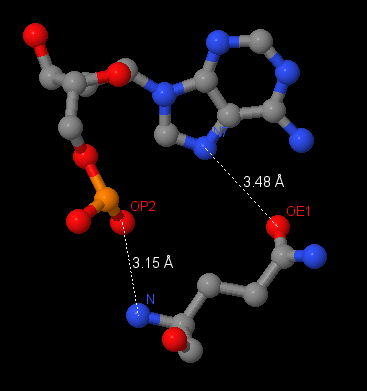
Будем считать полярными атомы кислорода и азота, а неполярными атомы углерода, фосфора и серы.
Мы видим, что больше всего контактов белок образовал с фосфатами остова. Также можно заметить, что он не взаимодействовал с малой бороздкой ДНК.
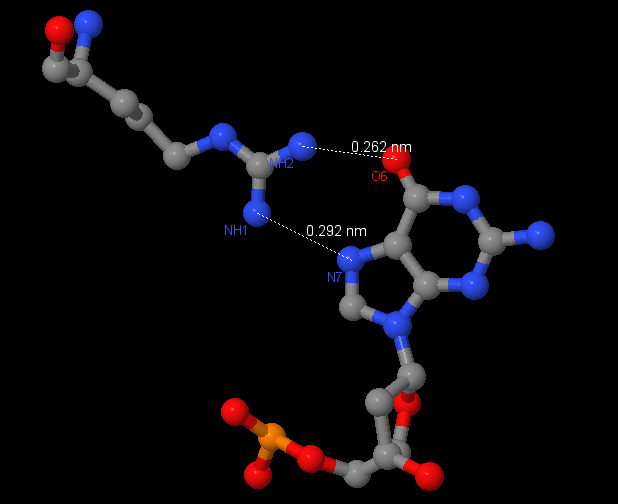
Аминокислотный остаток с наибольшим числом указанных на схеме контактов с ДНК - Arg74, ибо он образует контакты с NH2 в двух местах и c NH1 - в одном.
Аминокислотный остаток, наиболее важный для распознавания последовательности ДНК - Gln76 (связывается с самим азотистым основанием, хорошо ложится в бороздку ДНК).