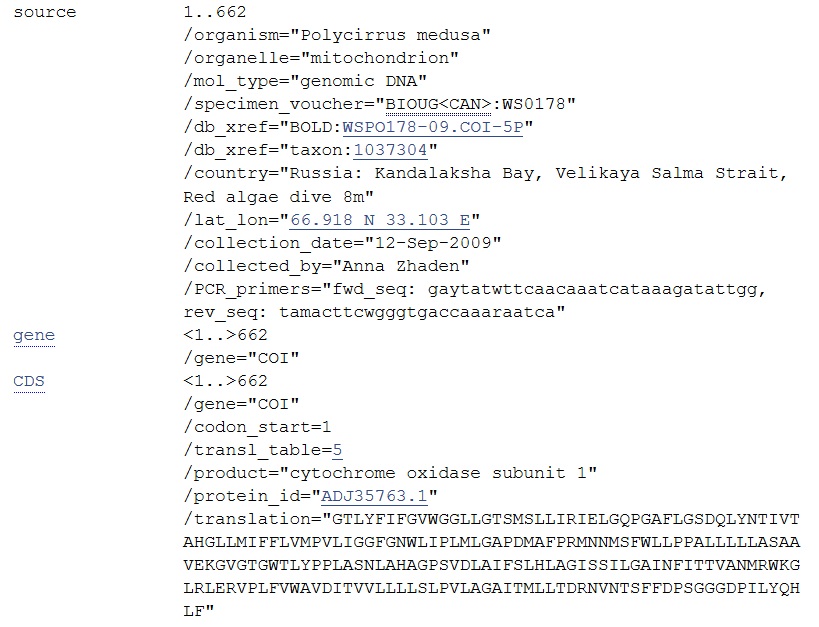
Задание 1. Определить таксономию и функцию прочтенной нуклеотидной последовательности
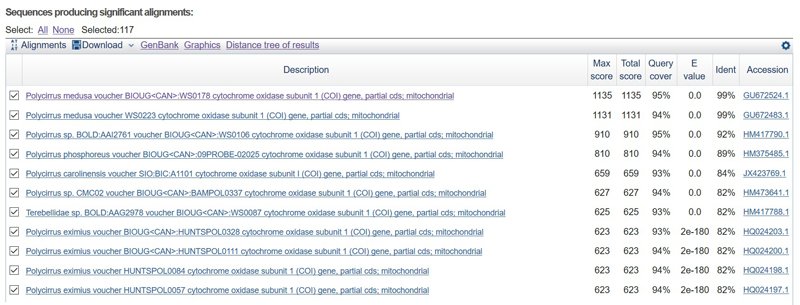
С помощью BLASTN было установлено к какому гену принадлежит эта последовательность, а так же определена таксономия организма. Из находок в BLAST>Nucleotide я выбрала 3 лучшие: Query cover выше 90%, E-value равно 0 и процент идентичных позиций превышает 90%.
 Последовательности во всех трёх находках представляют собой один и тот же ген, кодирующий субъединицу 1 митохондриального фермента цитохромоксидазу (COI).
Первые две находки - из организма Polycirrus medusa, третья находка - из организма Polycirrus sp. BOLD:AAI2761 .
Оба организма принадлежат к одному роду многощетинковых кольчатых червей - Polycirrus. [ссылка на источник]
Последовательности во всех трёх находках представляют собой один и тот же ген, кодирующий субъединицу 1 митохондриального фермента цитохромоксидазу (COI).
Первые две находки - из организма Polycirrus medusa, третья находка - из организма Polycirrus sp. BOLD:AAI2761 .
Оба организма принадлежат к одному роду многощетинковых кольчатых червей - Polycirrus. [ссылка на источник]
 Полученные три файла найденных в BLAST последовательностей в формате fasta были загружены для множественного выравнивания в программу Jalview.
° Polycirrus medusa voucher WS0178 [fasta-файл]
° Polycirrus medusa voucher WS0223 [fasta-файл]
° Polycirrus sp. BOLD%3AAAI2761 [fasta-файл]
По предоставленным ниже данным мы видим, что наша изучаемая последовательность почти не отличается от Polycirrus medusa voucher WS0178 и
Polycirrus medusa voucher WS0223. Поэтому мы можем сказать, что изучаемая последовательность так же является геном,
который кодирует первую субъединицу цитохромоксидазы полихет из рода Polycirrus.
Полученные три файла найденных в BLAST последовательностей в формате fasta были загружены для множественного выравнивания в программу Jalview.
° Polycirrus medusa voucher WS0178 [fasta-файл]
° Polycirrus medusa voucher WS0223 [fasta-файл]
° Polycirrus sp. BOLD%3AAAI2761 [fasta-файл]
По предоставленным ниже данным мы видим, что наша изучаемая последовательность почти не отличается от Polycirrus medusa voucher WS0178 и
Polycirrus medusa voucher WS0223. Поэтому мы можем сказать, что изучаемая последовательность так же является геном,
который кодирует первую субъединицу цитохромоксидазы полихет из рода Polycirrus.
Задание 2. Сравните списки находок нуклеотидной последовательности 3-я разными алгоритмами blast
° Highly similar sequences (megablast)
Область поиска была ограничена семейством, к которому принадлежала лучшая находка - Terebellidae (taxid:32261)(так как до этого число находок было неприемлемо),
Max target sequences был установлен на 1000.

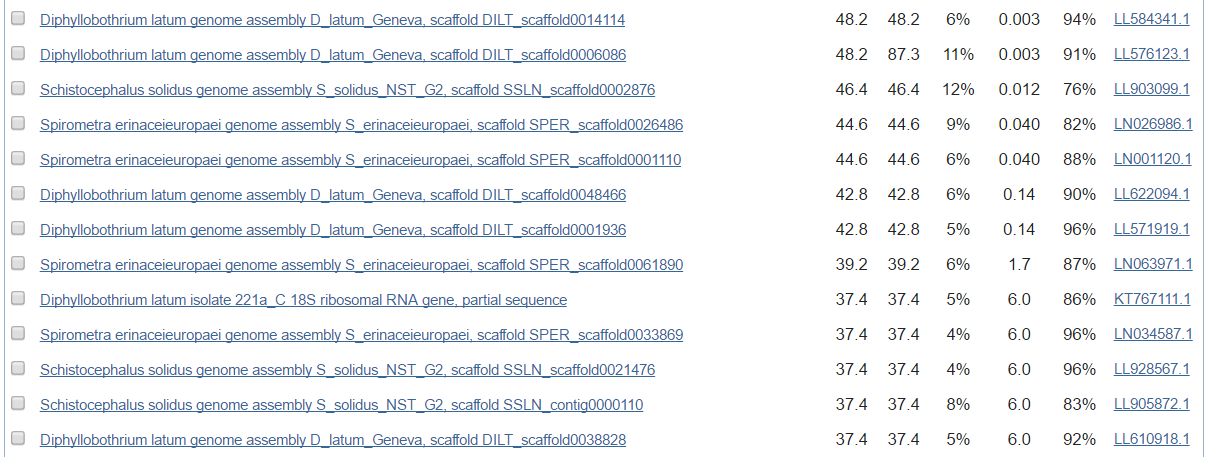
° More dissimilar sequences (discontiguous megablast)
Было получено 117 находок.
° Somewhat similar sequences (blastn)
С учетом прежних настроек было получено 115 находок.
 По сравнению с blastn discontiguous megablast нашёл ещё две находки. Было замечено, что у этих находок query cover горздо меньше по сравнению
с остальными.
Это удивительно, так как blastn ищет хоть что-нибудь сходное, и должен найти больше всех находок в сравнении с другими алгоритмами.
По сравнению с blastn discontiguous megablast нашёл ещё две находки. Было замечено, что у этих находок query cover горздо меньше по сравнению
с остальными.
Это удивительно, так как blastn ищет хоть что-нибудь сходное, и должен найти больше всех находок в сравнении с другими алгоритмами.
Задание 3.
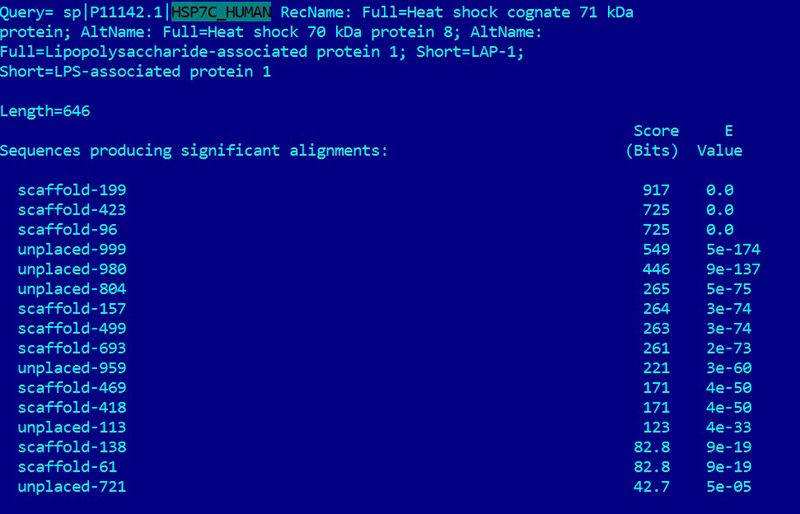 Лучшая находка имеет следующие параметры:
> scaffold-199
Length=1112851
Score = 917 bits (2369), Expect = 0.0, Identities = 474/607 (78%), Positives = 538/607 (89%), Gaps = 0/607 (0%), Frame = -2
Всего находок - 16.
У находки хороший E-value, достаточно высокие проценты Positives и Identities.
Параметры сходства достаточны, чтобы назвать ее гомологом исследуемого белка, вероятно выполняющим ту же функцию.
° TERT_HUMAN
Лучшая находка имеет следующие параметры:
> scaffold-199
Length=1112851
Score = 917 bits (2369), Expect = 0.0, Identities = 474/607 (78%), Positives = 538/607 (89%), Gaps = 0/607 (0%), Frame = -2
Всего находок - 16.
У находки хороший E-value, достаточно высокие проценты Positives и Identities.
Параметры сходства достаточны, чтобы назвать ее гомологом исследуемого белка, вероятно выполняющим ту же функцию.
° TERT_HUMAN
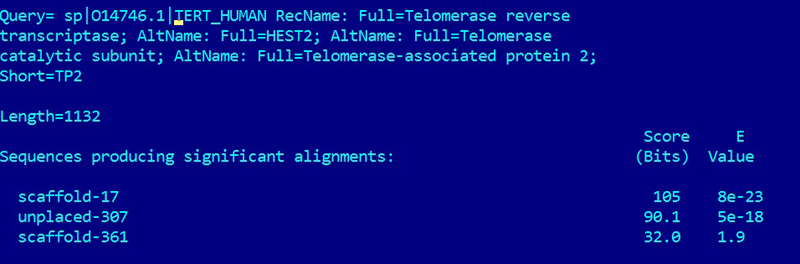 Лучшая находка имеет следующие параметры:
> scaffold-17
Length=2125590
Score = 105 bits (263), Expect = 8e-23, Identities = 151/568 (27%), Positives = 248/568 (44%), Gaps = 43/568 (8%), Frame = +1
Этот результат является условно положительным. BLAST выдал три находки, однако даже у самой лучшей из них параметры сходства слишком низкие,
чтобы утверждать сохранение функций. Гомология отдельных доменов также маловероятна, так как совпадения распределены по всей длине
последовательности относительно равномерно.
° CISY_HUMAN
Лучшая находка имеет следующие параметры:
> scaffold-17
Length=2125590
Score = 105 bits (263), Expect = 8e-23, Identities = 151/568 (27%), Positives = 248/568 (44%), Gaps = 43/568 (8%), Frame = +1
Этот результат является условно положительным. BLAST выдал три находки, однако даже у самой лучшей из них параметры сходства слишком низкие,
чтобы утверждать сохранение функций. Гомология отдельных доменов также маловероятна, так как совпадения распределены по всей длине
последовательности относительно равномерно.
° CISY_HUMAN
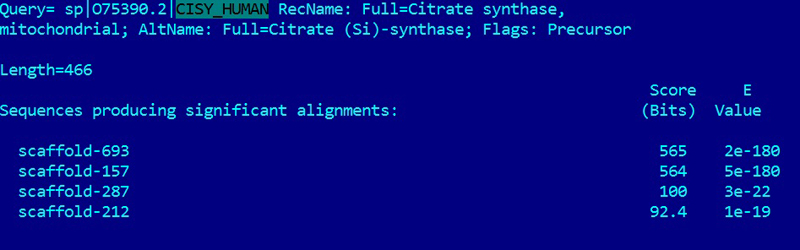 Лучшая находка имеет следующие параметры:
> scaffold-693
Length=1268102
Score = 565 bits (1457), Expect = 2e-180, Identities = 262/377 (69%), Positives = 315/377 (84%), Gaps = 3/377 (1%) Frame = +1
Всего находок - 4.
Наблюдаются относительно хорошие параметры сходства. Скорее всего, данный белок является гомологом изучаемого нами белка.
Лучшая находка имеет следующие параметры:
> scaffold-693
Length=1268102
Score = 565 bits (1457), Expect = 2e-180, Identities = 262/377 (69%), Positives = 315/377 (84%), Gaps = 3/377 (1%) Frame = +1
Всего находок - 4.
Наблюдаются относительно хорошие параметры сходства. Скорее всего, данный белок является гомологом изучаемого нами белка.