Для работы был выдан штамм бактерии Shewanella frigidimarina. При поиске в базе
Uniprot по заданным критериям и "Shewanella frigidimarina [56812]" не находилось ни одной
аннотированной записи. Поэтому я выбрала предложенную системой "Shewanella frigidimarina (strain NCIMB 400) [318167]"
И тогда в банке Uniprot было найдено 8 аннотированных записей.
Мнемоника выбранного штамма: SHEFN.
| Entry |
Entry name |
Protein name |
Gene name |
Upstream coordinates |
| Q07YK6 |
PURT_SHEFN |
Formate-dependent phosphoribosylglycinamide formyltransferase |
purT (Sfri_3070) |
3676745..3677935 |
| Q085S1 |
PUR4_SHEFN |
Phosphoribosylformylglycinamidine synthase, FGAM synthase, FGAMS, EC 6.3.5.3 |
purL (Sfri_1141) |
1343377..1347258 |
| Q07XS1 |
PURA_SHEFN |
Adenylosuccinate synthetase, AMPSase, AdSS, EC 6.3.4.4 |
purA (Sfri_3357) |
complement(4006494..4007789) |
| Q07ZX6 |
FOLD_SHEFN |
Bifunctional protein FolD [Includes: Methylenetetrahydrofolate dehydrogenase, EC 1.5.1.5; Methenyltetrahydrofolate cyclohydrolase, EC 3.5.4.9] |
folD (Sfri_2598) |
3108228..3109082 |
| Q085S4 |
GUAA_SHEFN |
GMP synthase [glutamine-hydrolyzing], EC 6.3.5.2 |
guaA (Sfri_1138) |
1339029..1340606 |
| Q084L1 |
PUR5_SHEFN |
Phosphoribosylformylglycinamidine cyclo-ligase, EC 6.3.3.1 |
purM (Sfri_1452) |
complement(1736045..1737082) |
| Q088G0 |
PUR9_SHEFN |
Bifunctional purine biosynthesis protein PurH [Includes: Phosphoribosylaminoimidazolecarboxamide formyltransferase, EC 2.1.2.3 (AICAR transformylase) ; IMP cyclohydrolase, EC 3.5.4.10 (ATIC) (IMP synthase) (Inosinicase) ] |
purH (Sfri_0493) |
complement(559676..561265) |
| Q07WS1 |
PUR7_SHEFN |
Phosphoribosylaminoimidazole-succinocarboxamide synthase, EC 6.3.2.6 |
purC (Sfri_3716) |
4410026..4411129 |
При помощи descseq были вырезаны Upstream-участки длиной 100 нуклеотидов для каждого гена. Они собраны в
fasta-файл.
Далее в вырезанных последовательностях производился поиск мотивов с помощью программы MEME, которая установлена на сервере kodomo. Для этого была
использована команда "ememe -dataset all_genes_pr5.fasta -outdir SHEFN_motiv -revcomp -nmotifs 3".
Результаты вы можете найти
[здесь].
[Ссылка в формате html-страницы].
Мотивы
Мотив 1
Рисунок 1. Мотив 1
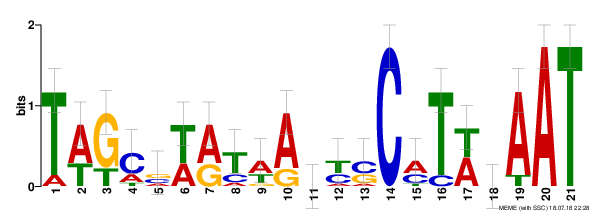
Таблица 2. Данные по мотиву 1
| Наименование величины |
Значение |
| Регуляторное выражение |
T[AT][GT][CA][CGA][TA][AG][TC][AT][AG][CT][TC][CG]C[AC]T[AT][AT]AAT |
| Длина нуклеотидов |
21 |
| E-value |
1.4e+001 |
| Встречаемость мотива |
8/8 (E-value порядка e-8) |
Мотив 2
Рисунок 2. Мотив 2
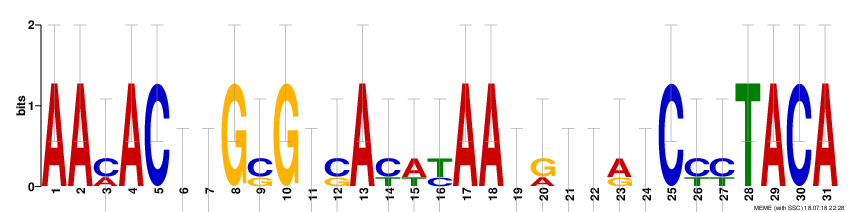
Таблица 3. Данные по мотиву 2
| Наименование величины |
Значение |
| Регуляторное выражение |
AA[CA]AC[ACT][ACT]G[CG]G[CGT][CG]A[CT][AT][TC]AA[ACG][GA][AGT][ACT][AG][ACT]C[CT][CT]TACA |
| Длина нуклеотидов |
31 |
| E-value |
7.6e+003 |
| Встречаемость мотива |
3/8 (E-value порядка e-14) |
Мотив 3
Рисунок 3. Мотив 3
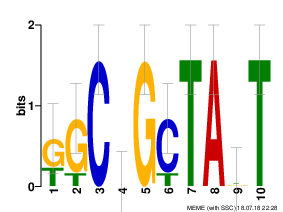
Таблица 4. Данные по мотиву 3
| Наименование величины |
Значение |
| Регуляторное выражение |
[GT][GT]C[CAGTA]G[CT]TA[GTA]T |
| Длина нуклеотидов |
10 |
| E-value |
1.2e+004 |
| Встречаемость мотива |
5/8 (E-value порядка e-5) |
Как можно видеть, ни одного хорошего мотива (E-value < 0,001) найдено не было.
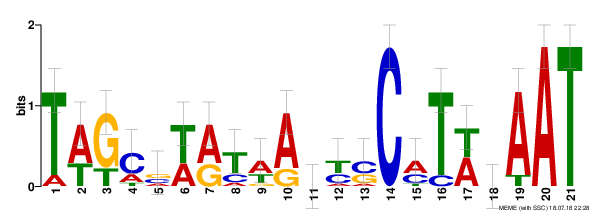 Таблица 2. Данные по мотиву 1
Таблица 2. Данные по мотиву 1
 Таблица 3. Данные по мотиву 2
Таблица 3. Данные по мотиву 2
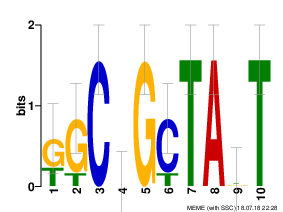 Таблица 4. Данные по мотиву 3
Таблица 4. Данные по мотиву 3
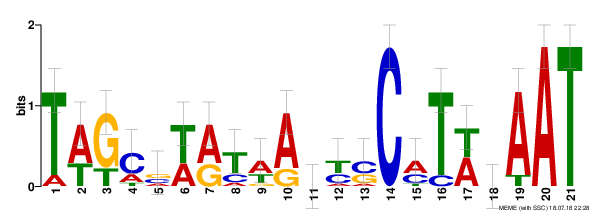 Таблица 2. Данные по мотиву 1
Таблица 2. Данные по мотиву 1
 Таблица 3. Данные по мотиву 2
Таблица 3. Данные по мотиву 2
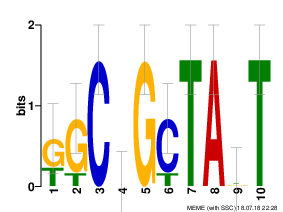 Таблица 4. Данные по мотиву 3
Таблица 4. Данные по мотиву 3
