Вторичная структура нуклеиновых кислот
Известно, что РНК образует великое множество мотивов вторичной структуры, да и ДНК не ограничивается одной формой B-спирали. Практикум 2 посвящён анализу строения различных форм нуклеиновых кислот, как полученных экспериментально, так и сгенерированных при помощи пакета 3DNA.
Генерация спирали ДНК
Команда fiber пакета 3DNA позволяет моделировать разнообразные спирали нуклеиновых кислот в формате pdb.
Команда fiber -m позволяет получить список доступных для моделирования структур. К сожалению, Z-ДНК с произвольным нуклеотидным составом получить нельзя (есть всего два варианта — поли-GC и поли-As4T-спираль). Вот какие команды были исполнены для генерирования файлов:
- fiber -seq=GATCGATCGATCGATCGATC -a gatc-a.pdb
- fiber -seq=GATCGATCGATCGATCGATC -b gatc-b.pdb
- fiber -z gatc-z.pdb
Важно отметить, что по умолчанию генерируется поли-GC-Z-спираль из 20 пар оснований.
Полученные модели можно скачать.
Сравнение одной из моделей с экспериментальными данными. Изучение трёх форм ДНК
Для сравнения я выбрал A-форму ДНК (PDB ID экспериментального файла 3V9D).
Названия «большая бороздка» и «малая бороздка» в случае A-спирали несколько контринтуитивны: корректнее было бы выразиться «глубокая» и «мелкая», не называя большой бороздкой более узкую. На рис. 1 показан тимин, находящийся на положении 7 цепи B экспериментальной модели. Изображение получено при помощи MarvinSketch.
- В сторону большой бороздки обращены атомы T7.C4, T7.O4, T7.C5, T7.C6, T7.C7.
- В сторону малой бороздки обращены атомы T7.N1, T7.C2, T7.O2, T7.N3.
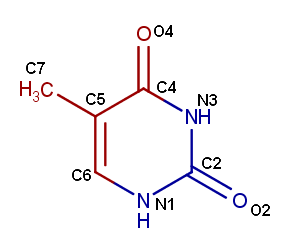
Красным показаны атомы, направленные в сторону большой (глубокой) бороздки, синим — в сторону малой.
Далее были проанализированы ключевые особенности спиралей ДНК, сгенерированных 3DNA, с помощью визуализации и измерений в JMol. Нельзя не отметить, что число оснований на виток (и шаг спирали в ангстремах) данным методом можно измерить только с точностью до целого числа мономеров. Итоги измерений изложены в табл. 1.
| A-форма | B-форма | Z-форма | |
|---|---|---|---|
| Тип спирали | правая | правая | левая |
| Шаг спирали, Å | 28,03 | 33,75 | 42,34 |
| Число оснований на виток | 11 пар | 10 пар | 12 пар |
| Ширина большой бороздки, Å | 7,98 (C8:A—T27:B) |
17,21 (A10:A—C28:B) |
7,20 (G13:A—G31:B) |
| Ширина малой бороздки, Å | 16,81 (C8:A—T35:B) |
11,69 (A10:A—T35:B) |
18,30 (C8:A—C30:B) |
Ширина бороздки должна была равняться локальному минимуму расстояний между фосфатами разноимённых цепей, и в случае Z-спирали не всегда можно было измерить их от одного атома фосфора (в данном случае пришлось выбирать разные: расстояние от фосфатов второй цепи до первого атома убывало вплоть до точки, где было произведено измерение другой бороздки).
Структура РНК
Определяем параметры структуры РНК с помощью программ find_pair и analyze пакета 3DNA.
Выданная мне структура имеет идентификатор PDB 1I9V. Я скачал соответствующий файл с сайта RCSB и преобразовал его для совместимости с 3DNA:
remediator --old ''1i9v.pdb'' > ''1i9v_old.pdb''
Вначале были определены торсионные углы нуклеотидов в структуре. Запускаем конвейер:
find_pair -t 1i9v_old.pdb stdout | analyze
Торсионные углы ищем в файле 1i9v_old.out. Для сравнения с ДНК посмотрим на наиболее характерные отличия в значениях для A- и B-спиралей (углы δ, ε, ζ, χ). На рис. 2 показано, как выглядит таблица торсионных углов. Сравнивать будем со значениями в презентации.
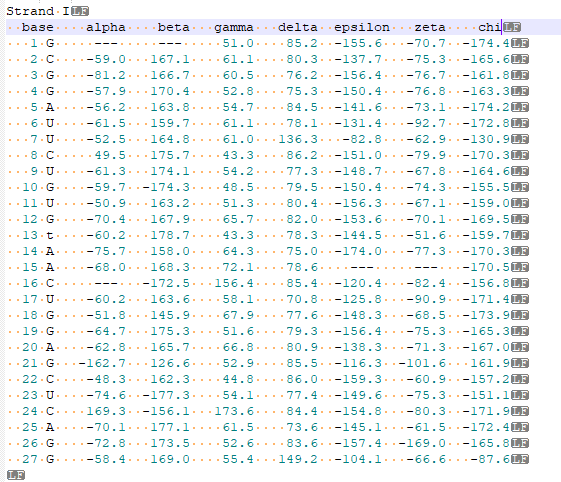
Перечислим, к каким значениям ближе получаются углы.
- δ ближе к значинию A-ДНК;
- ε ближе к B-ДНК;
- ζ занимает некое промежуточное положение;
- χ ближе к A-ДНК.
Обобщая, вспомнив, как выглядит стебель РНК при визуализации, а также держа в уме раздел файла Classification of each dinucleotide step in a right-handed nucleic acid structure, где встречаются только упоминания A, можно утверждать, что тяжи РНК похожи на A-форму ДНК.
Теперь определяем структуру водородных связей между основаниями. Рис. 3 показывает, как выглядит абзац, где указаны основные параметры водородных связей в парах оснований.
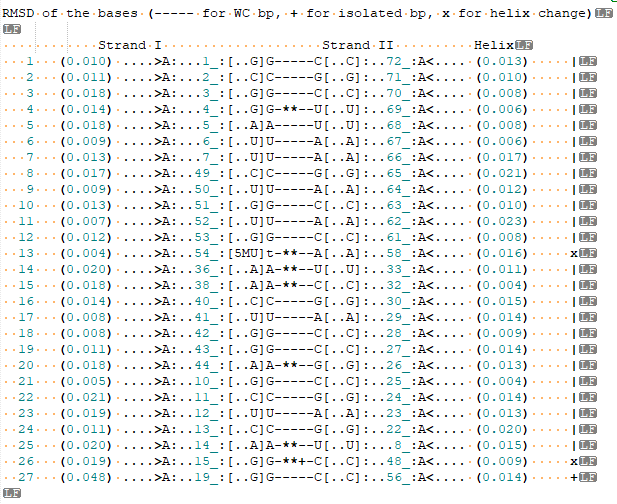
Видно, что структура содержит 7 неканонических пар:
- 4G..69U
- 54t..58A
- 36A..33U
- 38A..32C
- 44A..26G
- 14A..8U
- 15G..48C
Координаты стеблей (считаем хотя бы две подряд идущие пары оснований, неканоническое спаривание не учитываем на концах стебля):
- 1..7—72..66
- 49..53—65..61
- 40..43—30..27
- 10..13—25..22
Остальные 7 комплементарных пар, среди которых одна уотсон-криковская, не входят в стебли и служат для дополнительной стабилизации структуры.
Итак, здесь были смоделированы и изучены 3 вида спиралей ДНК, а также изучена вторичная структура РНК средствами пакета 3DNA.