Предсказание вторичной структуры РНК. Комплексы ДНК-белок
Одно задание посвящено предсказанию вторичной структуры РНК, а второе — ДНК-белковым контактам.
Предсказание вторичной структуры РНК
Здесь мы используем различные программы (einverted, RNAfold) для предсказания вторичной структуры РНК и сравниваем их результат с описанием, составленным программой find_pair по модели PDB.
Последовательность РНК для einverted была получена со страницы NDB, посвящённой молекуле (ссылка туда вела с RCSB). — Файл был назван tr0004.fasta, для корректной обработки RNAfold нестандартные нуклеотиды в скобках были изменены: (5MU) на U, (YG) — на X. Итоговые данные получены исполнением следующих команд (неуказанные параметры взяты со значениями по умолчанию):
- einverted tr0004.fasta -gap 20 -thr 20 -match 6 -mis -8
- export PATH=${PATH}:/home/preps/golovin/progs/bin
- cat tr0004.fasta | RNAfold --MEA -p > zuker.rnf
- ps2pdf *_ss.ps rnf_ss.pdf
- ps2pdf *_dp.ps rnf_dp.pdf
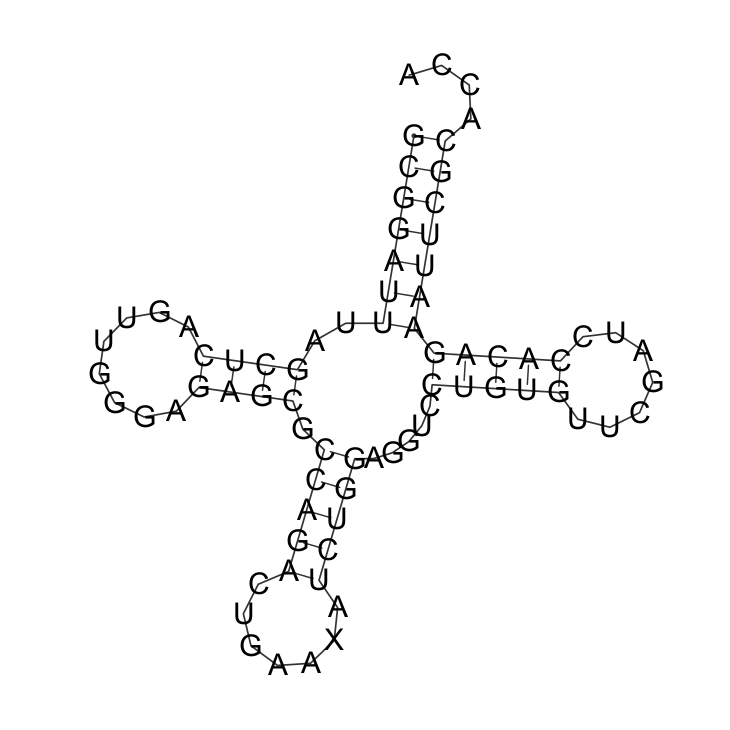
RNAfold удовлетворительно нашла все канонические взаимодействия в стеблях с первого запуска, а спарить неканонические 26 и 44 нуклеотиды не удавалось даже при использовании ключа --nsp -AG, технически это позволяющего — видимо, их действительно не стоит включать в стебель.
В таблице 1 приведено сравнение результатов работы трёх программ.
| Участок структуры | Позиции в структуре (find_pair) | Результаты предсказания с помощью einverted | Результаты предсказания по алгоритму Зукера (RNAfold) |
|---|---|---|---|
| Акцепторный стебель | 5'-1-7-3' 5'-66-72-3' Всего 7 пар Канонических 6 |
— | Предсказано 7 пар из 7 (6) реальных |
| D-стебель | 5'-10-13-3' 5'-22-25-3' Всего 4 пары |
— | Предсказано 4 пары из 4 реальных |
| T-стебель | 5'-49-53-3' 5'-61-65-3' Всего 5 пар |
Предсказано 5 пар из 5 реальных |
Предсказано 5 пар из 5 реальных |
| Антикодоновый стебель | 5'-27-30-3' 5'-40-43-3' Всего 4 пары |
Предсказано 4 пары из 4 реальных, а также 4 лишних |
Предсказано 4 пары из 4 реальных, а также 1 лишняя |
| Общее число канонических пар нуклеотидов |
19 | 9 предсказано | 19 предсказано |
Рис. 1 иллюстрирует результат работы алгоритма Зукера.
ДНК-белковые контакты
Здесь исследуются контакты нуклеиновой кислоты и белка в PDB модели 1MDM.
Вначале был написан скрипт, выделяющий в JMol атомы кислорода 2'-дезоксирибозы, остатков фосфорной кислоты и атомы азота остатков азотистых оснований. Потом — сценарий, показывающий по порядку все атомы, проволочную модель ДНК и выделяющий атомы вышеупомянутых множеств.
Для определения числа контактов был использован усовершенствованный скрипт и несколько команд select, по выполнении которых консоль отчитывается о числе атомов:
- select *.?*' and polaratom and within(3.5, polaratom and protein)
- select *.?*' and nonpolaratom and within(4.5, nonpolaratom and protein)
- select pacid and polaratom and within(3.5, polaratom and protein)
- select pacid and nonpolaratom and within(4.5, nonpolaratom and protein)
- select mingr and polaratom and within(3.5, polaratom and protein)
- select mingr and nonpolaratom and within(4.5, nonpolaratom and protein)
- select majgr and polaratom and within(3.5, polaratom and protein)
- select majgr and nonpolaratom and within(4.5, nonpolaratom and protein)
Данные о контактах сведены в таблицу 2.
| Контакты атомов белка с | Полярные | Неполярные | Всего |
|---|---|---|---|
| остатками 2'-дезоксирибозы | 3 | 13 | 16 |
| остатками фосфорной кислоты | 9 | 6 | 15 |
| остатками азотистых оснований со стороны большой бороздки |
4 | 11 | 15 |
| остатками азотистых оснований со стороны малой бороздки |
0 | 1 | 1 |
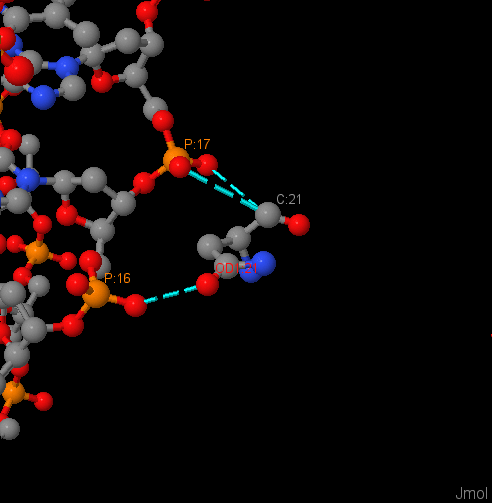
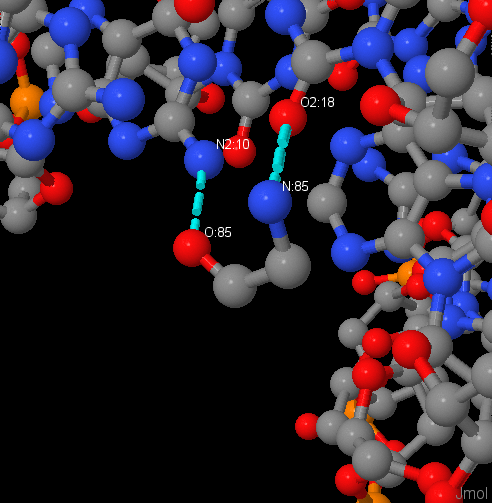
Самым большим числом контактов с ДНК (по 3) обладают аминокислоты Arg137 и Asn21 цепи A. Наиболее важным для узнавания цеппи ДНК остатком я предлагаю считать Gly85, потому что он связан с двумя остатками азотистых оснований, а не с остовом ДНК, и при этом в определённой ориентации N- и O- атомов. Рис. 3 и рис. 4 иллюстрируют приведённые примеры.
Хотя нуклеиновые кислоты и пептиды построены из мономеров различной природы, эти биомолекулы могут обнаруживать некоторое сродство и различным образом стыковаться.