Нуклеотидный BLAST
Как и во втором семестре, в третьем изучается BLAST, но работа теперь идёт не только с белками.
Определение таксономии и функции последовательности из практикума 6
В практикуме 6 фигурировала некая последовательность, секвенированная по Сэнгеру. Сейчас же для нас перестанет быть загадкой природа той последовательности.
Первым делом я запустил blastn по Nucleotide collection, используя в качестве запроса консенсусную последовательность из шестого практикума, потому что было неизвестно, чего ожидать. Самыми близкими находками стали цитохромоксидазы из митохондрий кольчатых червей, причём сходство было весьма велико (а РНК встречались исчезающе мало), поэтому я провёл затем и megablast (так как много сходных образцов) наряду с blastx (так как ген кодирует белок).
Выдача megablast так дифференцировала находки: 4 образца Polycirrus medusa со счётом от 1110 до 1218, затем 16 Polycirrus sp., у которых от 784 до 983 баллов. Ближайшие соседи в списке относятся к тому же семейству Terebellidae, что и род Polycirrus, набирая 592 балла и менее. Чтобы проверить, какое сходство вообще может быть между последовательностями, принадлежащими червям данного семейства, я скачал несколько примеров из выдачи и запустил blastn suite-2sequences (megablast). Вот что было скачано:
- Последовательность, которая имела лучший счёт и была первой в выдаче. Она выступала в роли первой в двух выравниваниях.
- Последовательности всех находок из рода Polycirrus.
- Последовательности худшей находки из рода Polycirrus и 4 следующих за ней.
- Последовательности двух худших находок Polycirrus medusa и двух следующих по списку.
- Последовательность одной из находок — Polycirrus phosphoreus. Её я выровнял со всеми последовательностями рода.
Рисунки 1-3 иллюстрируют результаты выравниваний.
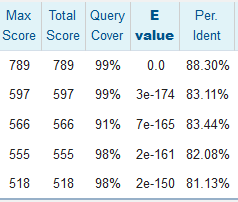
Характеристика выравнивания лучшего результата с последним из его рода и четырьмя следующими — из его семейства.
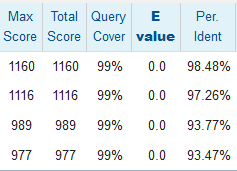
То же, но взято два образца того же вида и два следующих, Polycirrus sp.
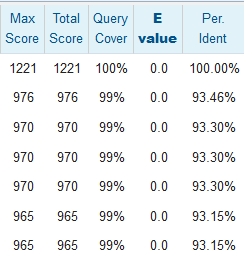
P. phosphoreus, первые строки таблицы с характеристикой выравнивания его последовательности с последовательностями всего рода (первая строка — выравнивание самой на себя).
Анализируя результаты, можно заключить, что исходный запрос явно принадлежит роду Polycirrus, так как именно с образцами этого рода счёт превышал 750 (даже 780), а E-value было равно нулю. (У разных родов семейства обычно до 600). Даже, скорее всего, виду P. medusa, потому что счёт выравнивания между разными видами обычно был меньше 1000, а у изучаемой последовательности с этим видом было больше 1100.
Интересно, что при blastx (кстати, генетический код был выбран Invertebrate Mitochondrial) первые результаты — при сортировке по E-value принадлежали не роду Polycirrus. Это связано с тем, что последовательность P. medusa в банке неполная (как и нуклеотидные последовательности были partial cds, так и здесь есть пометка partial). Некоторые более длинные записи имеют больше совпадений с запросом и, как следствие, выигрывают в счёте, однако только с P. medusa наблюдается 100% identity. Какие белки были найдены, можно увидеть на рисунке 4. (Практически все имеют одну функцию).

Разнообразие функций найденных белков (фрагмент).
Итак, с большой уверенностью можно утверждать: запросом был фрагмент гена цитохром c-оксидазы 1 кольчатого червя Polycirrus medusa.
Сравнение трёх алгоритмов BLAST
Теперь нужно сравнить результаты работы трёх запусков BLAST: megablast, blastn с параметрами по умолчанию, blastn с самыми чувствительными настройками.
Нужно было исследовать и последовательность из задания 1, и одну из CDS вируса из практикума 7 (выбранная). В таблице 1 приведены параметры запусков BLAST.
| Program Selection | megablast | blastn (по умолч.) | blastn (чувствит.) |
|---|---|---|---|
| General Parameters | |||
| Max target sequences | 1000 | 1000 | 1000 |
| Short queries | + | + | + |
| Expect threshold | 0.0001 | 0.0001 | 0.0001 |
| Word size | 28 | 11 | 7 |
| Max matches in a query range | 0 | 0 | 0 |
| Scoring Parameters | |||
| Match/Mismatch Scores | 1,-2 | 2,-3 | 1,-1 |
| Gap Costs | Linear | Existence: 5 Extension: 2 | Existence: 0 Extension: 2 |
| Filters and Masking | |||
| Filter | Low complexity regions | Low complexity regions | Low complexity regions |
| Mask | For lookup table only | For lookup table only | For lookup table only |
Отдельно покажем таксоны, в которых шёл поиск для червя и для вируса. (См. рис. 5-6).
Поле поиска для P. medusa.
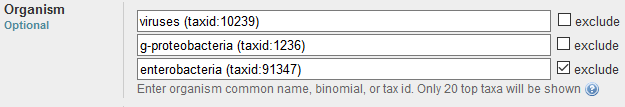
Поле поиска для вирусной CDS.
Таблица 2 показывает число находок алгоритмов.
| P. medusa | Pseudoalteromonas virus | |
| megablast | 14 | 4 |
|---|---|---|
| blastn по умолч. | 141 | 40 |
| blastn чувствит. | 144 | 244 |
Megablast во всех случаях нашёл выравнивания с identity от 76%. Разные параметры запуска blastn не так сильно отличаются по выдаче для червя, как для фага. Там только добавились последовательности с ещё меньшим процентом совпадений и счётом (см. рис. 7), при том это всё ещё цитохромоксидазы.
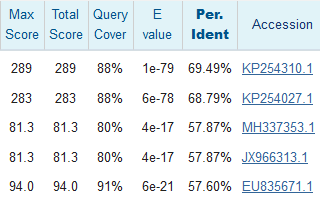
Три результата с худшим счётом и два результата до них.
Для вируса megablast нашёл родственные вирусы, blastn добавил 3 сборки бактерий, на которых данный фаг паразитирует, и некоторое количество других, уже близко не связанных; изменение параметров только увеличило число этих «других». Так как последовательность предположительно кодирует фактор репликации, то и встречаться он теоретически может много где. Есть и фрагменты с малым покрытием, напоминающие спейсеры из CRISPR-кассет к каким-то родственным последовательностям:
Query 1027 TTTGGCGTTCGCGTTGTTGAGCCACATCACGATGCAACCCCGCACTGGCATATGCT 1082
||||||||||| |||| ||||| |||||||||| || || ||||||||| ||||
Sbjct 1551554 TTTGGCGTTCGTGTTGCCGAGCCTCATCACGATGGCACACCACACTGGCATTTGCT 1551609
(Выравнивание с Vibrio rotiferianus AM7 DNA, chromosome 2, complete genome — AP019799.1).
Гомологи в неаннотированном геноме
Нужно было выбрать три типично эукариотических белка и проверить наличие их гомологов в сборке Amoeboaphelidium protococcarum X5 с kodomo.
Я выбрал тубулин (альфа-1 субъединицу), гистон H3.1, обратную транскриптазу-теломеразу. Нашёл нужные ID в Swiss-Prot, а затем выполнил команды:
- seqret sw:h31_mouse h31.fasta
- seqret sw:tba1a_mouse tubulin.fasta
- seqret sw:tert_mouse rt.fasta
- tblastn -query h31.fasta -db X5.fasta -db_gencode 6 > h31.hits
- tblastn -query tubulin.fasta -db X5.fasta -db_gencode 6 > tubulin.hits
- tblastn -query rt.fasta -db X5.fasta -db_gencode 6 > rt.hits
Использована таблица генетического кода 6, потому что она упоминалась на странице вида в NCBI Taxonomy. Полученные файлы: h31.hits, tubulin.hits, rt.hits. Для всех трёх белков результат положительный. Охарактеризуем достоверные находки (см. табл. 3).
| Файл | Число достоверных находок | Мин. счёт | Макс. счёт | Макс. E-value | Мин. E-value | Мин. % identity | Макс. % identity |
|---|---|---|---|---|---|---|---|
| h31.hits | 6 | 63,9 | 254 | 5e-13 | 4e-78 | 94 | 95 |
| tubulin.hits | 5 | 135 | 833 | 2e-37 | 0,0 | 41 | 93 |
| rt.hits | 2 | 129 | 138 | 4e-30 | 8e-33 | 27 | 27 |
У тубулина высокое сходство между альфа- и бета-субъединицами, поэтому среди находок, вероятно, обе. В целом тубулин и гистон оказались намного более консервативны, чем обратная транскриптаза (может быть, это связано с тем, что они выполняют структурную функцию, а теломераза — только каталитическую). Наличие малоконсервативных регионов отражено и на странице фермента в Pfam. Каталитические и трансмембранные домены расположены после 450-й аминокислоты, а этот участок и попал в выравнивание.
Поиск гена белка в контиге
Требуется взять контиг или скэффолд в неаннотированном геноме и найти в нём наиболее достоверный ген белка.
В геноме, который я рассматривал в практикуме 7, белки аннотированы, поэтому я нашёл новый. Потом я выбрал скэффолд длиной около 40 килобаз. Поиск гомологов был выполнен на сайте NCBI BLAST (blastx по refseq_protein, таксон Chlorophyta/Embryophyta group (taxid:33090)).
Самый большой счёт набрал ген пируваткиназы. Приведём его выравнивания с первым результатом из выдачи, не имеющим пометки PREDICTED (plastidial pyruvate kinase 4, chloroplastic isoform X1 [Camellia sinensis], Sequence ID: XP_028077586.1).
- Range 1: 165 to 615
Query 39429 RNYVVSATNLIHYLALKCLDVEQLKEDLSSVGLLNLETINPCVIANLTAGIQILENLKSC 39250 R Y+VSA NLIHYLALKCLDVEQLKEDLSS+GLLNLETIN V+A+L+AG Q+LENLKS Sbjct 165 RKYLVSAANLIHYLALKCLDVEQLKEDLSSIGLLNLETINSHVLASLSAGTQMLENLKSN 224 Query 39249 PMSIKENTGGEISNRKSLDKHKKGEIIIDTMRRKASSNMQLLLGPLAEERTAHIMVTVGQ 39070 + KE+ I +KSLD KKGE I+ MR KA N + LLGPL EERT HIMVTVGQ Sbjct 225 SLDRKESVVEGIFTQKSLDIQKKGEFTINVMREKACFNRESLLGPLQEERTTHIMVTVGQ 284 Query 39069 EVTESEMLITDILKRGATVIRINCAHGNPSIWSEIIRRVRRSSQMLEKPCRILMDLAGPK 38890 EV ESE LITD++ G T+IRINCAHGNPS+WSEIIRRV+RSSQMLEKPCRILMDLAGPK Sbjct 285 EVLESETLITDLVTTGTTIIRINCAHGNPSVWSEIIRRVKRSSQMLEKPCRILMDLAGPK 344 Query 38889 LRTGKMKAGPCVVKISPKKNAYGDIVLPAQVWLCHKGANPPAGHISPDAILSIDGQEFLN 38710 LRTGK+KAGPC +KISPKKNA G+++ PAQVWLC K A PP ++SPDA++S+DGQEFL+ Sbjct 345 LRTGKLKAGPCAMKISPKKNARGEVIFPAQVWLCPKQAGPPPANLSPDAVISVDGQEFLS 404 Query 38709 KLQVGDTVRLRDARGRKRSLKICKRFPIFAGTAFMAECSRTAYVESGTQLYMKRQKGRSA 38530 KL++GD V+ DARG++R LKI ++FP+F+G MAECS+TAYV+SGT+LY+K +KG+ Sbjct 405 KLELGDAVKFFDARGKQRILKISQKFPVFSGVGLMAECSKTAYVQSGTELYIKGKKGKFT 464 Query 38529 IGEVVEVPSVEQYVRLRVGDLLIISRESSVGMVELTETAIDAYRVTCSSNYLFDSVKPGD 38350 +G +V++P+VE +VRLRVGDLLIISR+SS + T I A+++TC S YLFDSVKPG Sbjct 465 VGLMVDIPAVEPFVRLRVGDLLIISRDSSDDQIASTGATIGAHKITCCSGYLFDSVKPGQ 524 Query 38349 PIAFDDGKIWGIIQGASASEIIVSITHASPKGTKLGAEKSINIPESSIKFEGLTTKDLMD 38170 PIAFDDGKIWG+IQG S SEIIVSITHA +GTKLG+EKSINIPES+I+FEGLT+KDL D Sbjct 525 PIAFDDGKIWGMIQGTSISEIIVSITHAGLRGTKLGSEKSINIPESNIRFEGLTSKDLKD 584 Query 38169 LEFVAAHADMVGISFIRDVCDIVVLQAELEK 38077 L+FVA+HADMVG+SF+RDV DI+VL+ ELEK Sbjct 585 LDFVASHADMVGVSFVRDVRDIIVLRQELEK 615 - Range 2: 632 to 708
Query 37839 GFEKLPLLLLEAMKSSNPLGVMIARGDLAVECGWERLGDIQEQILSICSAAHVPVIWATQ 37660 GFE LPL+LLEAMKS NPLGVMIARGDLAVECGWERL DIQE+I+SICSAAH+PVIWATQ Sbjct 632 GFENLPLMLLEAMKSPNPLGVMIARGDLAVECGWERLADIQEEIISICSAAHIPVIWATQ 691 Query 37659 VLESLVKSGVPTRAELT 37609 VLESLVK GVPTRAE+T Sbjct 692 VLESLVKFGVPTRAEIT 708 - Range 3: 616 to 630
Query 37945 RKLQNLGIVLKIETQ 37901 R+LQNLGI+LKIET+ Sbjct 616 RRLQNLGIILKIETK 630 - Range 4: 3 to 165
Query 41080 MKS*FIDIRHNTSSFVFQAIPTANIFYSRSSMPSGNVRALQFRLKIKNSVFRYQIFPLFL 40901 M S I + N ++ AI TAN RS+ G L +K VFR +I L Sbjct 3 MGSSSICVVTNQATLSNYAIQTANTSNFRSTNFLGKFTYQPSILHLKELVFRDKIAQLLK 62 Query 40900 KEKGSLAPTTLVFSTQNESDESESGNSQSCIEDQIHVALENNDVSRYP-DVEVAPFTHSE 40724 +++ + APTT+VF+ NE+DE+E G S + +DQ+ A ENN + P D VA + Sbjct 63 RQRRNPAPTTIVFAIPNENDEAERGISHTFTDDQMLTASENNGNFKDPGDEVVASLSQLG 122 Query 40723 TDLSQSAEQLGNQGNLLDKLKAVHLHILAMEQWNASRIKLCHR 40595 + LSQ+A L NQ +LLDKLKAV LH+LAMEQWNASR+KLCHR Sbjct 123 SGLSQNAVSLENQADLLDKLKAVQLHVLAMEQWNASRLKLCHR 165 - Range 5: 715 to 759
Query 36974 RANCIMLNKGKHVVDAISTLDVILQSKSTKMKAELKPLVLSSHLF 36840 R++CIMLNKGKH+++A+STLD IL STK+KAEL+PL++S+HLF Sbjct 715 RSSCIMLNKGKHILEAVSTLDNILHGNSTKVKAELRPLIISNHLF 759
Для этой находки суммарный счёт составил 1031; E-value 0,0; 69,62% identity — то есть можно уверенно утверждать гомологию.
В четырёх заданиях восьмого практикума применялись самые разные алгоритмы как нуклеотидного, так и нуклеотидно-белкового BLAST, причём не только на сайте NCBI, но и на kodomo. Может быть, после этого будет проще запомнить, где tblastn, а где blastx!