Последовательности в fasta формате с исправленными проблемными нуклеиотидами и полиморфизмами.
JalView-проект с выравниванием прямого и обратного прочтений.
Участки с проблемными нуклеотидами выделены красными рамками.
| |
534й нуклеотид в верхней последовательности я определила как гуанин, так как пик гуанина значительно выше остальных. К тому же, в нижней последовательности на этом участке явно выделяется гуанин, и посторонние пики практически отстутствуют. |
| |
На 549 позиции нижней последовательности я решила поставить g, так как в верхней хроматограмме довольно однозначно определяется гуанин. |
| |
Нуклеотид на 620й позиции в нижней последовательности я обозначила с, так как в верхней хроматограмме однозначно определен цитозин. К тому же, и в самой нижней хроматограмме пик цитозина заметно выше остальных. |
| |
Приведённую позицию я в обеих последовательностях обозначила g, так как и в верхней, и в нижней хроматограмме преобладает пик гуанина. |
Общая характеристика хроматограммы:
Исходные файлы в .ab1 формате:
Прямое прочтение
Обратное прочтение
Ниже приведён фрагмент нечитаемой хроматограммы.
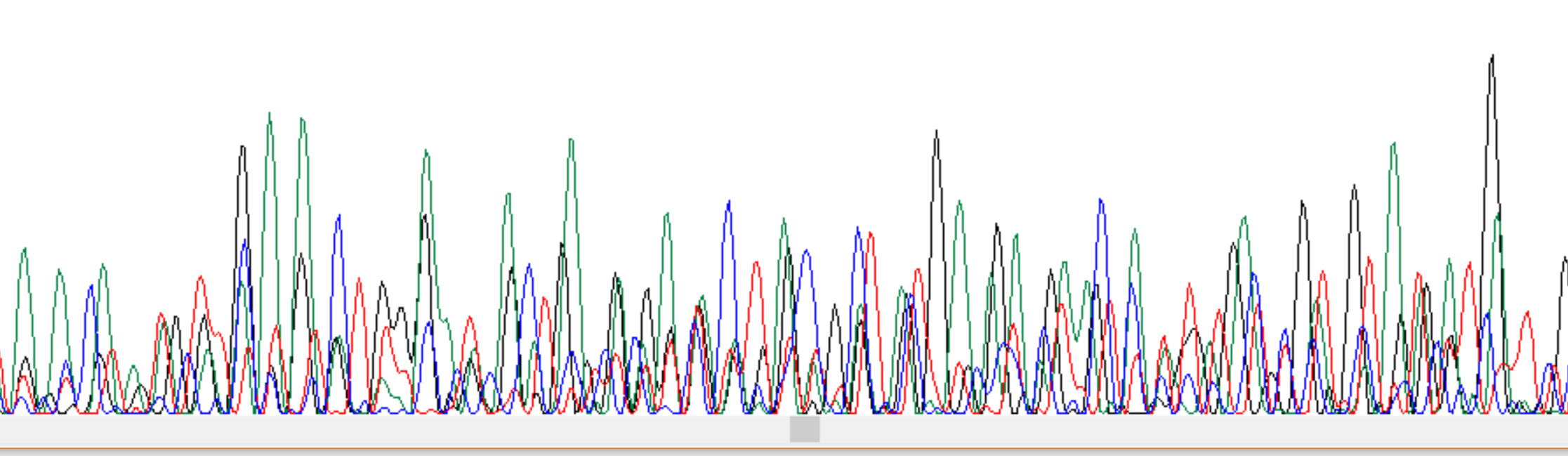
Видно, что пики часто пересекаются и накладываются друг на друга, поэтому определить по ним искомую
последовательность невозможно. Возможно, в анализируемом образце было несколько различных ДНК, так как
можно заметить, что на некоторых позициях разные пики практически совпадают по высоте.