Сигналы, мотивы, PWM.
Цель практикума: поискать с помощью программы MEME сайт связывания транскрипционного фактора, регулирующего синтез пуринов у одной из гаммапротеобактерий.
Для поиска мотивов было предложено найти у своей бактерии в базе данных Uniprot 8-10 генов, принимающих участие в биосинтезе пуринов (поиск по ключевому слову "Purine biosynthesis"). Далее приведены данные о найденных белках у Salmonella typhimurium LT2 (Uniprot-мнемоника - SALTY).
| Entry | Entry name | Protein names | Gene names | Coordinates |
| P74881 | PUR4_SALTY | Phosphoribosylformylglycinamidine synthase | purL STM2565 | complement(2708255..2712142) |
| Q8ZNW5 | PURT_SALTY | Formate-dependent phosphoribosylglycinamide formyltransferase | purT STM1883 | 1975661..1976839 |
| P65882 | PURA_SALTY | Adenylosuccinate synthetase | purA STM4366 | 4609111..4610409 |
| P58688 | FOLD_SALTY | Bifunctional protein FolD | folD STM0542 | complement(602705..603571) |
| P26977 | PUR2_SALTY | Phosphoribosylamine--glycine ligase | purD STM4175, STMF1.31 | complement(4391174..4392463) |
| Q8ZN60 | GUAA_SALTY | GMP synthase [glutamine-hydrolyzing] | guaA STM2510 | complement(2622804..2624381) |
| O68446 | PURR_SALTY | HTH-type transcriptional repressor PurR | purR STM1430 | complement(1507911..1508936) |
| P74883 | PUR5_SALTY | Phosphoribosylformylglycinamidine cyclo-ligase | purM STM2499 | complement(4392475..4394064) |
| P26978 | PUR9_SALTY | Bifunctional purine biosynthesis protein PurH | purH STM4176, STMF1.30 | 2613072..2614109 |
| P65889 | PUR7_SALTY | Phosphoribosylaminoimidazole-succinocarboxamide synthase | purC STM2487 | complement(2602103..2602816) |
Сборка генома Salmonella typhirium LT2 (AC: AE006468.2)
По координатам кодирующих последовательностей в геноме были найдены координаты 100 предшествующих им (со стороны 5'-конца от стартового кодона) нуклеотидов. Такие нуклеотиды составляют Upstream-регионы генов. Далее с помощью команды descseq upstream-регионы были собраны в единый файл upstream.fasta.
Затем была запущена программа MEME, которая и должна найти мотивы в последовательностях. Командная строка: ememe upstream.fasta -nmotifs 3 -revcomp
Полная выдача программы МЕМЕ.
Разберём, что говорят эти результаты.
Для каждого мотива приведено его информационное содержание (Information Content) - числовой параметр,
позволяющий оценить, является данная последовательность мотивом или случайной находкой. По грубым оценкам, слово длины n = I/2 (где I - инормационное содержание)
может встретиться в геноме раз в 4^n пар нуклеотидов.
E-value показывает, насколько велика вероятность находки с таким же или большим весом.
Также приведены LOGO - схематичные диаграммы, в которых высота столбца равна I этой позиции и показывает его вклад в суммарное I всего мотива, а высота букв равна
I столбца, умноженному на вероятность встретить данную букву на данной позиции. Таким образом, LOGO отображает наиболее правдоподобную последовательность искомого мотива.
Основные характеристики предполагаемых мотивов приведены ниже:
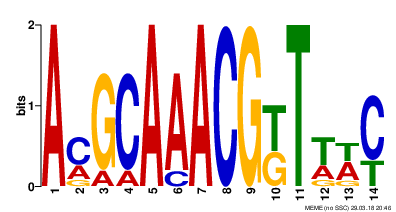 |
|
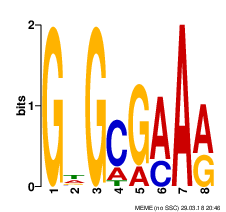 |
|
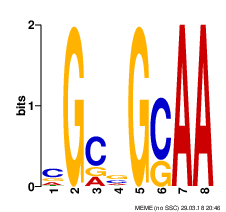 |
|
В данном случае мотивом можно считать только 1ую последовательность, найденную программой. Такой вывод можно сделать на основании низкого E-value (тысячные доли единицы) и довольно высокого информационного содержания.