Множественное выравнивание последовтельностей
1. Алгоритм
Алгоритм для сравнения множественных выравниваний был взят у Кирцовой Ксении, 102 группа. Безмерная ей благодарность!:)
2. Сравнение множественных выравниваний
Для сравнения я выбрала 11 последовательностей человеческих химотрипсин-подобных протез. Все выбранные мной структуры принадлежат одному клану в базе данных MEROPS. Однако не смотря на то, что все струрктуры имеют общий домен и единое строение каталитическо центра, выбранные для выравнивания белки выполняют разные функции и принадлежат разным компартментам в организме человека. Из-за этого я ожидаю, что при выравнивании точно можно будет определеить общий для всех блок, соответсвующий каталитическому центру и его окружению (химотрипсиновая складка). Но так же, я предполагаю, что в выравнивании любым из алгоритмов будет заметно отсутсвие сходства на протяженных участках последовтельностей. Так как при участии в разных каскадах в организме белкам требуются разные структурно-функциональные части. Список AC в базе данных UniProt. Эти последовательности были выровнены с помощью программ Mafft, Muscle, Tcoffee with defaults. Проект Jalview с выравниванияими.
Далее в таблице приведены попарные координаты одинакого выровненых блоков для разных алгоритмов множественного выравнивания.
| Muscle vs Mafft | Muscle vs Tcoffee |
| Блок 1 (703-713) (632-642) Блок 2 (723-739) (652-668) Блок 3 (756-774) (685-703) Блок 4 (777-778) (706-707) Блок 5 (782-785) (711-714) Блок 6 (792-817) (721-746) Блок 7 (826-839) (755-768) Блок 8 (857-867) (785-795) Блок 9 (885-890) (813-818) Блок 10 (903-913) (831-841) Блок 11 (921-931) (847-857) Блок 12 (943-962) (869-888) Блок 13 (979-986) (900-907) | Блок 1 (623-643) (755-775) Блок 2 (648-668) (780-800) Блок 3 (685-693) (817-825) Блок 4 (699-700) (831-832) Блок 5 (711-712) (843-844) Блок 6 (724-749) (857-882) Блок 7 (756-768) (889-901) Блок 8 (785-802) (916-933) Блок 9 (810-812) (942-944) Блок 10 (814-818) (946-950) Блок 11 (831-841) (963-973) Блок 12 (850-858) (982-990) Блок 13 (869-888) (1001-1020) Блок 14 (891-894) (1023-1026) Блок 15 (904-907) (1036-1039) |
Кроме такого для каждой пары выравниваний есть идентично выровненные колонки, не вошедшие в блоки.
| Muscle vs Mafft | Muscle vs Tcoffee |
| Колонка 1 (871) (799) | Колонка 1 (645) (777) Колонка 2 (705) (837) Колонка 3 (721) (854) Колонка 4 (806) (937) Колонка 5 (862) (994) |
По сравнительным таблицам видно, что, во-первых, идентичные блоки выравниваний начинаются далеко не с начала последовательностей. Как я и предполагала, "смысловое" выравнивание получлось только на участке, близком к каталитичекому центру. Соотвественно и идентичные блоки выравнивай у разных алгоритмов заметны только на участках, начинающихся примерно с позиции большей, чем 600. Во-вторых, заметно, что в первом приближении (опираясь только на таблицы сравнения) для данных последовательностей алгоритм Tcoffee дает в результате более схожее с алгоритмом Muscle выравнивание, чем Mafft. Это проявляется и в количестве блоков сходных при выравнивании, и в количестве единиченых идентичных позиций выравниваний.
3. Выравнивание по совмещению структур
Для данного задания я использовала белки того же клана, опираясь на единстве заведомо известной пространсвенной структуры химотрипсиновой складки. Для этих последовательностей также было произведено выравнивание алгоритом программы Muscle. Ссылка на проект MSA. Выравнивание по структуре проводилось с помощью Expresso Tcoffee. Ссылка на результат выравнивания. На веб-ресурсе выравнивание отображается в окрашенном виде по степени "качества" выравнивания. Заметно, что красным цветом, который показывает хорошо выровненненные участки по структуре, как раз окрашен участок, окружающий каталитический центр. При этом в последовательностях друг на друга выравниваются и разные аминокислоты, которые, однако, в пространсве создают одинаковый мотив. Именно это является отличительной особенностью выравнивания по структуре. Использование такого типа выравнивания позволяет выравнивать структуры, которые давно разошлись в эволюционной перспективе, но при этом сохранили пространственное сходство, формируемое сходными мотивами в 3D cтруктуре аминокислот. Для наглядости: на рисунке 1, изображено совмещение в PyMol. Заметно, что общая стурктура химотрипсиновой складки присутсвует в обеих приведенных структурах, не смотря на то, что представлены белки, занимающие в организме человека разные места.
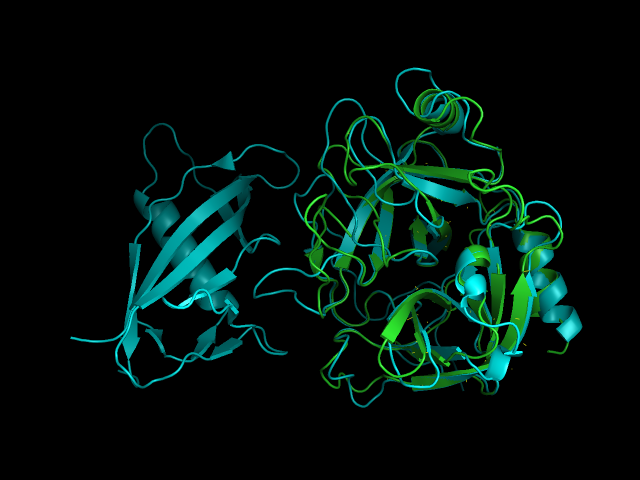
Рисунок 1. Совмещение cтурктур каликреина-8 и протромбина
4. Описание алгоритма
В данном задании я выбрала алгоритм множественного выравнивания Muscle. Приведенное далее описание опирается на статью.
Глобально данный алгоритм опирается на суммарную оценку попарных выравниваний. MUSCLE использует две меры расстояния для пары последовательностей: расстояние в k-мерах (для невыровненной пары) и расстояние, вычисленное по методы Кимуры (для выровненной пары). Сам алгоритм разбит на 3 функциональные части. Сначала происходит попарное выравнивание всех последовтельностей с вычислением расстояния в k-мерах. Получается матрица расстояний, на основе которой просходит кластеризация по методы UPGMA c построением дерева расстояний. На основании данного дерева, идя от "листьев" к "корню", происходит построение множественного выравнивания первой версии. Далее, чтобы учесть неравноценность переходов между разными классами нуклеотидов, проиходит построение новой матрицы расстояний с учетом расстоний по методу Кимуры. Далее точно так же, как и на первых этапах, проиходит построение дерева расстояний и множетсвенного выравнивания второй версии. На третьем этапе проиходит циклическая проверка "эффективности" выравнивания. Так, последовательно удаляют по одному ребру из второго по счету дерева. Получаетя уже два выравнивания, для каждого из которых считается профиль. Далее проиходит повторное выравнивание двух профилей деревьев. Здесь же проиходит расчет суммарной оценки попарных выравниваний, если данная величина улучшается, то новый расклад в выравниваниях сохраняется, в противном случае он отбрасывается и итерации продолжаются до нахождения наилучшего выравнивания.