Сравнение работы megablast и blastn
Выбор организмов
В данном практикуме использовалась геномные сборки двух штаммов одгого вида энтробактерий. Известно, что организмы данной группы могут довольно сильно отличаться ввиду экологической разности. Выбранные организмы:
| Enterobacter cancerogenus strain MiY-F(NZ_CP045769.1) Enterobacter cancerogenus strain CR-Eb1(NZ_CP025225.1) |
Графики выравниваний
Мною использлвались алгоритмы megablst и blastn с настройками по умолчанию с запросом по AC геномного браузера.
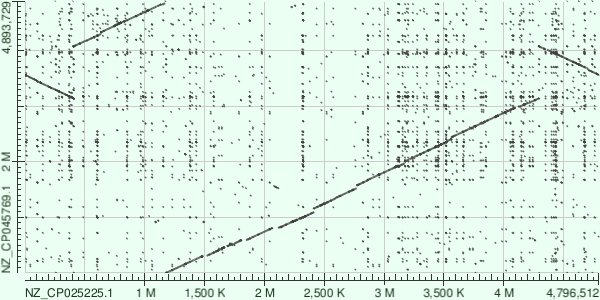
Рис. 1 Dotplot megablast
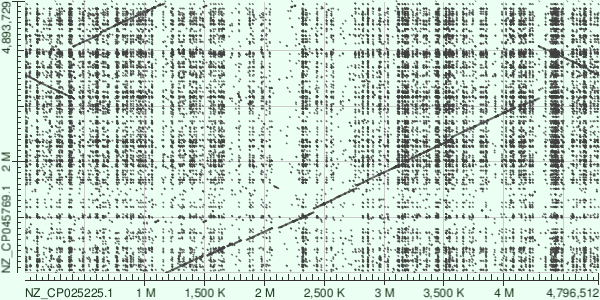
Рис. 2 Dotplot plot_blastn
Заметно, что "общие очертания", то есть крупные геномные перестройки видны на обоих графиках. Однако, явно заметно, что карта выравниванием blastn имеет большое количство точек, вне основной оси генома. Это указывает на большое количество коротких повторов.
Описание графика
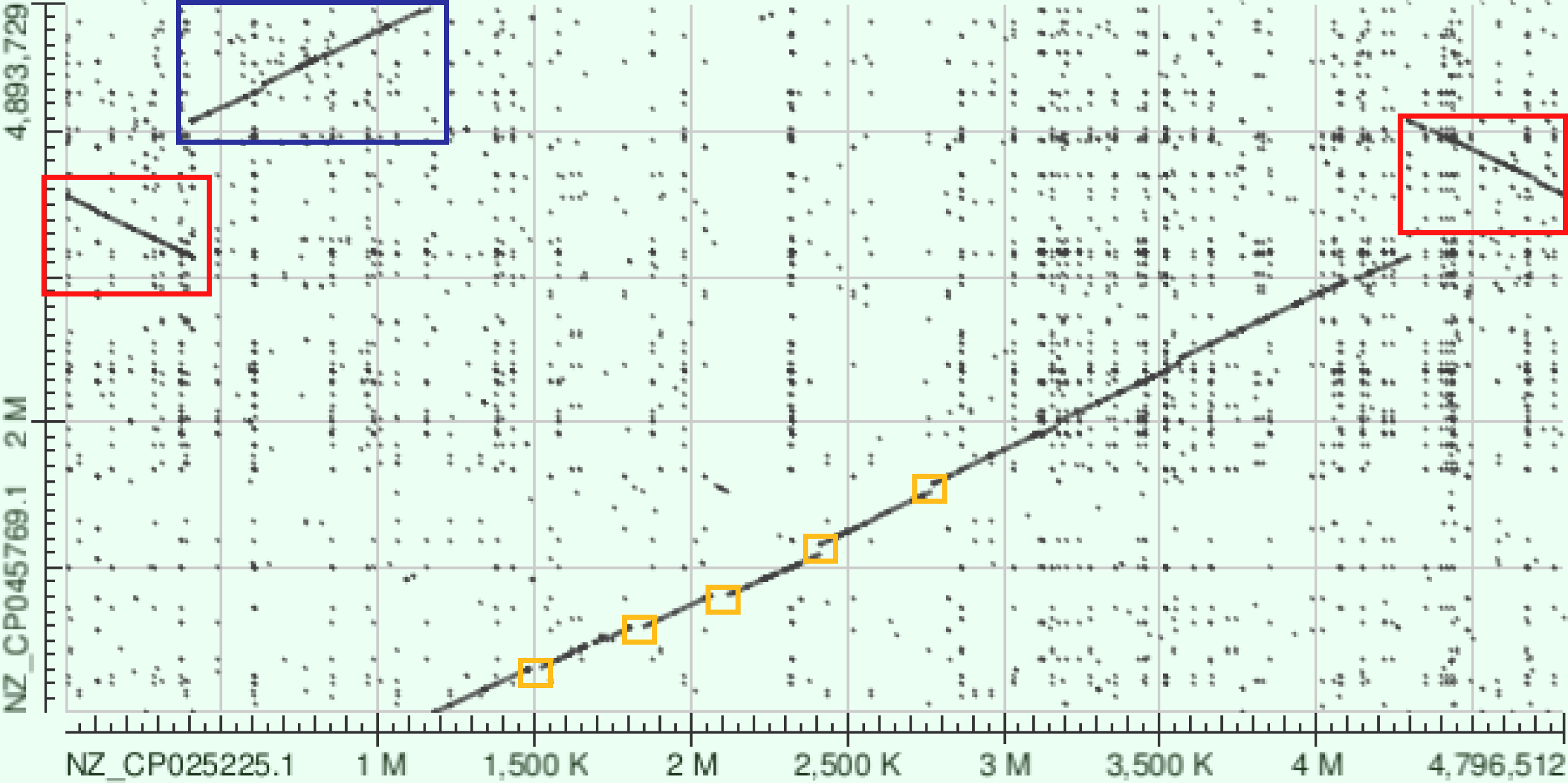
Рис. 3 Dotplot plot_blastn с метками
Синим цветом выделена часть геномов, которая по моему мнению отбражает разный старт начала прочтения кольцевых геномов, на это указывает различное вертикальное расположение, но сохранение положительного коэфициента наклона прямой графика. Красным цветом, вомзожно тоже обозначены часть геномов, которые имеют такой вид из-за разных точек начала. Однако, в отличие от первого случая наклон в обратную сторону. Это по всей видимости указывает на инверсию в одном из геномов, относительно другого. Несколькими желтыми участками показаны участки инсерций/делеций, однако каждый из них имеет сравнительно небольшую длину.