BLAST
Поиск в геноме гена, кодирующего δ-субъединицу АТФ-синтазы
В данном практикуме использовалась геномная сборка организма Equus caballus(Домашняя лошадь) В файле транслированных последовательностей текстовым поиском была найдена запись описывающая белковую последовательность δ-субъединицы АТФ-синтазы, которая имеет идентификатор XP_023502124.1
| >XP_023502124.1 ATP synthase subunit delta, mitochondrial [Equus caballus]
MLPAALLRRPGLGRLVRQARAYAEAAAAPAPAAGPGQMSFTFASPTQVFFNSANVRQVDVPTQTGAFGILAAHVPTLQVL
RPGLVVVHAEDGTTSKYFVSSGSVTVNADSSVQLLAEEAVTLDMLDLGAAKANLEKAQSELSGAADEAARAEIQIRIEAN EALVKALE |
По идентифкатору в таблице локальных особенностей былана найдена информация о координатах в геноме и идентификаторе геномной записи в базе данных:
| >NC_009150.3:642220-644748 ATP5F1D [organism=Equus caballus] [GeneID=100146850] [chromosome=7] TCCGCCCGCCGCTGCCGCCGCCGCCATGCTGCCCGCCGCGCTGCTCCGCCGCCCCGGCCTGGGCCGCCTC GTGCGGCAGGCCCGCGCCTACGCTGAGGCTGCCGCCGCCCCGGCCCCCGCCGCGGGCCCGGGACAGATGT CCTTCACTTTCGCCTCCCCAACGCAGGTTCGGACGCCGGCCGGGCTCGGGGCCCCGCCATGGCCGCCGCC CCTTTCCGGACCCGGAACCCGGACCCTCGTCCCCCCACCCCTGCCCGTGTGGTCCCGGATCCGAGCCCCC AGCCCCAGGGTCACCGCCCCCCATTCCAGACCCGCGCCCTGTGCCCTTCCCCCGTGCAGTCCCAAATCTG AGACCCCCAACCTCGCTGGTCACCGCCCCCCATTGCGGACCCGTGCCCTGTGCCCTTCCCCCGTGTGTTC CCGAATCTGAGACCCCCCCCCCACCCGCCCGGGTCACTGCTGCCCATTCCGGACTCGCCTCCTGTGTCCT TCCCCCGTGCGGCCCCGAATATGAGATCTCCCAAACCCCCTGATCACTAGCCCCCCATTCCTGACCTGTG TCCTGTGCCCTTCTCCCCTGTGGTCTAGAATCTGAGGTTTCCCACCCCATCCCTGGGTCACTGACTCCCT TTCCAGACCCCCACTCCCACCTGCTATGTGGTCCAGGATCCAGCGTCCCAACTTCGGGGTCACTGCTCCT GTTTCTGCTCCTCTGTCTGTGGTGCAGGATCTGATTTCCTTAATTTCCGGGTGACCGATCCCATGCTTTT CCCCCGTGTGGTCTCAAATCTGAACCTACCAACACTTAGGTTGCCGCCCCCCATTCCAGACCCGGTCTTT CGTCCTTCCCACATGGGGTCTGGAGTCCGAAATCCTGGAATTTCCACTTGGAACTCCACACCCCAGGGCC GGGGCAGGTCCAGAGCCTCTGCCCCTCTCCCGAGAGCAGGTCCTCCTGGGTTGGCACCCTGTGCTGCTTG TGTCCTCGCAGCGCGCGCCTGTGAAGGGGCTGGGGTCACTTGACTTCCCCACCTTCTTGTGGACAGAGGG TCTCGGGTGCCATCCAGAGAGCAGAAGCCGAGCGCCCGCCCCAGCGCCCCGTTCTCCCCCCACCTGGCCC GTTTCACTGCAGGGAAACTGAGTCCAGGCTGCAGGGGCCTGGCCTGCGGTCACCCATTGCCCGTGTGTCC TCCTCAGGTGTTCTTCAACAGCGCCAACGTCCGGCAGGTGGACGTGCCCACGCAGACGGGGGCCTTCGGC ATCCTGGCCGCACATGTGCCCACCCTGCAGGTCCTGCGGCCGGGGCTGGTGGTCGTCCACGCTGAGGACG GCACTACCTCCAAGTACTTTGGTGAGTGGAGTGCGAGGAGGGGGCTGAACCAGGCCAAGGCAGGGTCCAT GTGTCACTTGAGGCCACAGCGAGAAGCCAGCCAGAGGCACTGCGCGCGGCCCAGTCTTGGCTGCGGTTGA GTAGCGTGTCGCGGGGGGAATGGGCGCCAGGAGGAGAAGTAAAGCCTCGTGGGGGGGCTGGTTTGAGGAG GAAGGAAGCCCGTGCGGGAGGAAATGCTCTGAGAAGACCTTGCTCCAACTTCTCGTTCCCAGCCTAGGAC CACGGTGTGAAGCCGGGTCTCTCACCCCTGACCCTGACTCAGCTCGGGGACTAGTGCGTGAAGGGGGGCT CTGCTCTCTCCGGATGTCAACTCCCAGTGCCGGGCCCCAGGGCCACCCTGTGGGGCTGCTCTCCTGGTTC CAGGGCTGACCTCGTCCCAGGTGGACGTGGAGCCCTCACTCCTGCTTGTGCCCTCGCAGTGAGCAGCGGC TCGGTCACCGTGAACGCGGACTCCTCAGTGCAGCTCTTGGCCGAAGAGGCCGTGACACTGGACATGTTGG ACCTTGGGGTGAGTGTCCCCAAGGGTGAAACTGAGGCATTGAGCAGGCGGGAGCTGCGGGAGGGCTCCCA GGATGGGGGAGGGGTGGGCACCAGGCCCGGGTCTGCTGGGCCCTCACCCGCCACCCACCCCGTGCAGGCC GCCAAGGCGAACTTGGAGAAGGCGCAGTCGGAGCTGTCAGGGGCGGCGGACGAGGCCGCCAGGGCCGAGA TCCAAATCCGCATCGAGGCCAACGAGGCCCTGGTGAAGGCGCTGGAGTAGGCGGTGCGCGGTCCTGGCCT GCAGGGACCCCGAGGCTGGCGCTGGGTGGGGGCTGCTCTGGGTTGGCAGAACCAGCTTCTGCAGGTCTAG ACTGGGTGCTGGGGGGCGTCCTTACAGAGGAGGCCGCCAGGGGGCAGCGCAGTGCCGGCGTCTGCCCCAG GAGCACCCACGGGGCCCCATCCCCTGGCCCAAGTGGGCGGGCGTGGGGGTGAGCTCAGAGACTTTGCCCT GCCCTCGGAGGGGCTGTCACCCCGGGGGCTCTTCTCCCGTCTCCCAAGATCCCCCAACCTGACCCACCGC TGCCACCACCCCTCCTCTCTGGCTGCCCCATGGGACAGCCGGCCCCGGCCCGCGCCCCCATTAAAGACCT GGGACCCGC |
Так как, информация о последовтельноти гена указывает на расположение на 7 хромосоме, видимо эта субъединица митохондриальной АТФ-синтазы синтезируется с ядерных транскриптов.
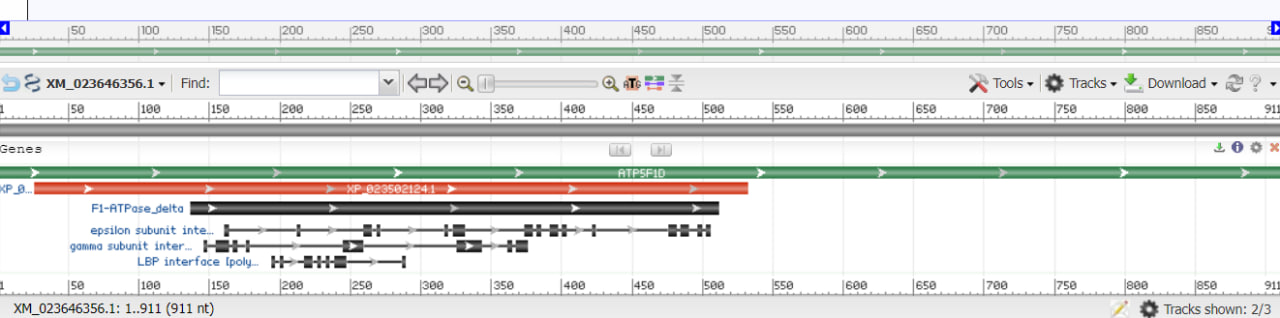
Рис. 1 Карта гена, кодирующего субъединицы АТФ-синтазы
На карте гена видно, что с него синтезируется не одна субъединица фермента, причем кодирующие области пересекаются, по видимому это реализуетсяя за счет альтернативного-спласинга.
Разные варианты BLAST для фрагмента ДНК
В данном упражнении для оценки сходства изучаемого белка с гомологичными белками других организмов была проведена оценка сходствва по группе экариотических организмов группы Araneae(Пауки). Столь эволиционно отдаленная группа сравнения была выбрана не случайно. Так, используемый мной организм является вторичноротым, а организмы сравнения принадлежат группе первичноротых. Такой выбор был сделан так как предполагается, что изучаемый фермент является эволюционно консервативным и у большства организмов должен иметь схожую струтуру, для поддержания эффективности его функции.
Первым я использовала для поиска алгоритм blastn с паремтрами по умолчанию по базе данных refseq_genomes, которая содержит 4 вхождения для поиска в выбранном таксоне. Однако по моему запросу не нашлось ни одной статистически значимой находки.
Далее был использован tblastn с аналогичными параметрами. Здесь уже было 4 находки, однако с невысоким счетом. Результаты поиска
Таким образом, мною было установлено, что при сравнении структур у отдаленных групп при применении различных алгоритмов поиска получаются разные результаты. Как я понимаю, такие результаты показывают, что даже конервативные последовтельности могут быть неодинаковыми на уровне поледовательности ДНК, однако сохранение структуры белка явно наблюдается даже у представителей разных таксонов.
Поиса в геноме эукариота гены основных рибосомальных РНК по далекому гомологу
В данном задании иcпользовалася локальный blast для поиска в описываемом геноме гомологичнных некодирующих последовтельностей. Использовались последовтельности 16S и 23S бактериальные рРНК Escherichia coli. Известно, что у экариот гомологичными им являются 18S и частично 28S рРНК. Они выполняют следующие функции:
| рРНК | Функция |
| 16(18) рРНК | Поддержание структуры рибосомальных белков Связывание с факторами инициации трансляции Cтабилизация правильных кодона и антикодона в А-сайте |
| 23(28) рРНК | Катализ петидилтрансферазной реакции |
Для того, чтобы искать в исследуемом геноме гомологичные последовтельности необходимо было созддать базу данных на основе геномной сборки:
| makeblastdb -in GCF_002863925.1_EquCab3.0_genomic.fna -dbtype nucl |
Далее были сделаны два запроса по последовательноям рРНК кишечной палочки:
| blastn -task blastn -query 16S.fna -db GCF_002863925.1_EquCab3.0_genomic.fna -outfmt 7 blastn -task blastn -query 23S.fna -db GCF_002863925.1_EquCab3.0_genomic.fna -outfmt 7 |
Выдачу в виде таблицы можно посмотреть: 16S, 23S
Видно, что находок много при обоих запросах. Однако статичтически значимых относиетльном немного. Есть и находки, которые по проценту идентичности близки к 100, только по координатам видно, что длина выровненых последовтельностей крайне мала и соответвено e-value превышает 6, что исключает гомологию. Так, для 16S рРНК приемлемый e-value (хотя бы <1) только для трех находок. Это находки в геноме с идентификаторами: NC_009161.3, NC_009144.3, NW_019643269.1. При этом идентификатор NW_019643269.1 указывает на скаффолд, другие два на собранные хромосомы. Кроме того, я заметила что координаты и статистичсекие показатели находки на скаффолде полностью сопрадают с находной на одной их хромосом, что указывает на то, что данный скаффолд попал в сборку данной хромосомы. Таким образом, для 16S найдено два гомолога на хромосоме 1 и хромосоме 18. С 23S РНК дела обстоят несколько иначе. Находок в целом больше и так как в геноме собраны все хромосомы наверное имеет смысл рассмотреть находки именно на хромосомах. Статистичсеки значимые находки указывают на хромосомы 3 и 1. Эта информация согласуется с находками для 16S, так как рРНК синтезируется путем созревания предшественника, содержащего разные рРНК. Однако, заметно что на каждой из этих хромосом не одна находка, а несколько участков сходства. При этом на хромосоме 1, хоть две значимые находки и удалены на расстояние около 2000 нуклетидов, судя по координатам а запросе (последовтельности из E.Coli) находки все же являются частями одной гомологичной находки. Учитывая общую длину последовтельности запроса и расстояние в координатах между находками, можно предположить гомологичную находку с большим сходством в "начале" и "конце" последовательности. На хромосоме 3, координаты указывают на близкое расположение: около 200 нуклеотидов, и учитывая длину и последовательность расположения находок, можно предположить, что это лишь один гомолог. Также большее количсетво находок по сравнению с 16S РНК, как мне кажется связано с тем, что описываемый мной организм эукариотический и у данного типа рРНК меньше необходимость сохранения полной гомологии, так как у эукариот нет последовтелности Шайна-Дальгарно, для которой нужна строгая последовательность.