Практикум 10
1. Поиск консервативных мотивов в выравнивании
Для данного практикума мной было выбран домен pfam Connexin (PF00029). Белки с данным доменом встречаются только у эукариот и играют ключевую структурную функцию в образовании межклеточных щелевых контактов. Оно содержит 111 соответсвующих записей swissprot и 169 последовательностей в выравнивании seed. Выравнивание seed было скачано и с помощью Jalview окрашено по консервативности позиций. Среди таких позиций был выбран наиболее продолжительный консерватиный паттерн.
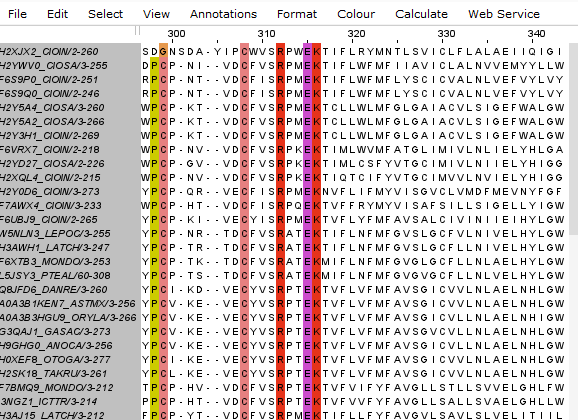
Рис.1 Найденный в Jalview мотив
Он имеет как строго консервативные позиции, так и позиции с высокой вариацией. Соответсвующий паттерн: C[FWY][IV][SA]RP[TSA][ED]K. Далее в этом же выравнивании был произведен поиск по составленому паттерну.
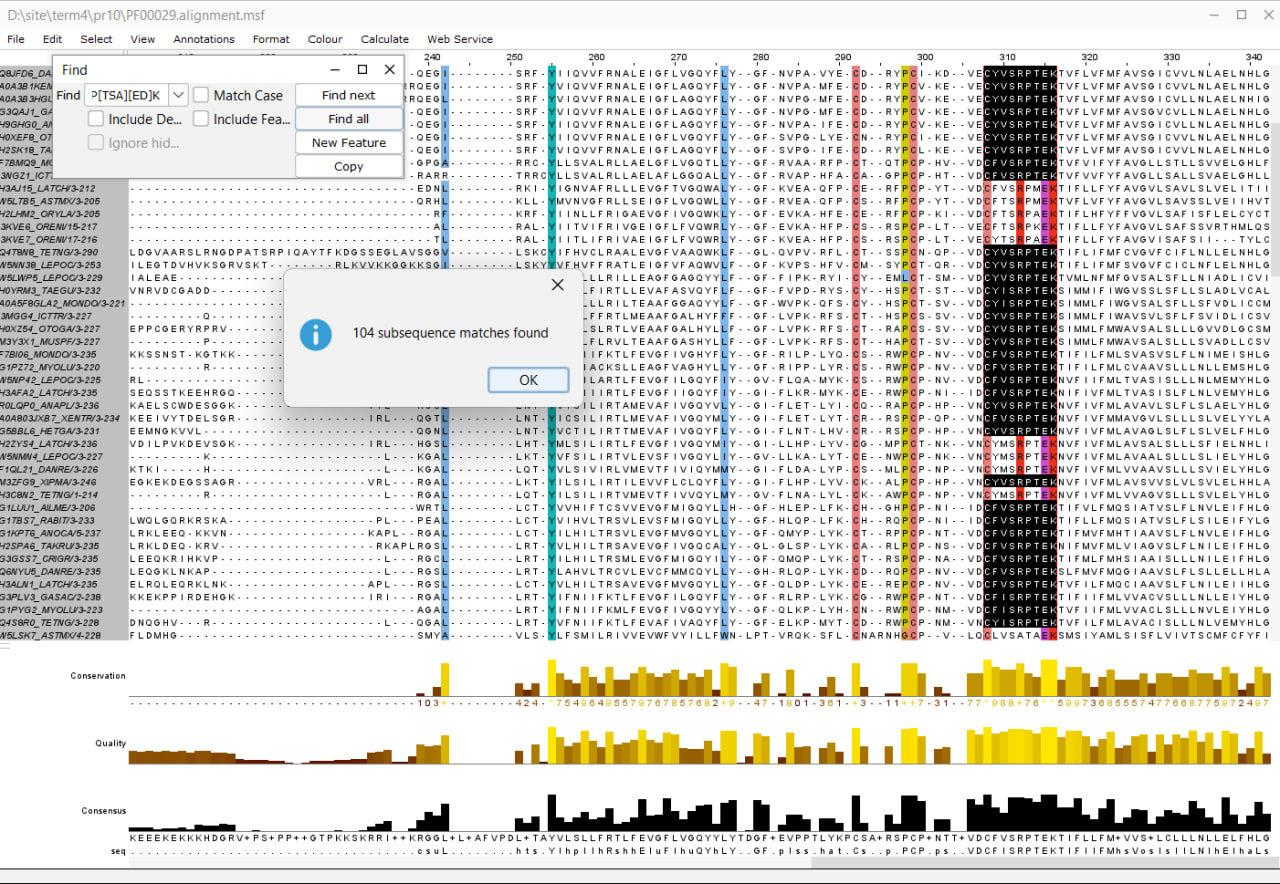
Рис.2 Поиск в Jalview по паттерну
В резултаты поиска не попало 7 последовтельностей, варианты указанные в них в неконсерватиынх позициях встречаются слишком редко.
Переведем паттерн в формат Prosite: C-[FWY]-[IV]-[SA]-RP-[TSA]-[ED]-K. Теперь с помощью этого паттерна проведем поиск в этой базе данных.
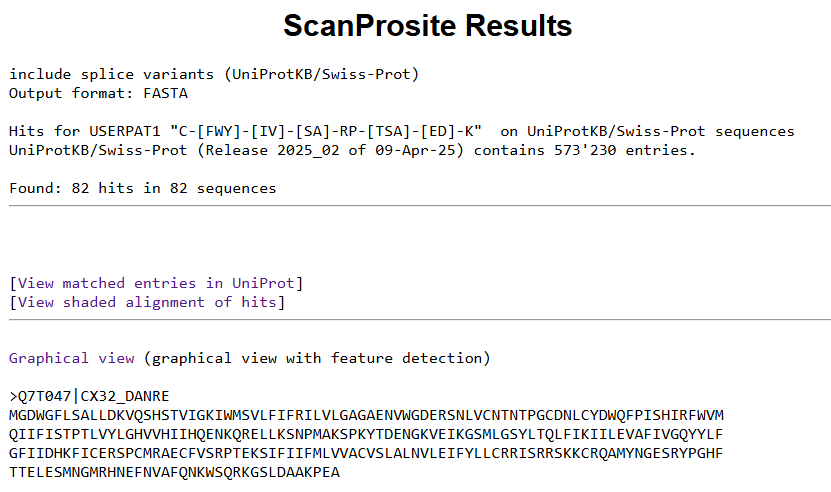
Рис.3 Результаты поиска в Prosite
Было найдено 82 вхождение паттерна в 82 последовательностях. Выходной формат поиска - fasta. Далее с помощью mapping по последовтельностям в UniProt были получены последовательности в которых нашелся заданный паттерн. Заметно, что большинсво находок аннотированы как Gap junction protein.
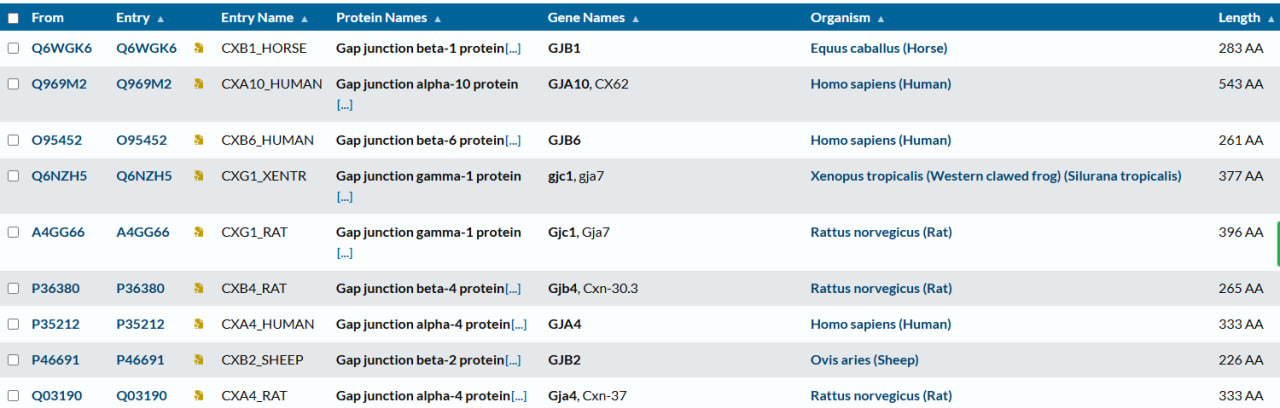
Рис.4 Записи находок в UniProt
Эти последовтельности были выровнены, так как оказались разной длины и паттерн не находился в одной колонке. После выравнивания паттерн оказался во всех последовтельностях в одних и тех же позициях выранивания.
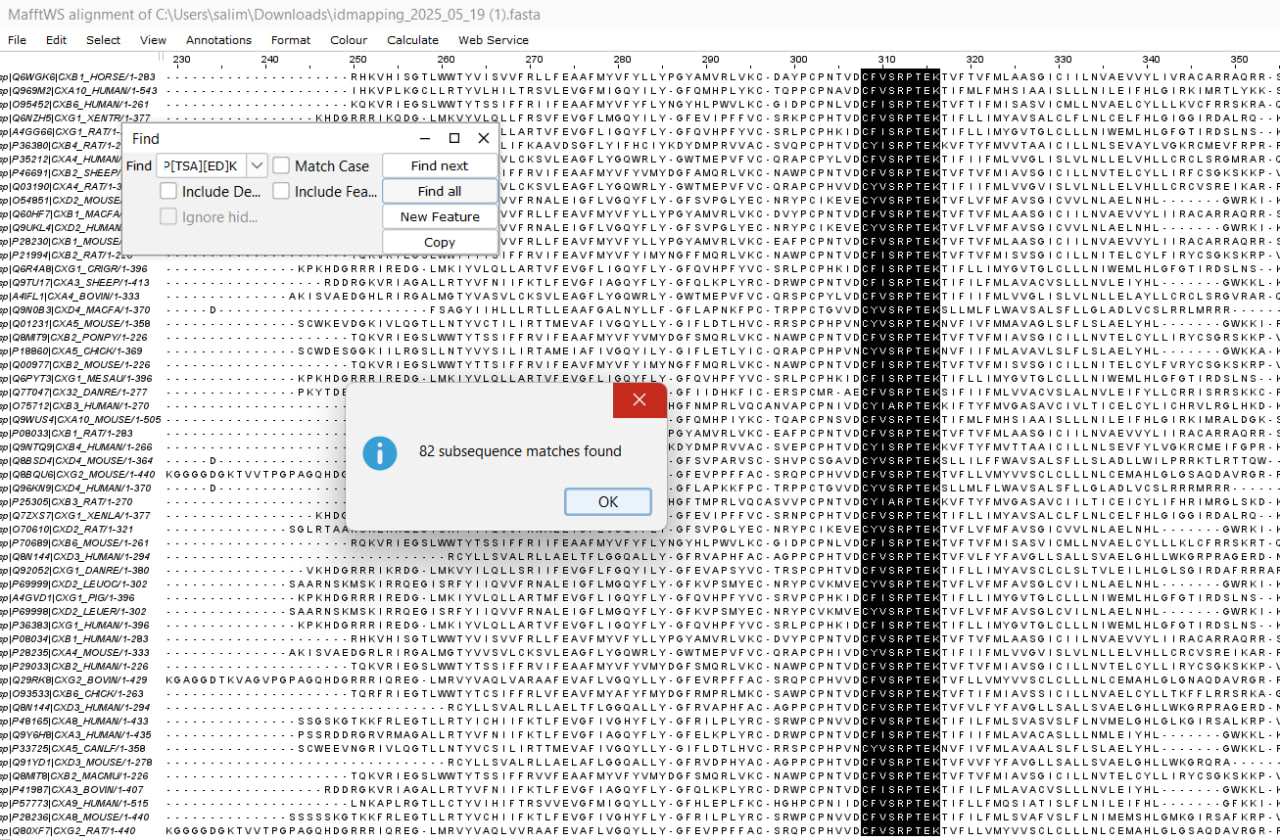
Рис.5 Паттерн в находках Prosite
2. Поиск мотива, специфичного для одной клады
С помошью NJ в Jalview было построено дерево, на нем отделена ветвь (показана на рисунке синим цветом).
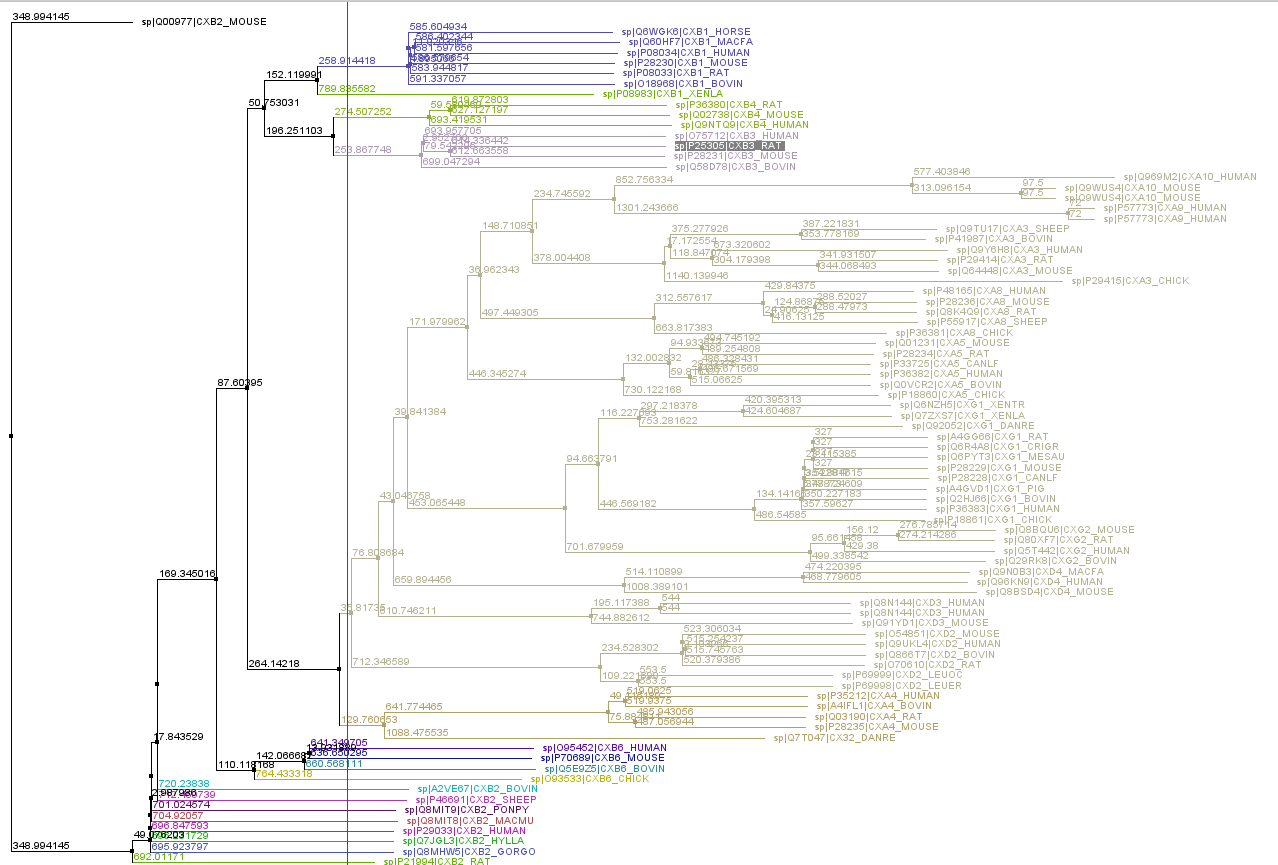
Рис.6 Дерево, построенное по выраниванию

Рис.7 Выбранная клада
Далее в выбранной кладе был найден паттерн:

Рис.8 Паттерн в кладе
Оказалось, что для последовательностей этой клады характерен консервативный паттерн SFVSRPTEK, не встречающийся в других последовательностях (поиск по всем последовтельностям выделяет только данные).
3. PSI-BLAST
Мною был выбран идентификатор Q67XL4. Ему соответсвует название в UniProt Неизвестный белок At3g25440, содержащий домен CRM, хлоропластный из Arabidopsis thaliana (Mouse-ear cress).
По нему был выполнен поиск PSI-BLAST по банку Swiss-Prot. Информация на каждой итерации представлена в таблице ниже.
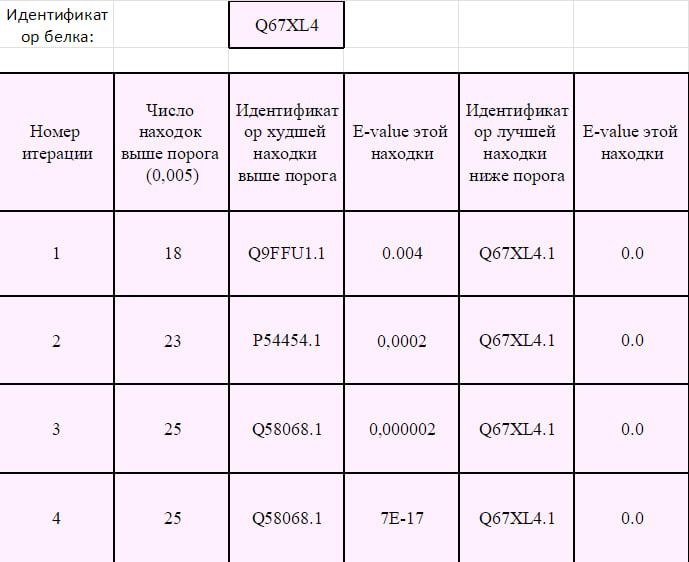
Рис.9 Данные об итерациях PSI-BLST
Число надпороговых по статистической значимости находок перестало уувелививаться после 3 итерации. Наилучай находка всегда оставалась одна, наихудшие находки всегда были вновь добавленными. При этом заметен четкий скачок на 2 порядка в E-value для худшей находки.
4. Поиск мотивов de novo
Для этого пункта использовались последовтельности с доменом из пункта 1 из swiss-prot. Изначально их количество 111. Далее с помощью remove redundancy с порогом 93 была выбрана 71 последовательность. На ней с помощью meme были найдены паттерны. Результаты поиска meme. Среди находок точного паттерна из пунта 1 найдено не было, есть частичное сопадение по позициям для одной находки meme.
Рис.10 Лучшая находка meme
Далее с помощью fimo был произведен поиск наденный паттернах в исходных 111 последовательностях. Результаты поиска fimo. Примечательно, что чаще находки fimo соответсвуют паттерну 2 в meme. Но находок первого паттерна тоже достаточно.
5. Представленность сайта GATC в геноме
Я когда-то давно выбрала не бактерию, а архею Natrinema halophilum. С помощью cbcalc был произведен поиск по всем перестановкам без посторений букв GATC. Результаты показаны на гистограмме.
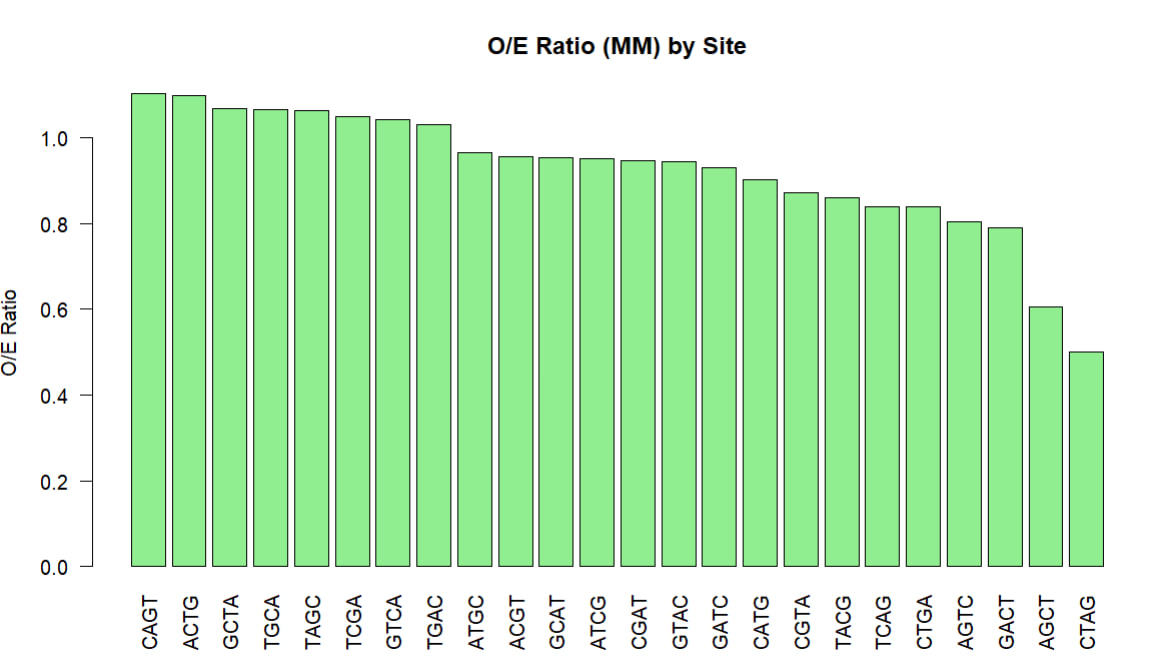
Рис.11 Представленность сайтов перестановок GATC в геноме Natrinema halophilum
Для самого сайта GATC искомое отношение равно 0.931, что близко к 1, однако все равно указывает на недопредставленность относительно ожидаемого.
Хотя для архей и не свойственно метилирование по GATC сайту, а механизм метилирования ближе к эукариотам, для отдельных групп описано сайт-специфичное метилирование по GATC. Такое показано как раз для некоторых галофильных арехей, к котрым относится и выбранная мной. Однако, кроме того GATC сайт играет роль у таких же архей в системах рестрикции-модицикации, в связи с чем может подавляться в геноме.