Практикум 2
1. Выравнивание последовательностей цитохрома b
Для начала был создан файл со списком USA исследуемых последовательностей. Далее с помощью seqret были получены сами последовательности. С помощью Muscle получено множественное выравнивание, с привлечением скрипта python оно было преобразовано в формат phylip-relaxed. Далее все программы работают с файлом в этом формате.
2. Топология дерева, построенного на основе систематики
На исходном дереве от корня отходили две большие группы животных Archelosauria и Lepidosauria. В описываемой группе первая включает пртиц и черепах, а вторая змей и прочих рептилий. Подробнее на рисунке 1.
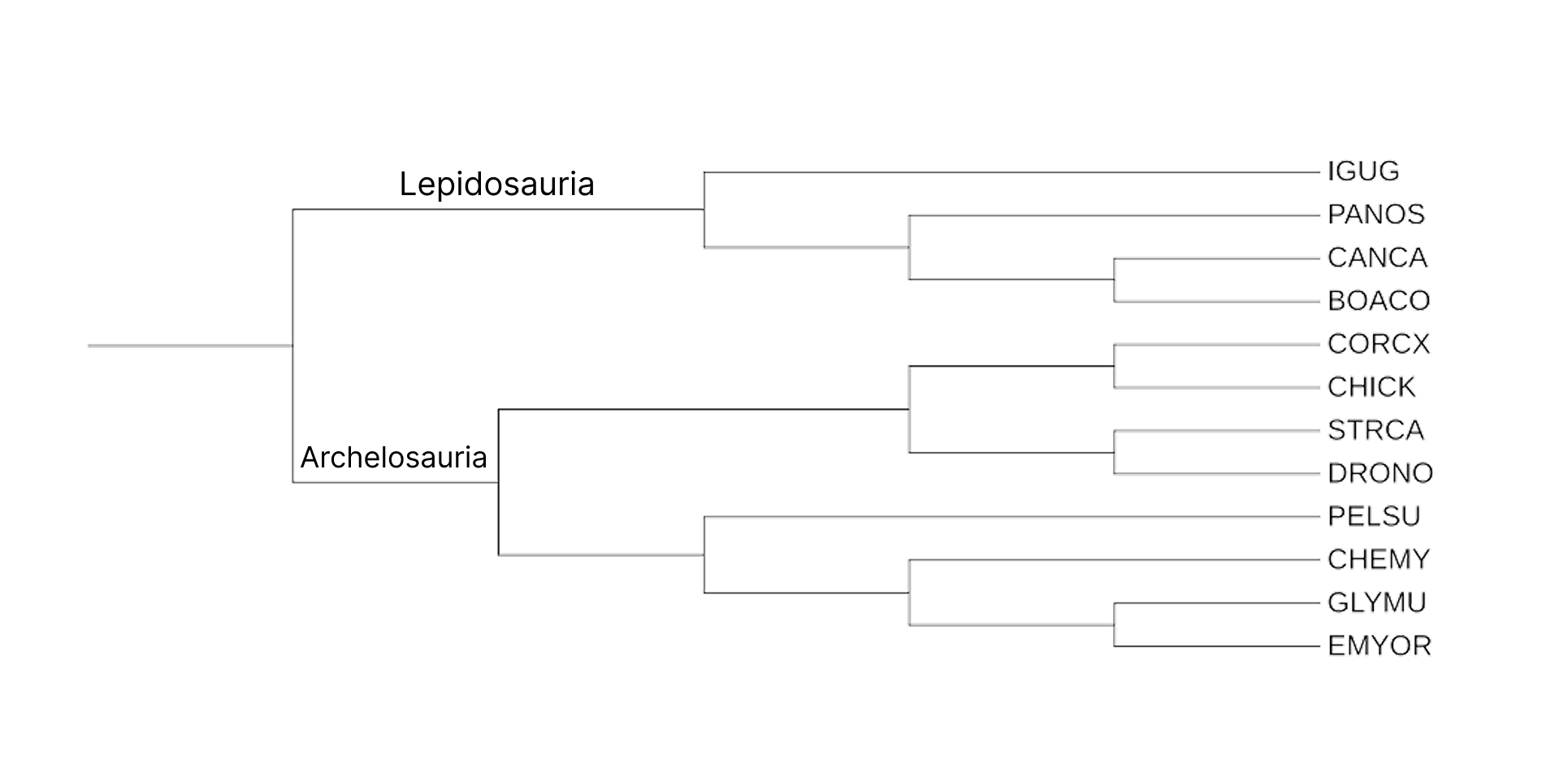
Рис. 1 Реконструкция дерева на основе систематики
3.1. Реконструкция дерева программой FastME с p-distance
Для реконструкции дерева с оценкой расстояния с помощью p-distance была использована команда:
| fastme -i cyb.phy -o p_dist.tre -pp-distance |
Здесь под параметрами -i и -o указаны входной и выходной файлы, соответсвенно, -p задает модель.
В результате получено дерево в формате Newick:
((((CYB_IGUIG:0.10092508, ((CYB_CHEMY:0.05461187, (CYB_EMYOR:0.02506461, CYB_GLYMU:0.02454374):0.03283081) :0.04506758, CYB_PELSU:0.13817013):0.01410436) :0.01095461, ((CYB_CANCA:0.07519582, CYB_BOACO:0.06579634) :0.03688618,CYB_PANOS:0.10615995) :0.17619801): 0.03409873,CYB_CORCX:0.18883552) :0.04156264,(CYB_DRONO:0.01899544, CYB_STRCA:0.02539098):0.01808004, CYB_CHICK:0.04446834);
С использованием ITOL дерево было визуализировано и укоренено в месте разделения Archelosauria и Lepidosauria.
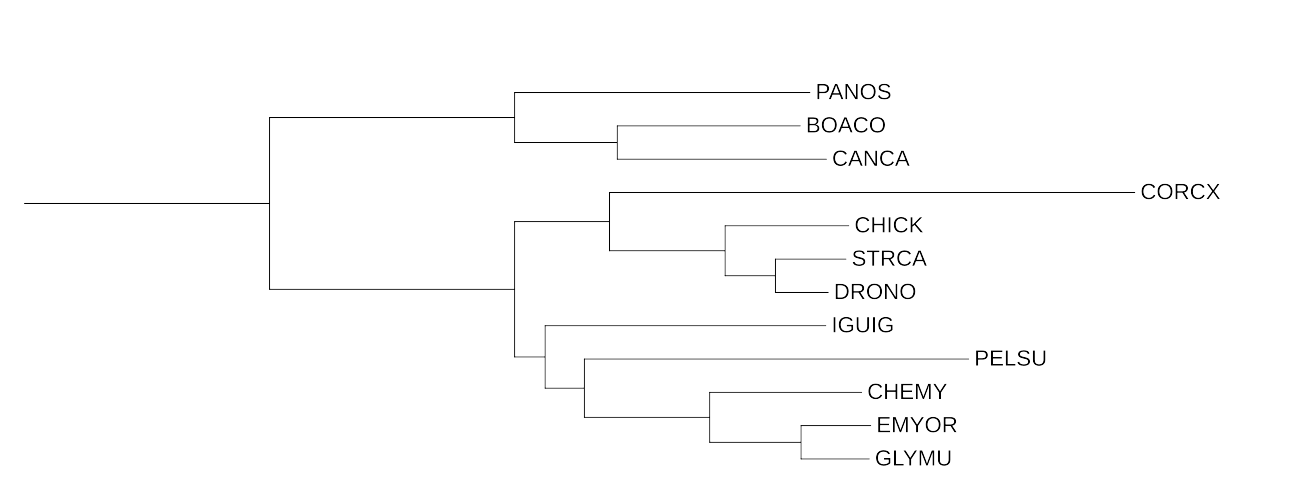
Рис. 2 Реконструкция дерева FastME c p-distance
По результатам реконструкции заметны отличия от дерева, построенного с помощью систематики. Конечно, сохранилась основаная топология разделения на две группы. Однако, заметно что Iguana iguana(IGUIG) на построенном дереве оказалась не в своей группе. Она оказалась в одной группе с архозаврами, что нарушает систематику. Еще одно отличие состоит в том, что в группе птиц систематически происходит разделение на Palaeognathae и Neognathae. Однако на восстановленном на основании последовтельностей дереве Gallus gallus(CHICK) (относится к Neognathae) в одной группе с представителями Palaeognathae.
3.2. Реконструкция дерева программой FastME с MtREV
Для реконструкции дерева с оценкой расстояния с помощью MtREV была использована команда:
| fastme -i cyb.phy -o Mt.tre -pMtREV |
В результате получено дерево в формате Newick:
((((((CYB_CHEMY:0.06032053, (CYB_EMYOR:0.02350962, CYB_GLYMU:0.02748622):0.03653035) :0.06198772, CYB_PELSU:0.18372733):0.01871822, CYB_IGUIG:0.13916379):0.01848928,((CYB_CANCA:0.08395294, CYB_BOACO:0.07836486):0.03937517, CYB_PANOS:0.11317093):0.34185343):0.07373421, CYB_CORCX:0.04549023):0.03638305,(CYB_DRONO:0.02114271, CYB_STRCA:0.02881712):0.02491685, CYB_CHICK:0.04515908);
Аналогично дерево было визуализировано и укоренено.
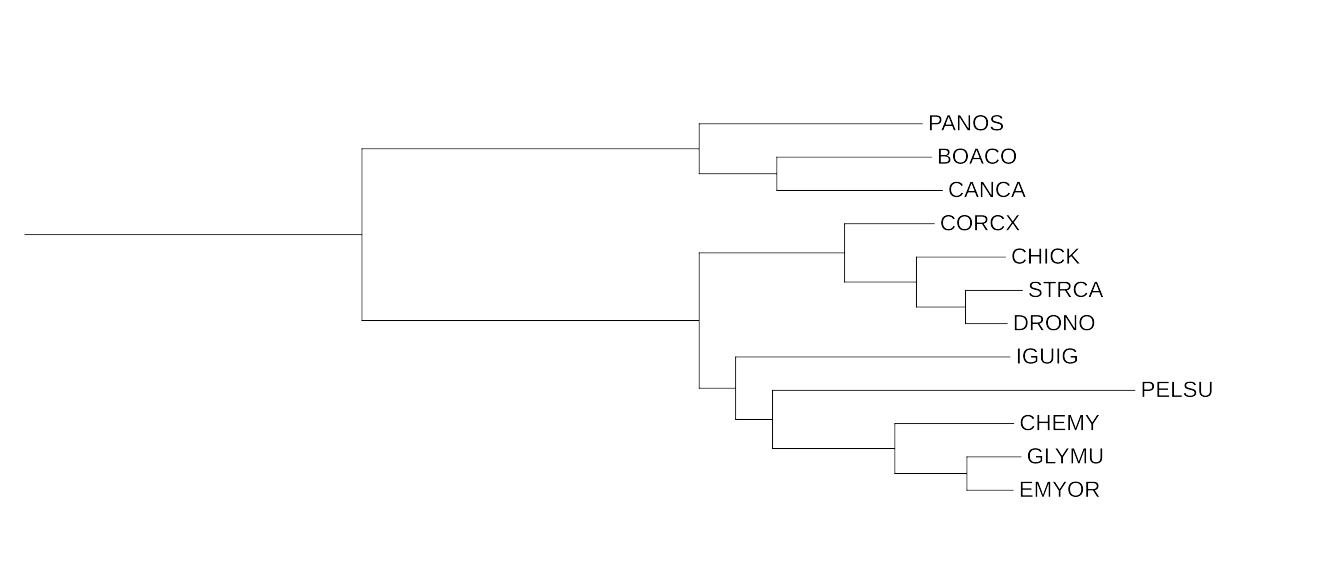
Рис. 3 Реконструкция дерева FastME c MtREV
Топология дерева полностью совпадает с предыдущей, однако заметны различия в длинах листьев. Так, например, в предыдущем дереве длина листа Corvus corax(CORCX) заметно длиннее.
4. Реконструкция дерева программой iqtree
Для реконструкции дерева с оценкой расстояния с помощью iqtree была использована команда:
| iqtree -s cyb.phy |
В результате получено дерево в формате Newick:
(CYB_STRCA:0.0427867993, (((((CYB_BOACO:0.1139412301, CYB_CANCA:0.0758279071):0.0812058967, CYB_PANOS:0.1345764158):0.8825619509,(CYB_IGUIG:0.1789810077,(((CYB_GLYMU:0.0233585106, CYB_EMYOR:0.0289457860):0.0607225321, CYB_CHEMY:0.0500565658):0.0711707305, CYB_PELSU:0.3199016101):0.0875416332):0.0787464458):0.1071317678, CYB_CORCX:0.0849513023):0.0415341908, CYB_CHICK:0.0487376224):0.0328772735, CYB_DRONO:0.0118214907);
Аналогично дерево было визуализировано и укоренено.
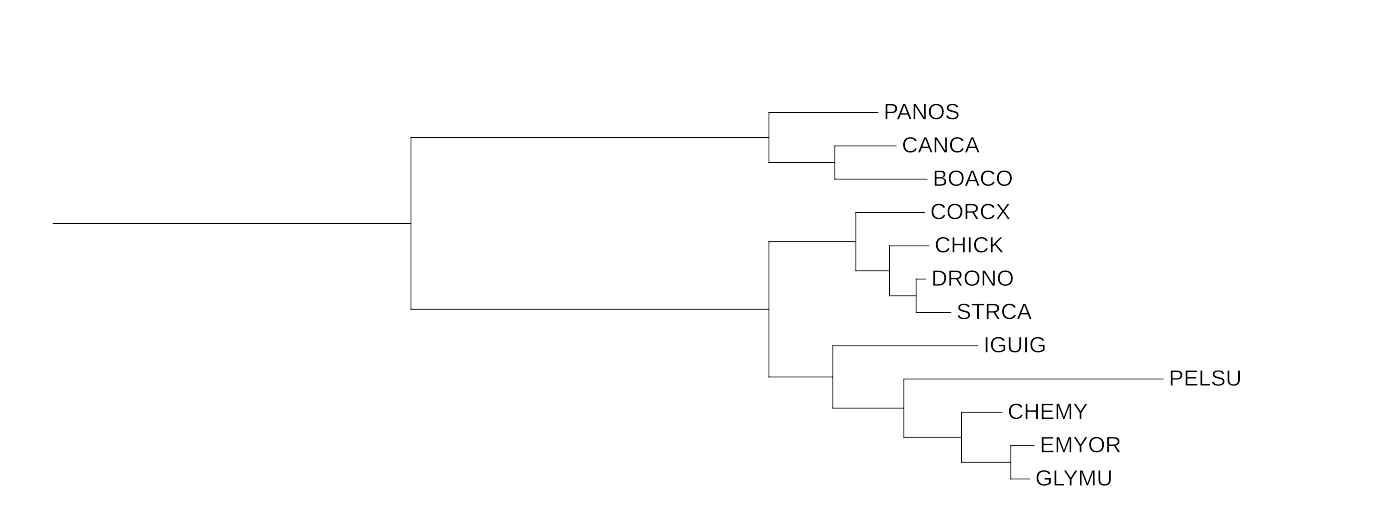
Рис. 4 Реконструкция дерева iqtree
Здесь так же совпадает топология, отличия заметны только в относительной длине ветвей.