Практикум 4
1. Поиск гомологичных последовтельнсотей
Среди представленных организмов я выбрала 8 со следующими мнемониками: HAEIN, PARDP, POLAQ, PSEAE, RHIME, SACD2, SHEDO, YERPE. На основании их протеомов была создана база данных. В этой базе данных сделан запрос по белку CLPX_ECOLI. Результат работы blast с установленным приемлемым e-value можно посмотреть здесь.
2. Построение деревьев
По последовтельностям, найденным blast, было построено множественное выравнивание, а на его основении с помощью iqtree реконструировано дерево. Посмотреть Newick форму можно здесь. На рисунке 1 показано реконструировано дерево с уоренением в средную точку. Заметно, что из-за длиной ветви разделяющей HSLU и CLPX против находок с непонятными мненимониками четкие группы паралагов не реконструировались. Как мне кажется, строго говря, нельяз сказать что группа с мнемоникой на HSLU является отдельной ортологичной. Поэтому дерево имеет соответсвующие цвета. HSLU - субъединица HslU АТФ-зависимой протеазы (ATP-dependent protease ATPase subunit HslU). Она работает в комплексе с субъединицей HslV, обеспечивая энергию, необходимую для разворачивания и транслокации белковых субстратов в протеолитическую камеру. АТФ-зависимая активность HslU позволяет протеазе эффективно разрушать неправильно свернутые или поврежденные белки. CLPX - субъединица, связывающая АТФ, в Clp протеазе (ATP-dependent Clp protease ATP-binding subunit ClpX). Она имеет точно такую же функцию, что и предыдущий белок A1B8N4 - ClpA – АТФ-связывающая субъединица в Clp-протеазе (ATP-dependent Clp protease ATP-binding subunit ClpA) A0A5P8YB42 - белок семейства магний-хелатазы A1AZV8, Q92M98, A0A5P8YCE6 - FtsH – АТФ-зависимая металлопротеаза, регулирующая turnover мембранных и цитоплазматических белков, а также участвующая в ответе на стресс
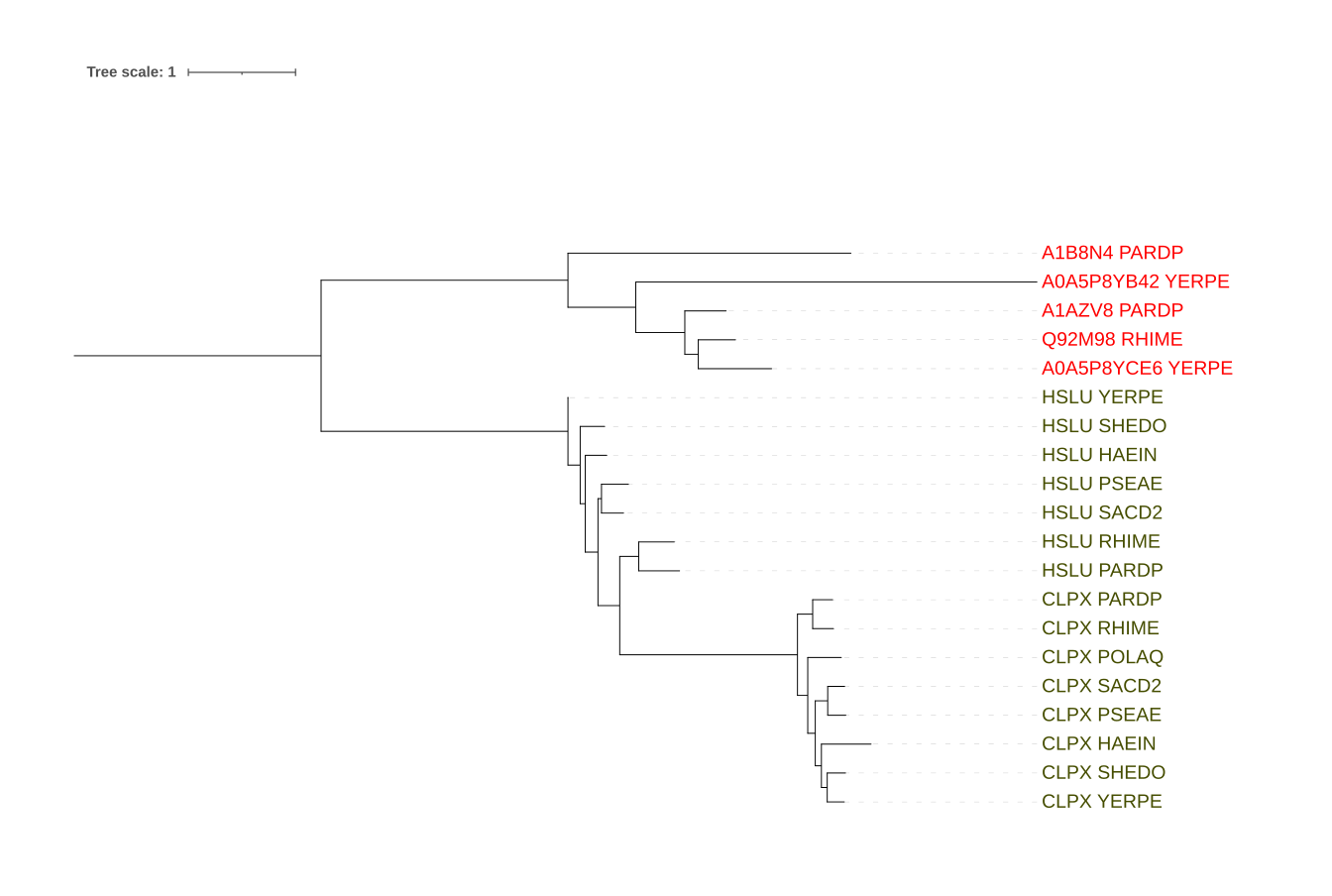
Рис.1 Дерево, реконструированное iqtree
На этом дереве, строго понимая ортологов и паралагов и при этом считать в целом гомологичными белками белки, которые имеют общее эволюционное происхождение, но при этом разные функции: Ортологи: CLPX_PSEAE и CLPX_SACD2, CLPX_PARDP и CLPX_RHIME, HSLU_YERPE и HSLU_SHEDO Паралоги: A1B8N4_PARDP и HSLU_PARDP, A0A5P8YB42_YERPE и CLPX_YERPE, Q92M98_RHIME и CLPX_RHIME.
При этом, если построить дерево, не включая "дальние" находки, то группы при укоренении в среднюю точку разделяются однозначно, как показано на рисунке 2
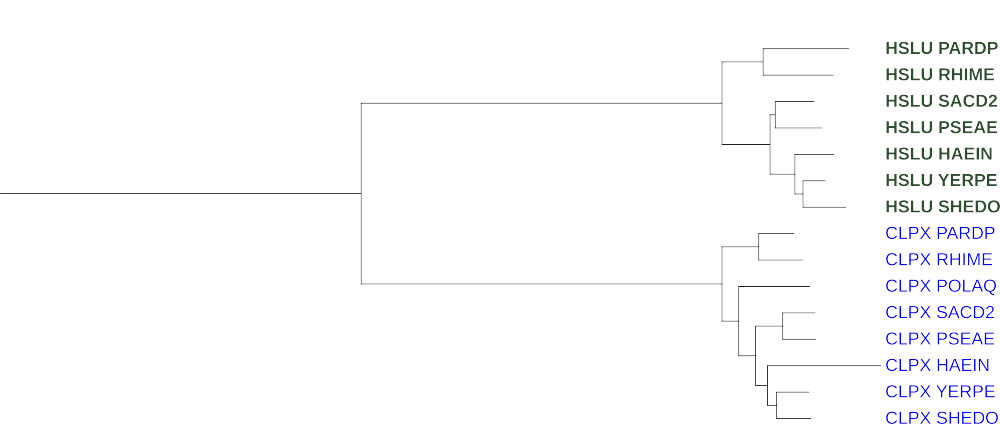
Рис.2 Дерево, реконструированное iqtree без дальних находок
На рисунке 3 показfно дерево, отображающее топологию разделения на две группы. Построено оно путем схлопывания двух клад в дереве номер 2.
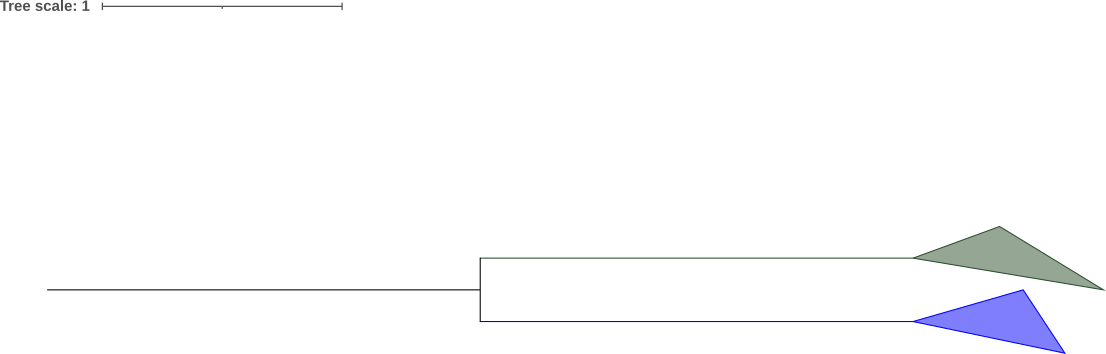
Рис.3 Дерево c объединенными ветвями
Кроме того, полное дерево реконсруировано не соотвественно филогении: даже в отдельной ветке с мнемониками СLPX, например, SHEDO оказался в одной кладе с YERPE, хотя согласно филогении SHEDO лежит во внешней группе отосительно HAEIN и YERPE.
На рисунке 2 можно выделить пары паралогов: CLPX_YERPE и HSLU_YERPE, CLPX_SHEDO и HSLU_SHEDO, CLPX_SACD2 и HSLU_SACD2. А так же пары ортологов: CLPX_PSEAE и CLPX_SACD2, CLPX_PARDP и CLPX_RHIME, HSLU_YERPE и HSLU_SHEDO.