Практикум 7
1. Выбор и описание белка с бета-складками
С помощью поиска по уровням классификации в базе данных OPM в разделе Mycobacterial Porin (MBP) был найден белок Porin MspA из организма Mycolicibacterium smegmatis. Интересным я его посчитала, потому что есть данные, хоть и об ограниченном, но все же использовании в нанопоровом секвенировании. Данные о выбранном белке представлены в таблице 1.
Таблица 1 Данные о выбранном белке
| Название белка (русский) | Порин MspA |
| Название белка (английский) | Mycobacterium smegmatis porin A (Porin MspA) |
| Идентификатор PDB | 1uun |
| Идентификатор UniProt | MSPA_MYCS2 |
| Организм | Mycobacterium smegmatis |
| Мембрана | Внешняя мембрана |
| Функция белка | MspA образует поры в клеточной стенке, позволяя транспортировать молекулы через мембрану. Способствует проникновению катионов, аминокислот, железа Fe³⁺ и, в меньшей степени, фосфатов. Играет роль в переносе бета-лактамазы и гидрофильных фторхинолоновых антибиотиков, таких как норфлоксацин, а также хлорамфеникола. |
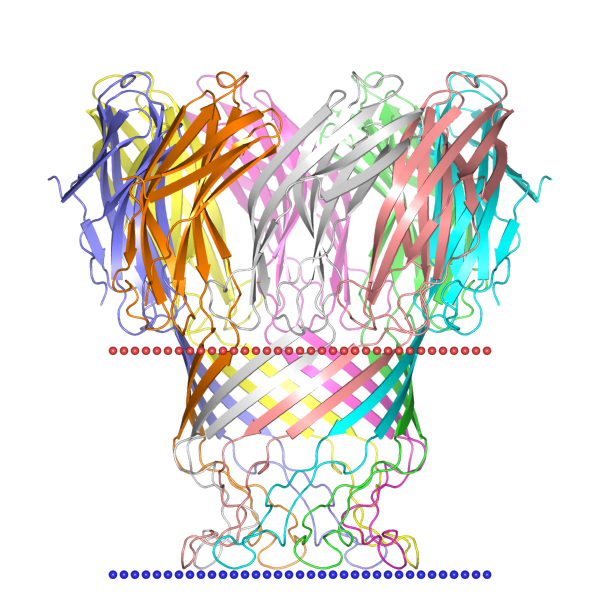
Рис.1Mycobacterium smegmatis porin A (Porin MspA) из ОРМ
Сам белок состоит из восьми субъединиц, то есть является октамером. Координаты трансмембранных участков субъединиц представлены в таблице ниже:
| Название | Трансмемебранные участки |
| A | 1(74-83), 2(111-119) |
| B | 1(74-83), 2(111-119) |
| C | 1(74-83), 2(111-119) |
| D | 1(74-83), 2(111-119) |
| E | 1(74-83), 2(111-119) |
| F | 1(74-83), 2(111-119) |
| G | 1(74-83), 2(111-119) |
| H | 1(74-83), 2(111-119) |
2. Предсказание DeepTMHMM
Последовательность была вязята из соответсвующей записи Uniprot. С помощью веб интерфейса было проведено предсказание структруной прналежности частей белка. С табличной выдачей можно ознакомиться. В графическом виде результаты показаны на рисунке 1.
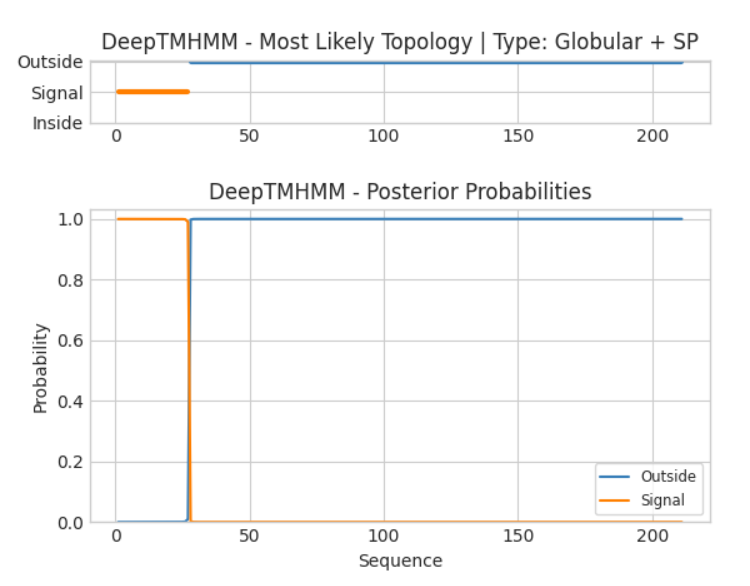
Рис.2 Графическая выдача программы DeepTMHMM для выбранного порина
Видно, что трансмембранные участки вовсе не предсказаны. Такое можно объяснить, во-первых, преобладающей внешней частью, то есть трансмембранная часть сама по себе небольшая. Во-вторых, бета-бочка формируется целиком с участием всех субъединиц.
Так как очевидно, что белок хоть и интересный его структура не позволяет освоить все анализы данных персдказания трансмембранных участков, так как они не пресказываются вовсе. Пришлось взять другой белок, пусть и менее интересный.
Вообще неитересный, но потенциально хорошо предсказываемый белок - CymD hydrocarbon channel из организма Pseudomonas putida.
Таблица 2 Данные о CymD hydrocarbon channel
| Название белка (русский) | CymD углеводородный канал |
| Название белка (английский) | CymD hydrocarbon channel |
| Идентификатор PDB | 6z34 |
| Идентификатор UniProt | O33458_PSEPU |
| Организм | Pseudomonas putida |
| Мембрана | Внешняя мембрана |
| Функция белка | CymD является трансмембранным белком, который образует канал, позволяющий углеводородам проникать через клеточную мембрану |
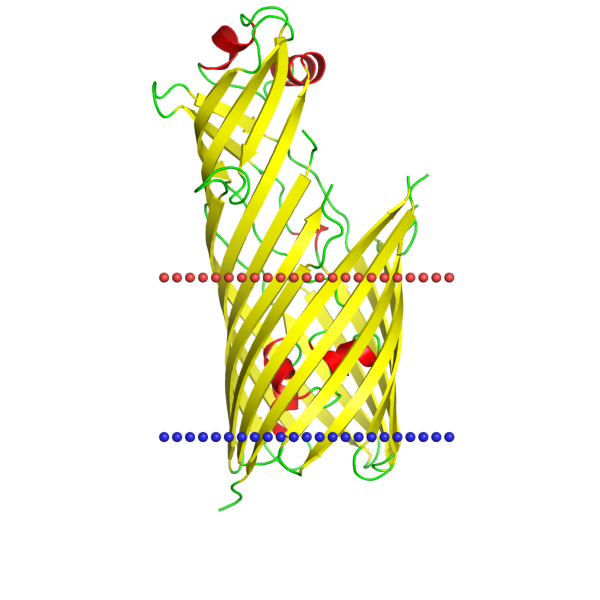
Рис.3 CymD hydrocarbon channel из ОРМ
Трансмембранные участки: 44-53, 81-89, 94-103, 136-144, 152-159, 217-227, 235-241, 279-288, 295-301, 336-344, 352-358, 378-386, 394-401, 425-434.
2.1 Предсказание DeepTMHMM
Последовательность была вязята из соответсвующей записи Uniprot. С помощью веб интерфейса было проведено предсказание структруной прналежности частей белка. С табличной выдачей можно ознакомиться. В графическом виде результаты показаны на рисунке 4.
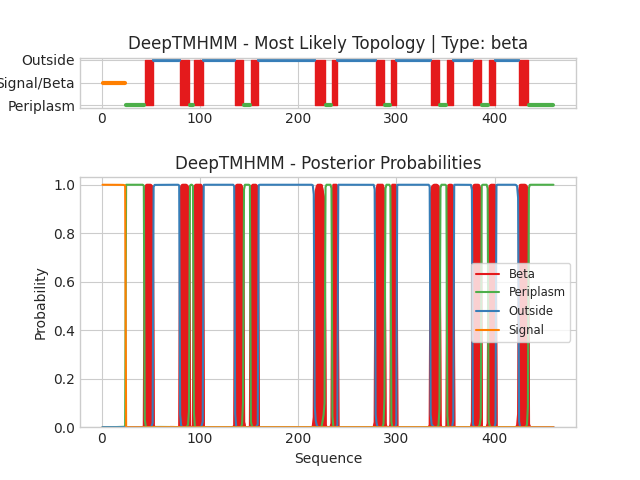
Рис.4 Графическая выдача программы DeepTMHMM для канала CymD hydrocarbon channel
Далее предсказанные трансмемтранные участки сравнены с указанными в базе данных. Таблица сравнения:
| ОРМ | DeepTMHMM |
| 44-53 | 44-52 |
| 81-89 | 80-89 |
| 94-103 | 94-103 |
| 136-144 | 136-144 |
| 152-159 | 152-159 |
| 217-227 | 217-227 |
| 235-241 | 235-240 |
| 279-288 | 279-288 |
| 295-301 | 295-300 |
| 336-344 | 335-344 |
| 352-358 | 352-358 |
| 378-386 | 378-386 |
| 394-401 | 394-401 |
| 425-434 | 425-434 |
Видно, что координаты почти идельано совпадают с данными ОРМ, максимально различие всего 1 позиция.
3. Альфа-спиральный белок
Информация о выданном белке в таблице ниже.
Таблица 5 Данные о выданном белке с альфа спиралями
| Название белка (русский) | Кальциевая АТФаза, состояние E2 (без Ca), комплекс с фосфоламбаном |
| Название белка (английский) | Calcium ATPase, E2 state (Ca-free), complex with phospholamban |
| Идентификатор PDB | 4kyt |
| Идентификатор UniProt | AT2A1_RABIT, PPLA_CANLF |
| Организм | Oryctolagus cuniculus |
| Мембрана | Мембрана эндоплазматического ретикулума |
| Функция белка | Ключевой регулятор работы поперечно-полосатых мышц, выступающий в качестве основной Ca2+-АТФазы, отвечающей за возврат цитозольного Ca2+ в саркоплазматический ретикулум. Катализирует гидролиз АТФ в сочетании с перемещением кальция из цитозоля в просвет саркоплазматического ретикулума. |
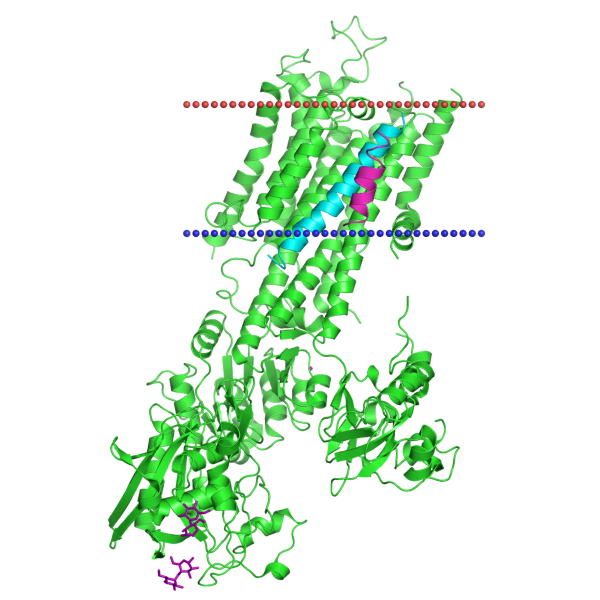
Рис.5 Calcium ATPase из ОРМ
Сам белок состоит из 3 субъединиц, то есть является тримером. Координаты трансмембранных участков субъединиц представлены в таблице ниже:
| Название | Трансмемебранные участки |
| A | 60-78, 86-105, 260-279, 291-306, 760-781, 789-807, 832-853, 896-916, 933-950, 967-987 |
| B | 28-48 |
| C | 27-38 |
3.1 Предсказание DeepTMHMM
Последовательность субъединицы, указанной в таблице как A, была вязята из соответсвующей записи Uniprot. С помощью веб интерфейса было проведено предсказание структруной прналежности частей белка. С табличной выдачей можно ознакомиться. В графическом виде результаты показаны на рисунке 4.
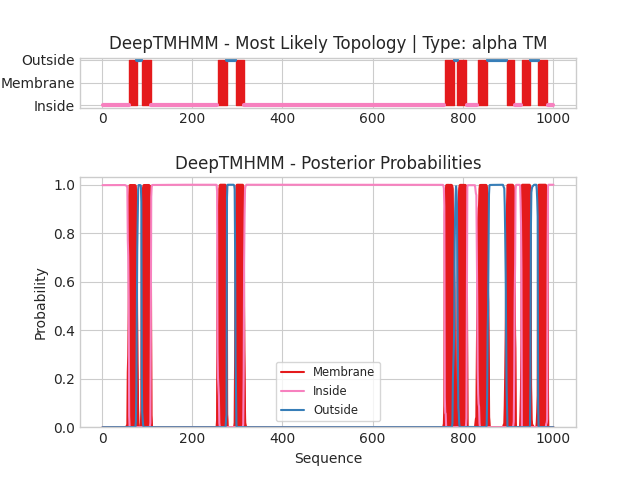
Рис.6 Графическая выдача программы DeepTMHMM для A цепи Calcium ATPase
Далее предсказанные трансмемтранные участки сравнены с указанными в базе данных. Таблица сравнения:
| ОРМ | DeepTMHMM |
| 60-78 | 60-77 |
| 86-105 | 88-108 |
| 260-279 | 256-276 |
| 291-306 | 296-314 |
| 760-781 | 760-781 |
| 789-807 | 788-808 |
| 832-853 | 833-854 |
| 896-916 | 897-914 |
| 933-950 | 931-950 |
| 967-987 | 967-987 |
Здесь так же координаты практически свопадают с максимальм отличем в 5 позиций.
Таким образом, DeepTMHMM - хорший инструмент для предказания структуры белков на основании их последовательностей, причем работает он и для белков с альфа-спиралями и бета-скалдками. Однако, есть ограничения на мультубъединичные белки и превалирующие нетранмембранные участки.