|
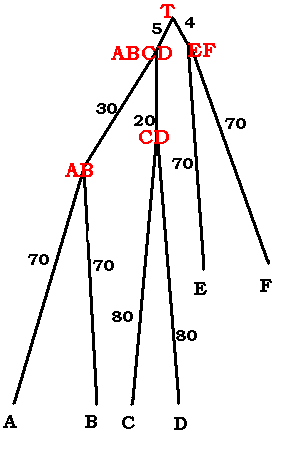
(((A:70,B:70):30, (C:80,D:80):20):5, (E:70,F:70):4);
|

|
Длина последовательности - 3592 нт
Мутант ABCD - (3592/100)*5=180
EF - 35.92*4=144
CD - 35.92*20=718
AB - 35.92*30=1078
A - 35.92*70=2514
B - 35.92*70=2514
C - 35.92*80=2874
D - 35.92*80=2874
E - 35.92*70=2514
F - 35.92*70=2514
последовательности мутантных генов были получены с помощью следующего скрипта |
|
Последовательности, соответствующие листьям дерева, я поместила в файл
ali.fasta .
Реконструкция дерева алгоритмом максимального правдоподобия (программа fdnaml, переборный алгоритм, строит неукорененное дерево): fdnaml ali.fasta -ttratio 1 -auto (результат - в файле ali.fdnaml) +---------------b | | +f | +--4 | | +---------------e 1------3 | | +-------------------d | +----2 | +-------------------c | +----------------a Реконструкция дерева алгоритмами UPGMA или Neighbor-joining. С помощью программы fdnadist считаем попарные расстояния между последовательностями (результат - в файле ali.fdnadist): fdnadist ali.fasta -ttratio 1 -auto Алгоритм Neighbor-joining (эвристический, неукорененное дерево). Подаем файл на вход программе fneighbor: fneighbor ali.fdnadist -auto -outfile alinj (результат - в файле alinj.fasta) +----------------b ! ! +-------------------c ! +------2 ! ! +-------------------d 1-------3 ! ! +----------------e ! +-4 ! +f ! +----------------a Алгоритм UPGMA (эвристический, ультраметрическое дерево): fneighbor ali.fdnadist -treetype u -auto -outfile aliup (результат - в файле aliup.fasta)
+--------------------------------a
+-2
! ! +---------------e
+--------3 +----------------1
! ! +---------------f
! !
--5 +---------------------------------b
!
! +--------------------------------------c
+---4
+--------------------------------------d
Согласно таблице, равными исходному (имеющими одинаковую топологию) можно считать деревья, построенные с помощью алгоритмов ML и Neighbor-joining. UPGMA алгоритм построил дерево с ветвью AEF вместо ветви AB. Вероятно, ошибка возникла в связи с принципом "молекулярных часов", на котором основан данный алгоритм: скорость накопления мутаций - то, что составляет длину ветвей - одинакова для каждого листа, если считать от корня. Так же, это единственный из представленных алгоритмов, строящий укорененные деревья. Сравнения деревьев и таблица разбиения представлены в отчете Protocol2. |
| 4 семестр |
| на главную |